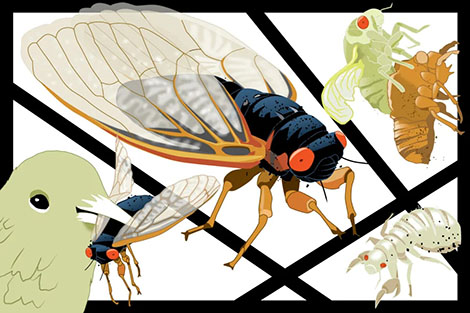
 Die Fressorgie des Jahrhunderts naht – zumindest wenn man Zikaden mag. In diesem Frühjahr werden sich im Osten der USA innerhalb weniger Wochen schätzungsweise eine Billion Larven aus der Gattung der Magicicada, der Periodischen Zikaden, nach oben graben und aus ihren Erdlöchern schlüpfen. Mit bis zu 400 Individuen pro Quadratmeter. Bilder einer biblischen Wanderheuschreckenplage drängen sich förmlich auf.
Die Fressorgie des Jahrhunderts naht – zumindest wenn man Zikaden mag. In diesem Frühjahr werden sich im Osten der USA innerhalb weniger Wochen schätzungsweise eine Billion Larven aus der Gattung der Magicicada, der Periodischen Zikaden, nach oben graben und aus ihren Erdlöchern schlüpfen. Mit bis zu 400 Individuen pro Quadratmeter. Bilder einer biblischen Wanderheuschreckenplage drängen sich förmlich auf.
Zum Glück sind die Pflanzensaftsauger für Mensch und Tier harmlos. Weder stechen oder beißen sie noch übertragen sie Krankheiten. Das Außergewöhnliche an ihnen: Magicicada treten stets im Rudel auf – und diesmal in einem außerordentlich großen. Reihte man alle bis Ende April erwarteten Zikaden aneinander, so reichte die Insekten-Polonaise mehr als dreißigmal bis zum Mond und wieder zurück.
Ohne jahrelange minutiöse Vorbereitung wäre ein solches Massenauftreten natürlich unmöglich. Über ein Jahrzehnt durchlaufen Magacicada-Larven unterirdisch fünf Larvenstadien. Das letzte von ihnen arbeitet sich dann im Frühjahr des 13. oder 17. Jahres in Richtung Erdoberfläche … und wartet. Worauf? Auf die richtige Temperatur. Erreicht der Boden etwa 18 Grad Celsius, krabbeln sie ins Freie und häuten sich ein letztes Mal zur komplett schwarzen Imago mit ihren charakteristisch roten Komplexaugen. Das Besondere in diesem Jahr: Die 13- und 17-Jahresbruten überlappen. Zuletzt war das 1803 der Fall, das nächste Mal wird es voraussichtlich 2245 sein. Die Fressorgie kann also beginnen.
Allerdings weniger für die Zikaden selbst. Mehr für Spinnen, Raubwanzen, Ameisen und Vögel, die schon mal ihre Mandibeln, Cheliceren oder Schnäbel wetzen können. Für sie wird das Schlaraffenland wahr.
Übrigens auch für den Menschen – falls er denn will. Denn auf der Weltliste essbarer Insekten stehen auch alle Periodischen Zikaden. Gewiefte Insektenköche an der US-Ostküste werden sie ohne Zweifel auf Pizza, in Paella oder als Sushi verarbeiten. Mit einem physiologischen Brennwert von etwa 150 Kilokalorien pro Imago sind sie schließlich ebenso nahrhaft wie gebratenes Hühnchen – dafür aber garantiert aus Freilandhaltung und bei Selbstzubereitung zum Nulltarif.
Aber warum wird diese Delikatesse nur alle 13 beziehungsweise 17 Jahre serviert? Aus Überlebenskalkül. Indem die Magicicada nur alle 13 oder 17 Jahre in Scharen auftreten, werden ihre Fressfeinde übersättigt. Diese leben in der Regel in 1-, 2-, 4- oder 6-Jahresrhythmen und können ihre Reproduktionszyklen daher nur schwer mit den Primzahlen der Periodischen Zikaden synchronisieren. Es bleiben immer ausreichend Zikaden übrig, die nicht gefressen werden. Ihr Arterhalt ist gesichert.
So dramatisch wie ihre Zeit über der Erde ist, so kurz ist sie auch. Lediglich sechs Wochen bleiben Magicicada, um sich zu paaren – und zu sterben: die Männchen direkt nach dem Akt; die Weibchen, nachdem sie noch schnell Hunderte von Eiern in die Rinde lebender Zweige gelegt haben. Zwei Monate später schlüpft aus ihnen dann das erste Larvenstadium, fällt zu Boden und gräbt sich ein, um seine 13- oder 17-jährige Larvalentwicklung zu beginnen.
Die zwei Primzahlen haben die Tierchen also tief verinnerlicht. Welches Insekt „kann“ noch mehr Mathematik? Wir nehmen gerne Beispiele.
Henrik Müller
(Der Text erschien in leicht anderer Form als Editorial unseres aktuellen Laborjournal-NEWSLETTERS. Wer den NEWSLETTER samt solcher Editorials regelmäßig alle zwei Wochen per E-Mail zugeschickt bekommen möchte, klicke sich bitte hier entlang!)

 Natürlich könnte sich Dionaea muscipula, die Venusfliegenfalle, von Sonnenlicht, Kohlendioxid, Wasser und Mineralien des Bodens ernähren. Doch jeden Morgen wacht sie auf und entscheidet sich aufs Neue für etwas anderes: Gewalt!
Natürlich könnte sich Dionaea muscipula, die Venusfliegenfalle, von Sonnenlicht, Kohlendioxid, Wasser und Mineralien des Bodens ernähren. Doch jeden Morgen wacht sie auf und entscheidet sich aufs Neue für etwas anderes: Gewalt! Oft heißt es in der Wissenschaft: Wichtiger als Antworten zu finden ist es, die richtigen Fragen zu stellen. Bleiben wir kurz dabei – und fragen unsererseits: Wodurch zeichnen sich „richtige Fragen“ aus? Wohl dadurch, dass sie den Weg zu klaren Experimenten ermöglichen, deren Resultate zu wichtigen neuen Erkenntnissen führen.
Oft heißt es in der Wissenschaft: Wichtiger als Antworten zu finden ist es, die richtigen Fragen zu stellen. Bleiben wir kurz dabei – und fragen unsererseits: Wodurch zeichnen sich „richtige Fragen“ aus? Wohl dadurch, dass sie den Weg zu klaren Experimenten ermöglichen, deren Resultate zu wichtigen neuen Erkenntnissen führen. Klar, was unserem Urin die gelbe Farbe gibt – das weiß man bereits seit über hundert Jahren: Urobilin heißt der Farbstoff, und der entsteht seinerseits aus dem orangefarbenen Bilirubin.
Klar, was unserem Urin die gelbe Farbe gibt – das weiß man bereits seit über hundert Jahren: Urobilin heißt der Farbstoff, und der entsteht seinerseits aus dem orangefarbenen Bilirubin.
 Ein wenig stiefmütterlich scheint die Forschungsgemeinde bislang hingegen den Faktor Zeit ins Kalkül genommen zu haben. Oder genauer gesagt: Wie vorgegebene Zeitspannen für das Einrichten konkreter Regulationsmechanismen genutzt werden.
Ein wenig stiefmütterlich scheint die Forschungsgemeinde bislang hingegen den Faktor Zeit ins Kalkül genommen zu haben. Oder genauer gesagt: Wie vorgegebene Zeitspannen für das Einrichten konkreter Regulationsmechanismen genutzt werden. Microsoft hat es endlich getan: Seit einigen Wochen kann man in deren Tabellenkalkulationsprogramm Excel Daten eingeben, ohne dass die eingebaute automatische Datenkonvertierung diese notorisch ungewollt verunstaltet – eine Funktion, die der biomedizinischen Forschung sehr lange durchaus große Schäden zugefügt hat.
Microsoft hat es endlich getan: Seit einigen Wochen kann man in deren Tabellenkalkulationsprogramm Excel Daten eingeben, ohne dass die eingebaute automatische Datenkonvertierung diese notorisch ungewollt verunstaltet – eine Funktion, die der biomedizinischen Forschung sehr lange durchaus große Schäden zugefügt hat. Morbus Kobold macht, dass Sie sich vor Schmerz zusammenkrümmen – auch wenn Sie selbst gar nicht von dem Krankheitsbild betroffen sind. Seien Sie also gewarnt und lesen Sie mit Bedacht weiter!
Morbus Kobold macht, dass Sie sich vor Schmerz zusammenkrümmen – auch wenn Sie selbst gar nicht von dem Krankheitsbild betroffen sind. Seien Sie also gewarnt und lesen Sie mit Bedacht weiter!
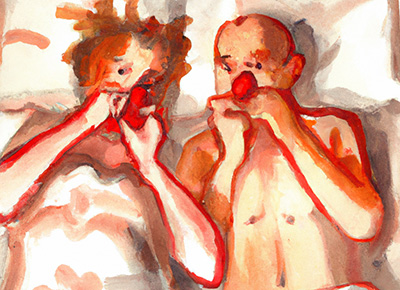 Kribbelt der Heuschnupfen auch mal wieder in Ihrem Rachen? Und Ihre Augen brennen? Sie brauchen nur ins Sonnenlicht zu zwinkern und schon entlädt sich – übrigens mit bis zu 160 Stundenkilometern – der nächste Nieser? In Zeiten zukünftiger Epidemien und Endemien und Pandemien vielleicht interessant zu wissen: Wie viele Meter schaffen Sie mit Ihrem Nasenauswurf? Das hängt ganz von der Schlagkraft Ihrer Sekret-Tröpfchen ab. Messen sie mehr als 100 Mikrometer im Durchmesser, folgen sie einer ballistischen Flugbahn. Dann ist nach etwa einem halben Meter Schluss. Zehnfach kleinere Partikel hingegen werden von Turbulenzen und Wirbeln der erzeugten Gaswolke weiter transportiert. Sie erreichen alles, was bis zu 2,50 Meter entfernt ist (J. Fluid Mech.
Kribbelt der Heuschnupfen auch mal wieder in Ihrem Rachen? Und Ihre Augen brennen? Sie brauchen nur ins Sonnenlicht zu zwinkern und schon entlädt sich – übrigens mit bis zu 160 Stundenkilometern – der nächste Nieser? In Zeiten zukünftiger Epidemien und Endemien und Pandemien vielleicht interessant zu wissen: Wie viele Meter schaffen Sie mit Ihrem Nasenauswurf? Das hängt ganz von der Schlagkraft Ihrer Sekret-Tröpfchen ab. Messen sie mehr als 100 Mikrometer im Durchmesser, folgen sie einer ballistischen Flugbahn. Dann ist nach etwa einem halben Meter Schluss. Zehnfach kleinere Partikel hingegen werden von Turbulenzen und Wirbeln der erzeugten Gaswolke weiter transportiert. Sie erreichen alles, was bis zu 2,50 Meter entfernt ist (J. Fluid Mech. 



