Immer wieder widersprechen gewisse Entdeckungen auf den ersten Blick jeglicher Erwartung oder Intuition – sodass ein großer Teil der Forscherwelt sie zunächst nicht glauben will. Als Paradebeispiel dient seit jeher die Maisgenetikerin Barbara McClintock, die vor achtzig Jahren herausfand, dass es im Erbgut mobile DNA-Elemente gibt, die nahezu beliebig von einer Stelle im Genom in eine andere hineinspringen können. Das Dogma vom stabilen Informationsträger DNA besagte damals, dass diese unbedingt unverändert an die nächste Generation weitergegeben werden müsse, da sonst das schiere Mutations-Chaos drohe. Folglich lieferte McClintock eine geradezu ungeheuerliche Erkenntnis, die dennoch in den folgenden Jahrzehnten klar bestätigt wurde. 1983 erhielt sie den Nobelpreis.
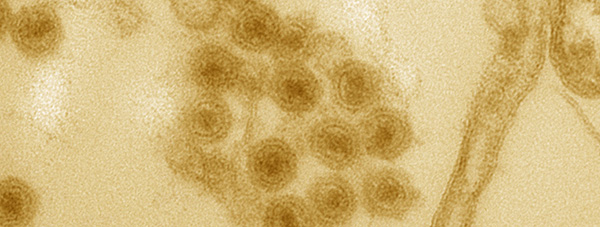
Mit den Standardmethoden der Virus-Jäger nicht zu fassen: Bakterien-mordende Autolykiviridae
Oft genug läuft es aber genau andersherum. Da entdeckt jemand etwas – und die Kollegenschaft reibt sich verwundert die Augen, warum man das nicht schon viel früher aufgespürt hatte.
So geschehen etwa bei der Entdeckung der sogenannten „kleinen RNAs“. Heute weiß man, dass die Zellen von Pflanzen und Tieren einen ganzen Zoo dieser kurzen RNA-Ketten aus meist zwischen 20 und 40 Nukleotiden produzieren. Ihre Entdeckung begann allerdings erst in den 1990er-Jahren. Was umso erstaunlicher war, als sich herausstellte, dass sie jede Menge Zellprozesse auf ganz entscheidende Weise mitsteuern.
Warum aber dieses „Spätzünden“ bei den kleinen RNAs? Weil die Forschung komplett auf ihre schon länger bekannten „großen Vettern“ fokussiert war – also Boten-, Transfer- und ribosomale RNA. Diese RNA-Moleküle bestehen aus deutlich längeren Nukleotid-Abfolgen und sind allesamt in den Prozessen der Proteinsynthese gemäß der Anleitung des genetischen Codes aktiv. Klar, dass sie aufgrund dieser zentralen Rolle mannigfach studiert wurden. Was dabei allerdings methodisch passierte, war folgendes:
Um diese bekannten RNA-Spezies isolieren und voneinander trennen zu können, entwickelten die Spezialisten die sogenannte RNA-Gel-Elektrophorese – und optimierten sie gezielt auf möglichst feine Trennung im Massebereich großer RNA-Moleküle. Doch was bedeutete die derart standardisierte Technik für die vielen bis dato unentdeckten kleinen RNAs? Sicherlich trieben sie oft genug neben ihren großen Vettern im selben RNA-Extrakt umher. Doch kaum auf ein RNA-Standardgel aufgetragen, schwammen sie während der Trennprozedur dem Rest des RNA-Gemischs aufgrund ihrer geringen Größe umgehend davon. Und bevor die großen Exemplare sich im Gel erst richtig voneinander trennten, flutschten die kleinen RNAs vom Experimentator unbemerkt an dessen anderem Ende schon wieder heraus. Oder Letzterer stoppte den Gel-Lauf vorher, interpretierte die Signale der kleinen RNAs „ganz vorne“ im Gel aber lediglich als Bruchstücke der großen RNA, die während der Prozedur entstanden waren. Schließlich sind RNA-Ketten besonders empfindliche Makromoleküle – und gerade außerhalb der Zelle immer und überall von schnellem Abbau bedroht.
Das Problem war also die Standardmethode. Zum Standard wird eine Methode immer dann, wenn man sie in allen möglichen Zusammenhängen stets für den gleichen Zweck einsetzt – wie eben RNA-Extrakte aus unterschiedlichster Herkunft auftrennen und analysieren. So gesehen ist eine Standardmethode also eigentlich was Schönes, auch weil sie zuverlässig reproduzierbare Resultate produziert. Doch aus unserem RNA-Beispiel wird ebenso klar: Immer kann es spezielle „Randgruppen“ geben, zu denen die Methode eben doch nicht passt – und die einem damit gerne durch die Lappen gehen.
Natürlich haben solche Standardmethoden der Forscherwelt nicht nur bei den kleinen RNAs diesen Streich gespielt. Nach dem gleichen Muster ist das auch öfter anderswo passiert – und passiert bis heute weiterhin.
So spürte etwa ein US-Forscherteam erst kürzlich eine große Gruppe von Viren auf, die massenhaft im Oberflächenwasser sämtlicher Meere treiben und dort jede Menge verschiedene Bakterien killen (Nature 554: 118-22). Kaum zu glauben, dass sie dennoch bis vor kurzem unentdeckt geblieben waren. Und nachvollziehbar, dass die Entdecker ihnen daher den Namen Autolykiviridae gaben – nach dem Meisterdieb Autolykos aus der griechischen Mythologie, der aufgrund seiner Gerissenheit nur schwer zu fangen war.
Tatsächlich zeichnen sich die Autolykiviridae durch einige durchaus ungewöhnliche Eigenschaften aus: So haben sie im Vergleich zu den meisten anderen Bakterien-mordenden, doppelsträngigen DNA-Viren keinen Schwanz und beherbergen überdies ein deutlich kleineres Genom mit großteils einzigartigen Genen. Doch warum sie den Virus-Jägern so lange entfleuchen konnten, liegt eher daran, dass sie gleich unter mehreren gängigen Standardmethoden der Virusjäger hindurchtauchen konnten:
- Da die Autolykiviridae-Partikel ungewöhnlicherweise von einer Lipid-Doppelschicht umhüllt werden, braucht man auch einen ungewöhnlich niedrigen Dichtebereich im Medium, um sie via Gleichgewichts-Dichtegradientzentrifugation aus einem Virus-Gemisch zu isolieren. Bei Verwendung der dichteren Standardmedien, die weithin zur Auftrennung verschiedener Virus-Partikel verwendet werden, blieben sie daher schlichtweg an der Oberfläche hängen.
- Auch die Standardanalysen von Virus-DNA, mit denen man die Zusammensetzung von Virusproben testet, versagten im Fall der Autolykiviridae. Denn im Gegensatz zu anderen Viren gaben die Autolykiviridae ihre DNA nur frei, wenn sie mit dem Enzym Protease behandelt wurden. Dieses jedoch fehlte in den gängigen Standardmedien zur Extraktion viraler DNA.
- Weiterhin vertrugen Autolykiviridae keine Behandlung mit Chloroform, mit der Viruspräparationen standardmäßig von kontaminierenden Bakterien befreit werden. Dummerweise ging damit auch deren eigene Bakterien-Infektivität in die Knie, sodass sie in anschließenden Standardtests auf Bakterien-zerstörende Viren zwangsläufig gar nicht auffallen konnten.
- Wenn sie bei einem solchen Test dennoch mal ohne vorherige Chloroform-Behandlung in eine mit Bakterien zugewachsene Schale getropft wurden, brauchten sie viel länger als andere Bakterien-Killer, um sichtbare Löcher in den Bakterienrasen zu fressen – und damit auch viel länger, als die Forscher die Schalen dafür standardmäßig im Brutschrank lassen.
Kein Wunder, mahnten die Autolykiviridae-Entdecker am Ende generell an, die Verwendung jeglicher Standardmethoden im Einzelfall stets kritisch zu prüfen! Schließlich deutet die Geschichte der kleinen RNAs und der Autolykiviridae darauf hin, dass noch einiges andere mehr unbemerkt unter dem Radar solcher Standardmethoden hindurchfliegen könnte.
Von daher könnte es durchaus bald wieder heißen: Warum haben wir das jetzt erst entdeckt?
Ralf Neumann
(Foto: Kauffman et al., Nature)
Schlagworte: Entdeckung, experimentelle Methode, kleine RNAs, mobiles DNA-Element, Standardmethode, Transposon, Virus





