Zusammenfassung aktueller Sequenziertechniken
Karin Hollricher
(13.09.2021) In aller Kürze: Illumina - im Nahfeld: PacBio - im Kanal: Oxford Nanopore Technologies (ONT) - im elektrischen Feld: Genapsys - in der optischen Karte: Bionano Genomics.
In aller Kürze: Illumina
Illumina dominiert mit etwa achtzig Prozent den weltweiten Sequenziermarkt, in den USA liegt der Anteil noch etwas höher. Illumina-Sequenzierer lesen 50 bis 300 bp lange Fragmente (Short Reads). Dafür werden die Einzelstrang-Fragmente an beiden Enden mit jeweils unterschiedlichen Adaptern versehen, die als Sequenzierprimer und zur Fixierung auf dem Chip dienen. Die Fragmente werden auf Flow Cells über Brücken-PCR vervielfältigt, wodurch Cluster mit identischen Fragmenten entstehen. Die Sequenzierung folgt dem Prinzip des Sequencing by Synthesis (SBS) mit fluoreszenzmarkierten Terminator-Nukleotiden. Wird das passende Nukleotid eingebaut, bricht die Synthese ab, die Fluoreszenzsignale werden optisch ausgelesen, die Terminatorgruppe entfernt, und es geht in die zweite Runde.
Auf diese Weise können Abermillionen DNA-Cluster pro Flow Cell parallel sequenziert werden. Nacheinander werden beide DNA-Stränge sequenziert, bestenfalls durchgehend. Doch auch wenn ein Stück fehlt, weiß man, dass sie benachbart sein müssen. Diese sogenannte Paired-End-Sequenzierung erleichtert die Auswertung und erhöht die Genauigkeit der Daten. Lücken, die durch lange Strecken repetitiver DNA entstehen können, lassen sich damit aber nicht schließen.
Zu Beginn arbeitete Illumina mit vier Fluorophoren. In der neuen Geräte-Generation reichen jedoch ein oder zwei Farbstoffe, die statt vier nur zwei Bilder liefern. Bei der 2-Kanal-SBS-Sequenzierung wird zum Beispiel Thymin (T) mit einem grünen und Cytosin (C) mit einem roten Fluorophor markiert. Adenin (A) erhält beide Farbstoffe, Guanin (G) wird nicht markiert. Auf dem Kanal, der rot detektiert, leuchten also A und C, auf dem grünen Kanal A und T. Leuchtet gar nichts, wurde G eingebaut. Die 1-Kanal-Chemie arbeitet nur mit einem grünen Fluorophor. G wird nicht markiert, T und A fluoreszieren grün (erstes Bild). Das Fluorophor von A lässt sich wieder abspalten, diese Reaktion kann man auf dem zweiten Bild sehen, hier wäre A dann dunkel. C trägt einen Adapter für das Fluorophor. Gibt man den Farbstoff nach dem ersten Bild hinzu, leuchtet C auf.
Im Nahfeld: PacBio
PacBios PCR-freie Technologie zur Sequenzierung sehr langer DNA- oder mRNA-Moleküle basiert auf der sogenannten Single-Molecule-Real-Time(SMRT)-Sequenzierung – einer Variante des SBS-Verfahrens. An einer einzelnen SMRT-Zelle können die Sequenzen von Millionen Molekülen gleichzeitig analysiert werden. Diese Sequenzierttechnik wurde von Jonas Korlach und seinen Mitstreitern an der Cornell University Ende der Neunzigerjahre entwickelt (Science 323: 133-8). Seit 2011 wird sie von der US-Firma PacBio vertrieben und weiterentwickelt, die Korlachs Laborkollege und Mitentwickler der SMRT-Technik Stephen Turner 2003 gründete.
Für die SMRT-Sequenzierung wird sehr hochmolekulare DNA auf einen Chip gegeben, auf dem sich Millionen winziger Vertiefungen befinden, in denen jeweils ein einzelnes Molekül DNA-Polymerase untergebracht ist. Jede Polymerase „greift“ sich ein DNA-Molekül und synthetisiert einen neuen Strang. Immer wenn sie ein fluoreszenzmarkiertes Nukleotid an den neuen Strang heftet, entsteht ein Lichtsignal. Die fortlaufende Inkorporation neuer Nukleotide wird in Echtzeit optisch verfolgt, indem die Signale im sogenannten evaneszenten Feld (dem Nahfeld) direkt unterhalb der Probe mit speziellen Wellenleitern detektiert werden. Dieser Modus, mit dem man über 50 kb am Stück sequenzieren kann, wird als Continuous Long Read (CLR) Sequencing bezeichnet.
PacBio offeriert aber auch einen zweiten Modus: Bei diesem werden die DNA-Moleküle zunächst mithilfe von Adaptern zirkularisiert. Jede Polymerase kann das ringförmige Template durchlaufen und an jedem Molekül mehrfach neue Stränge synthetisieren. Diese Technik nennt PacBio Circular Consensus Sequencing (CCS) oder auch HiFi-Reads. Mit ihr lassen sich zwar nur wenige Kilobasen lange Fragmente sequenzieren, man erhält aber genauere Reads als mit der CLR-Methode, denn die Fehler werden durch das mehrfache Lesen derselben Sequenz ausgemerzt. In beiden Modi beschränkt die Lebensdauer der Polymerase die Gesamtlänge der maximalen Sequenz. Übrigens: Illumina wollte PacBio 2018 für etwas mehr als eine Milliarde US-Dollar übernehmen. Den Deal untersagte aber die US-Wettbewerbsbehörde (Federal Trade Commission) Ende 2019, weil Illumina damit einen Mitbewerber eliminieren und sein Monopol auf dem US-Markt weiter ausbauen würde.
Im Kanal: Oxford Nanopore Technologies (ONT)
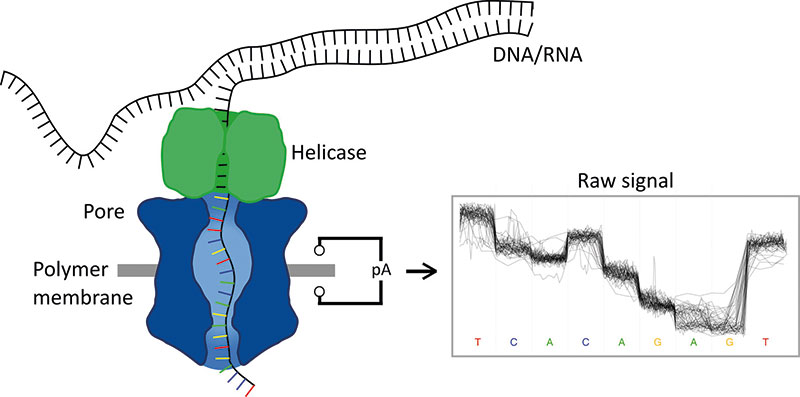
Die verrückt anmutende Idee, Nukleinsäuren in einem biologischen Kanal (Nanopore) ohne Amplifikation und Neusynthese und – im Falle von RNA – ohne reverse Transkription zu sequenzieren, hatten unabhängig voneinander verschiedene Forscher Ende der Achtziger- beziehungsweise Anfang der Neunzigerjahre. Ausgangsmaterial für die Nanoporen-Sequenzierung sind lange doppelsträngige (ds)DNA-Fragmente, deren Enden zunächst mit einer Helicase verknüpft werden. Die DNA wird danach auf eine nicht-leitende Membran aufgetragen, deren Nanoporen jeweils mit Elektroden an einen Sensor-Chip angeschlossen sind. Liegt an der Membran eine Spannung an, kann der Strom nur durch die Nanoporen fließen. Im elektrischen Feld bewegen sich auch die DNA-Stränge in Richtung Nanoporen, an die sie über die Helicase andocken. Die Helicase entwindet den Doppelstrang und schleust einen Einzelstrang in die Nanopore. Beim Tunneln der Nanopore verändert dieser den Ionenfluss im Kanal auf eine Weise, die spezifisch ist für jedes der vier Nukleotide. Die hierdurch induzierten individuellen Spannungsänderungen werden gemessen und in DNA-Sequenzen umgewandelt.
Die Nanoporen-Sequenzierung ermöglicht extrem lange Reads – ONTs Rekord liegt derzeit bei über 4 Mb. Es sollten allerdings viele Jahre ins Land ziehen, bis das erste Instrument auf den Markt kam – schön nachzulesen in einem Review (Nat. Biotechnol. 34: 518-24). 2015 demonstrierte ONT die Leistungsfähigkeit seines MinION-Sequenzierers mit der De-novo-Sequenzierung eines E.-coli-Genoms: Es war zu 99,5 Prozent korrekt im Vergleich zu publizierten Referenzen. Forscher setzen Mini-Nanoporen-Sequenzierer häufig in Feldstudien ein, etwa bei Ebola-Ausbrüchen in Afrika, oder auch zur Analyse von SARS-CoV-2.
ONTs Nanoporen-Technologie ist aber noch fehleranfällig, weil Informationen über einzelne Basen verloren gehen können. Daher gehören Deletionen zu den häufigsten Fehlern, die beim Nanoporen-Sequenzieren entstehen. Ein vielleicht noch größeres Problem ist, dass mehrere Nukleotide zu einem Spannungssignal beitragen können. Man kann dies zwar mithilfe klug gestalteter Software herausrechnen – aber nur, wenn keine längeren Sequenzen gleicher Nukleotide vorliegen. Im Mai verkündete ONT, die Sequenzierqualität mit einer verbesserten Chemie steigern zu können. Die Duplex-Sequenzierung soll die Fehlerrate ebenfalls weiter reduzieren. Bei dieser wird der komplementäre Strang direkt nach dem Template-Strang sequenziert.
Neben ONT entwickeln auch andere Firmen, beispielsweise das in San Francisco beheimatete Start-up Quantapore, Nanoporen-Sequenzierer. Und auch der Pharma-Riese Roche will nach dem offensichtlichen Misserfolg mit der 2014 übernommenen Nanoporen-Firma Genia Technologies endlich einen Fuß in das vielversprechende Geschäft mit Nanoporen-Sequenzierern bringen. 2020 erwarb Roche dafür die US-Firma Stratos Genomics, die das sogenannte Sequencing-by-eXpansion(SBX)-Verfahren erfand. Bei diesem wird der Abstand zwischen zwei Basen künstlich verlängert, um zu verhindern, dass mehrere Nukleotide gleichzeitig ein einzelnes Signal liefern. Hierzu wird der originale DNA-Strang mithilfe von vier sogenannten expandable NTPs (X-NTPs) kopiert, die jeweils zu einem der natürlichen Nukleotide passen. Jedes einzelne X-NTP ist mit einem schlauchförmigen Molekül verbunden, das wie ein kleiner Fallschirm an ihm hängt. Binden die X-NTPs an die korrespondierende Nukleinbase des Originalstrangs, werden sie von einer speziellen Polymerase zu einem Xpandomer verknüpft. In diesem ist der Abstand zwischen den einzelnen Nukleotiden durch die schlauchförmig dazwischenhängenden Moleküle viel weiter als in der Original-DNA. Durch die größere physikalische Distanz zwischen zwei Xpandomer-Basen schlüpfen diese einzeln durch die Nanopore und liefern ein genaueres und weniger fehlerhaftes Signal – so ist zumindest der Plan. Anfragen zum gegenwärtigen Stand des SBX-Projekts wurden von Roche bis zum Redaktionsschluss nicht beantwortet.
Im elektrischen Feld: Genapsys
Genapsys ist ein Newcomer im hart umkämpften Sequenziermarkt. Das erste Paper, in dem die Sequenzier-Methode der US-Firma beschrieben wird, ist noch nicht evaluiert (bioRxiv, doi: 10.1101/604553). Die Geräte des 2010 gegründeten Start-ups kann man aber bereits kaufen. Die Technik basiert auf der Sequenzierung klonaler, an Beads vermehrten DNA-Fragmenten. Das Verfahren ähnelt älteren Technologien, etwa 454 und Ion Torrent. Im Gegensatz zu diesen werden die Kügelchen aber nicht in echten Vertiefungen abgelegt, sondern in „virtuellen“: Sie werden magnetisch „fixiert“, und die Reagenzien werden mittels eines elektrischen Felds dorthin gebracht, wo man sie benötigt.
Wie das genau funktioniert, wird im Preprint nicht erklärt, da müsste man sich vermutlich durch die Patentschriften durchquälen. Auch die Detektion der Nukleinbasen funktioniert elektronisch. Genapsys verwendet hierzu CMOS-Chips mit mehreren Millionen einzelner Sensoren, die jeweils die Reaktion an einem einzelnen Kügelchen als Änderung des elektrischen Widerstandes detektieren, die von der Art des eingebauten Nukleotids abhängt. In dem bioRxiv-Preprint des Genapsys-Gründers Hesaam Esfandyarpou und seinen Kollegen heißt es, die Plattform könne 1,5 Gigabasen pro Lauf generieren und lese Short Reads von maximal 175 bp. Das kleine tragbare Gerät soll knapp 10.000 US-Dollar kosten. Es ist für gezieltes Sequenzieren (einer Region, eines kleineren Abschnitts eines Genoms in Diagnostik und im Labor), zur Untersuchung kleiner Genome (Mikroorganismen) und für die Transkriptomanalyse (Sequenzierung von RNA-Populationen) gedacht. Im Mai erzielte die Firma mit der dritten Finanzierungsrunde siebzig Millionen US-Dollar.
In der optischen Karte: Bionano Genomics
Das Optical Mapping von Bionano Genomics ist eigentlich keine neue Sequenziertechnologie – es ist eher eine Art Fluoreszenz-in-situ-Hybridisierung (FISH), also eine mit optischen Mitteln sichtbar gemachte Hybridisierung. Aber die Technik hilft, Fragmente von de novo sequenzierten Genomen anzuordnen und Strukturvarianten zu identifizieren. Insofern unterstützt Optical Mapping die Sequenzierung von Genomen. 2012 beschrieben Forscher der University of California San Diego und von Bionano Genomics das Prinzip der optischen Mapping-Technologie im Hochdurchsatz-Verfahren (Nat. Biotech. 30: 771-6). Bei diesem hybridisiert man hochmolekulare über 150 kb lange DNA-Moleküle mit markierten Oligos, deren komplementäre Sequenzen über das gesamte Genom verteilt sind und sehr häufig vorkommen. Bionano Genomics verwendet ein sechs Basenpaare langes Motiv, das 15-mal pro 100 kb vorhanden ist. Diese DNA-Mixtur gibt man auf Flow Chips, in die winzige Kanäle mit einem Durchmesser von 45 Nanometern eingeätzt sind. In jeden Kanal kann sich jeweils nur ein DNA-Molekül der Länge nach einfädeln – die Moleküle können sich nicht zurückfalten, dafür sind die Kanäle zu eng.
Das Fluoreszenz-Muster wird optisch ausgelesen und ähnelt einem Barcode. Anhand dieses Barcodes lassen sich die Positionen neuer DNA-Fragmente identifizieren. Vergleichende Analysen, etwa der DNA von erkrankten Kindern und ihren Eltern oder ein Vergleich mit vorhandenen Genomsequenzen identifizieren größere Strukturvarianten wie Deletionen, Insertionen, Inversionen und Translokationen. Mehrere Proof-of-Principle-Studien kamen zu dem Ergebnis, dass sich mit der Technik molekulare Veränderungen als Ursache für Erkrankungen erkennen lassen. Ein entsprechendes Experiment dauert nach Firmenangabe nicht einmal vier Tage – vorausgesetzt man beherrscht die Technologie. Anscheinend ist die Präparation der DNA nicht trivial. Aber Übung macht bekanntlich ja den Meister. Auf seiner Webseite stellt Bionano Genomics viele Vorträge und auch neueste und wichtige Publikationen zur Verfügung, die die Technologie genauer erläutern und ihre Verwendung erklären.