Schnell produziert
(27.11.2019) Die Synthese langer Einzelstrang-DNA mit Standard-Verfahren ist umständlich und teuer. Mit einer neuen Technik ist sie aber beinahe ein Kinderspiel und ruckzuck erledigt.
Die Nachfrage nach einzelsträngiger DNA (ssDNA) für Anwendungen in der synthetischen Biologie, biologischen Bildgebung und Bionanotechnologie ist groß. Und natürlich soll die Synthese möglichst wenig kosten und schnell gehen. Für kurze ssDNA-Sequenzen ist dies auch tatsächlich der Fall – komplizierter und vor allem teurer wird es jedoch ab einer Länge von 200 Nukleotiden. Hier sind entweder zusätzliche Aufbereitungsschritte nötig, um Verunreinigungen zu entfernen, oder man muss über doppelsträngige DNA an ssDNA herankommen: etwa durch enzymatischen Verdau von PCR-Produkten, funktionalisierte Magnet-Beads, Rolling-Circle-Amplifikation, asymmetrische PCR sowie Co-Polymerisation und elektrophoretische Methoden.
So richtig überzeugen kann allerdings keine dieser Methoden: Sie sind entweder sehr kompliziert und nicht skalierbar oder die Reinheit der Produkte lässt zu wünschen übrig. Eine wesentlich elegantere Technik, mit der sich ssDNA mit einer Länge von bis zu sieben Kilobasenpaaren schnell, effizient und sauber in vitro herstellen lässt, dachten sich William Shih und sein Team von der Harvard University Boston aus.
PCR mit Polymer-Primer
Die Gruppe stellte hierzu Primer mit einem linearen Polyacrylamid-Co-Acrylat-Tag her. Die getaggten Primer setzten Shihs Mitarbeiter anschließend in einer gewöhnlichen PCR, zusammen mit unmarkierten komplementären Primern ein. Sie erhielten hierdurch Polymer-getaggte Amplikons, deren Stränge sich voneinander trennen ließen. Dazu fällten die Forscher die DNA selektiv, indem sie NaOH zugaben und die Lösung anschließend mit Methanol mischten.
Nach der Zentrifugation extrahierte die Gruppe zunächst den unmarkierten Strang aus dem Überstand. Anschließend resuspendierte sie das Pellet mit der Polymer-DNA und versetzte die Suspension mit Uracil-DNA-Glycosylase. Um auch den markierten Strang isolieren zu können, hatten die Forscher eine Desoxyuridin-Base in die modifizierten Primer eingebaut. Mithilfe der Uracil-DNA-Glycosylase erzeugten sie eine abasische Stelle mit einer fehlenden Uracil-Base, an der sie den Strang anschließend mit Dimethylethylendiamin spalteten. Im letzten Schritt entfernte das Team schließlich überflüssige Polymer-getaggte DNA mit einer weiteren Methanol-Fällung. Das ganze Prozedere dauerte nicht länger als 90 Minuten, die PCR-Reaktion nicht mitgerechnet.
Mehr als 3.000 Nukleotide lang
Shihs Team erzeugte mit der neuen Methode, die es Methanol-Responsive Polymer-PCR (MeRPy-PCR) nennt, ssDNA mit Längen zwischen 89 und 3.115 Nukleotiden. Das Protokoll war für alle gewählten Längen und DNA-Templates nahezu identisch. Geringe Abweichungen gab es nur bei der alkalischen Denaturierung der längsten Amplikons. Die Ausbeute für den unmarkierten Strang lag bei 70 bis 90 Prozent, beziehungsweise bei bis zu 70 pmol pro 100 µl PCR-Reaktion. Etwas geringer fiel sie beim markierten Strang aus, vor allem bei langen Amplikons. Die MeRPy-PCR punktet aber auch bei der Reinheit der erhaltenen ssDNA. Sie war genauso hoch oder sogar höher wie bei einer vergleichbaren chemischen oder enzymatischen ssDNA-Synthese mit konventionellen Methoden.
Die Vorteile der neuen Methode demonstrierte das Team anhand von ssDNAs, die es für CRISPR/Cas9-vermittelte homologe Rekombination (HDR), Fluoreszenz-in-situ-Hybridisierung (FISH) und DNA-Origami einsetzte. Im HDR-Test wurden die ssDNAs verwendet, um Stopp-Codons aus einem nicht funktionellen GFP-Expressionsvektor herauszuschneiden. Die erzeugten MeRPy-ssDNA-Donorsequenzen mit 200 bis 1.000 Nukleotiden schnitten hier genauso gut ab wie eine chemisch synthetisierte 200 Nukleotide lange Donorsequenz.
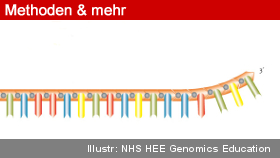
Auch als Gerüst geeignet
Auch beim Origami-Test überzeugte die MeRPy-ssDNA. Mit der DNA-Origami-Technik lassen sich aus DNA-Strängen zwei- und dreidimensionale Strukturen formen, wobei eine lange ssDNA als Gerüststrang dient. Shihs Mitarbeiter stellten mit der MeRPy-PCR ssDNAs mit 3.315 und sogar 7.308 nt aus M13-Phagen her, die sich als Gerüst-DNA eigneten.
Für die Synthese von FISH-Sonden hefteten die Forscher einen zusätzlichen Cy3-Tag an die Primer.
Miriam Colindres
Minev D. et al. (2019): Rapid in vitro production of single-stranded DNA. Nucleic Acids Research, DOI: 10.1093/nar/gkz998