Das Schweigen der Gene
Tobias Ludwig
(09.11.2022) KARLSRUHE: Die Gruppe um den Molekularbiologen Holger Puchta legt Chromosomen genetisch still und gibt so Pflanzenzüchtern ein neues Werkzeug an die Hand.
Schon lange vor den Entdeckungen Gregor Mendels im 19. Jahrhundert veränderten die Menschen durch Kreuzung und Selek-
tion die Eigenschaften von Pflanzen. Der Augustinermönch Mendel war es jedoch, der der Kreuzung und ihren Auswirkungen ein wissenschaftliches Fundament gab, als er in den 1860er-Jahren seine Vererbungsgesetze formulierte. Seit dieser Zeit haben immer neue molekulargenetische Errungenschaften wie zuletzt CRISPR/Cas das Methodenspektrum der Pflanzenzucht stetig erweitert. Die Forschungsgruppe um Holger Puchta vom Karlsruher Institut für Technologie (KIT) hat diesem Werkzeugkoffer nun eine neues Instrument hinzugefügt. Ihre Ergebnisse veröffentlichten die Karlsruher kürzlich in
Nature Plants (
doi:10/gqt7xj).
„Mich haben schon immer die Pflanzen stärker interessiert“, erzählt Michelle Rönspies. Sie ist Doktorandin in Puchtas Gruppe am KIT und Erstautorin der Studie. Nach einem Praktikum in der Genome-Engineering-Abteilung bei BASF im belgischen Ghent habe sie mit ihrer Doktorarbeit am Botanischen Institut des KIT begonnen.
Puchtas Weg zu den Pflanzen ist ungleich länger, jedoch nicht weniger zielstrebig. Der Inhaber des Lehrstuhls für Molekularbiologie und Biochemie begann seine wissenschaftliche Karriere Ende der 1970er-Jahre in Tübingen. „Als Tübinger Biochemie-Studenten hatten wir damals die Möglichkeit, für ein Jahr an das Max-Planck-Institut für Biochemie in München gehen zu können. Dort gab es eine Gruppe, die sich mit Gentechnik in Pflanzen beschäftigt hat“, erinnert er sich. Die Münchner waren damals eine der ersten Gruppen, die Viroide, also infektiöse RNA-Moleküle, die nur in höheren Pflanzen vorkommen, klonieren konnten. Puchtas Interesse war geweckt, und so beschäftigte er sich sowohl in seiner Diplom- als auch Doktorarbeit mit den kleinen RNA-Erregern. Nach Stationen in Basel und am Institut für Pflanzengenetik und Kulturpflanzenforschung in Gatersleben hat der Molekularbiologe nun schon seit 2002 seinen Karlsruher Lehrstuhl inne.
Gescheitert, aber viel gelernt
Angetrieben wurde seine Arbeit vor allem von einer bestimmten Idee: „Seit 30 Jahren war es mein Ziel, durch gezielte Induktion von DNA-Doppelstrangbrüchen die Meiose und damit auch den genetischen Austausch bei Pflanzen zu steuern.“ Die Kontrolle darüber, welche Eigenschaften vererbt werden und welche nicht, sei für Pflanzenzüchter sehr wichtig, jedoch nicht leicht zu erreichen. „Wenn zwei Gene auf einem Chromosom nah beieinander liegen, werden diese in der Regel auch zusammen vererbt. Die können Sie nicht trennen“, erläutert Puchta. Über mehrere Generationen von Doktoranden habe er sich diesem Ziel unter Verwendung immer neuer Nukleasen verschrieben und sei – wie er selbst sagt – kläglich gescheitert. Umsonst war die Arbeit jedoch keineswegs. „Wir haben im Laufe der Zeit gelernt, dass wir Rekombination auch durch eine Restrukturierung von Chromosomen beeinflussen können.“
Rönspies führt weiter aus: „Während der Evolution kommt es immer wieder zu chromosomalen Umstrukturierungen, und es ist schon länger bekannt, dass etwa Inversionen Cross-over unterdrücken“. Treten an einer bestimmten Stelle der Chromosomen keine Cross-over auf, kommt es nicht zu einer Rekombination des genetischen Materials. Kreuzt man Inversions-tragende Pflanzen mit Wildtypen ohne Inversion, können keine genetischen Informationen zwischen dem invertierten und nicht-invertierten Abschnitt ausgetauscht werden. Erst mit Aufkommen der Long-Read-Sequencing-Technologien in den letzten fünf Jahren habe man nachweisen können, wie stark verbreitet Inversionen in Kulturpflanzen eigentlich sind, so Puchta. „Bis dahin haben die Züchter einfach nicht verstanden, wieso sie bestimmte Eigenschaften nicht miteinander kombinieren konnten.“
Die Drehung macht‘s
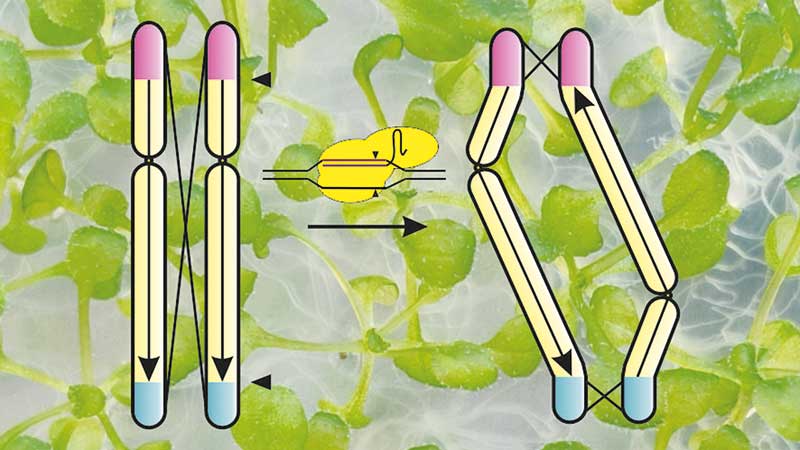
Durch eine gezielte Neuordnung chromosomaler Abschnitte müsste sich also das Rekombinationsverhalten der Pflanzen steuern lassen. Dies konnten die Karlsruher als Erste im Jahr 2020 zeigen. Dem Team um Puchta war es gelungen, eine bekannte Inversion im Genom der Ackerschmalwand Arabidopsis thaliana wieder in ihre Ursprungsstellung zurückzudrehen. Wurden die so behandelten Pflanzen mit dem Wildtyp gekreuzt, zeigte sich ein reger genetischer Austausch an einer Stelle, die zuvor stillgelegt war (Nat. Commun., doi:10/jhkk).
In ihrer kürzlich in Nature Plants veröffentlichten Studie ging das Forschungsteam noch einen Schritt weiter. „Nachdem das Umdrehen der verhältnismäßig kleinen Inversion so gut geklappt hat, dachten wir, jetzt machen wir was ganz Verrücktes und versuchen, ein ganzes Chromosom stillzulegen“, schildert Puchta. Statt eines 1,1 Megabasen großen Fragmentes drehten die Karlsruher fast das gesamte Chromosom 2 der Ackerschmalwand um – immerhin über 17 Megabasen und damit etwa ein Zehntel des gesamten Genoms der Pflanze. Doch wie dreht man ein ganzes Chromosom? „Genauso wie man kleine Abschnitte dreht,“ sagt Puchta und grinst. Dazu induzierte die Arbeitsgruppe mittels CRISPR/Cas9 des Bakteriums Staphylococcus aureus Doppelstrangbrüche in der DNA der Kulturpflanze. Nur 2 beziehungsweise 0,5 Megabasen der jeweiligen Chromosomen-Enden blieben unangetastet. Um die Pflanzen zu identifizieren, bei denen die gewünschte Inversion auftrat, verwendeten die Karlsruher ein sogenanntes Pooling-Verfahren. „Aus den Blättern von je 40 Pflanzen haben wir DNA extrahiert und auf das Vorhandensein der Inversion getestet. Es war nötig, sehr viele Transformanden zu screenen, da die Effizienz der Umstrukturierung erfahrungsgemäß nur bei etwa 0,1 Prozent liegt“, erläutert Rönspies. Insgesamt kamen 1.600 Pflanzen unter die Lupe – darunter befand sich nur eine positive Transformande. Dabei zeigten Nachkömmlinge mit dem gedrehten Chromosom 2 keinerlei Wachstums- oder Fortpflanzungsdefizite im homozygoten Zustand. „Die Gene werden trotzdem normal abgelesen, lediglich bei der Meiose verhält sich dieser Abschnitt anders“, so Molekularbiologe Puchta.
Was nicht passt, bleibt still
Kommt die Inversion jedoch nur von einer der beiden Elternpflanzen, sieht die Situation schon etwas anders aus. „Die Fertilität bei hemizygoten Pflanzen war etwa um ein Drittel reduziert“, fasst Rönspies ihre Ergebnisse zusammen. Dies sei laut Puchta wahrscheinlich ungünstigen Chromosomen-Paarungen geschuldet, die sich bei der Anlagerung von invertiertem und nicht-invertiertem Chromosom ergeben. Eine Vermutung der Karlsruher ist, dass sich die Chromosomen nur an den unveränderten Telomeren zuverlässig paaren. Dort konnten Puchta und Co. auch Cross-over detektieren. Eine weitere mögliche Variante ist, dass sich eines der beiden eigentlich homologen Chromosomen dreht. „Dann passen zwar die inneren 17 Megabasen zusammen, aber die Telomere nicht“, sagt Rönspies. In diesem Fall sei dann außerhalb der Telomer-Region auch ein einfacher Cross-over möglich, dieser resultiere jedoch in einem großen Verlust genetischer Information, was zu nicht-lebensfähigen Gameten führt.
Anders sieht dies bei doppeltem Cross-over aus: „Normalerweise kommen hauptsächlich einfache Cross-over vor. Dabei bricht ein Teil oder der ganze Arm eines Chromosoms ab und wird getauscht. Bei doppelten Cross-over kommt es jedoch nur zu einem Austausch zwischen den beiden Kreuzungspunkten. Dabei geht also keine genetische Information verloren, weil die ursprünglichen Telomer-Enden erhalten bleiben.“ Folglich gehen die Molekularbiologen um Puchta davon aus, dass es zwar schon zu meiotischer Rekombination in den invertierten Abschnitten kommt, diese aber seltener lebensfähige Gameten hervorbringt. Daher die Reduktion der Fertilität. „Aber es klappt noch“, wirft Puchta ein. „Diese Prozesse kommen natürlich auch während der Evolution vor. Es gibt sogar Pflanzen mit mehrfachen Chromosomen-Sätzen, und anders als bei Säugetieren werden solche Anomalien bei Pflanzen gut toleriert.“
Viele Pläne für die Zukunft
Die Methode könne laut Puchta dazu eingesetzt werden, mehrere Eigenschaften gleichzeitig neu in eine Kulturpflanze einzuführen. Auch wäre es möglich, Gene, die im Laufe der Domestizierung der Pflanzen durch chromosomale Umstrukturierungen stillgelegt wurden, für eine Rekombination wieder verfügbar zu machen. „Dieses ‚Chromosome Engineering‘ wird andere gentechnische Verfahren nicht ersetzen, kann aber eine große Rolle spielen, wenn Züchter Pflanzen kreuzen wollen.“ Zudem, so zumindest die Hoffnung des Molekularbiologen, seien derartig veränderte Pflanzen von ihren „natürlich“ entstandenen Pendants nicht zu unterscheiden, was sie als nicht gentechnisch veränderte Organismen qualifizieren müsste. Ein wichtiger Punkt für eine potenzielle kommerzielle Anwendung der Karlsruher Technik.
Eine der nächsten offenen Fragen, die die Karlsruher Molekularbiologen beantworten möchten, ist, ob sich die Centromere in Chromosomen versetzen lassen. Rönspies interessiert sich derzeit für die Möglichkeit einer Chromosomen-Fusion, also der Übertragung großer Chromosomen-Abschnitte, wie etwa ganzer Arme, auf ein anderes Chromosom. „Letztlich geht es darum, die Anzahl der Chromosomen zu verändern. Dann hätte man eine ganz andere Gruppe von Genen, die stets gemeinsam vererbt werden“, skizziert Puchta. In der etwas ferneren Zukunft sei die Schaffung nicht-auskreuzbarer Kulturpflanzen ein weiteres Ziel. So könne durch gezielte Umstrukturierungen eine Vermischung mit Wildpflanzen komplett unterbunden werden. Es bleibt also spannend bei den Karlsruhern.