Künstlich transkribiert
(07.12.2022) Mit der DNA-Bindedomäne von dCas9 oder pflanzlichen Transkriptionsfaktoren lässt sich ein Expressionssystem basteln, das in Salmonella funktioniert.
Bakterien möglichst gut zu verstehen und manipulieren zu können, zahlt sich immer aus. Sei es, um E. coli zu biotechnologischen Höchstleistungen anzutreiben oder um Infektionsmechanismen und Antibiotika-Resistenzen in Salmonella Typhimurium zu verfolgen. Die Eingriffe dürfen die Mikroorganismen aber nicht zu sehr aus dem Gleichgewicht bringen – die Bakterien sollen genau das tun, was man will und sich ansonsten völlig normal entwickeln und verhalten.
Die synthetische Biologie bietet hierzu vielfältige Möglichkeiten, etwa künstliche Promotor-Elemente, die mit fremden Transkriptionsfaktoren kombiniert werden. Wie man sich dabei aus dem reichhaltigen Angebot der Natur bedienen kann, zeigt Marc Erhardts Gruppe von der Humboldt-Universität zu Berlin, die sich dafür mit Emmanuelle Charpentier zusammentat.
Fremdgesteuerte Genexpression
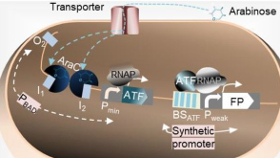
Um die Expression eines Gens in einem Mikroorganismus vollständig fremdzusteuern, benötigt man einen Promotor, einen Transkriptionsfaktor mit DNA-Binde- sowie Aktivierungsdomäne und das gewünschte Gen, beispielsweise ein Reportergen. Damit sich die Zelle nicht unnötig verausgabt, sollte der Transkriptionsfaktor aber erst nach Zugabe eines Signals aktiv werden. In E. coli eignet sich hierzu das Arabinose-induzierbare System, das gut charakterisiert ist und von vielen Forschern und Forscherinnen genutzt wird.
Arabinose gelangt über den Transporter AraE in die Zelle und wird von dem Regulator(repressor)protein AraC empfangen. AraC, das als Dimer den Arabinose-induzierbaren araBAD-Promotor (PBAD-Promotor) blockiert, ändert hierdurch seine Konfiguration, wodurch die RNA-Polymerase (RNAP) Zugang zu PBAD erhält – die Transkription der von PBAD gesteuerten Enzyme AraB, AraA und AraD kann beginnen.
Spielverderber entfernen
Die Arabinose-induzierte Genexpression via PBAD lässt sich aber auch verwenden, um die Expression eines gewünschten Gens anzukurbeln. Die Berliner checkten zunächst, ob das E.-coli-System auch in Salmonella funktioniert und nutzten GFP als Reportergen. Mit dem Durchflusszytometer beobachteten sie, dass die Salmonellen (Salmonella enterica, serovar Typhimurium) auf Arabinose-Konzentrationen zwischen 0,00001 und 0,2 Prozent reagierten und fluoreszierten. Schon mit 0,00005 Prozent Arabinose erreichte die Induktion eines Plasmid-Konstrukts den maximalen Wert, bei einem chromosomalen Konstrukt waren dazu 0,05 Prozent Arabinose nötig.
Mit der Aktivität des Reportergens war das Team aber noch nicht zufrieden. Um zu verhindern, dass die Expression durch den natürlichen Arabinose-Stoffwechsel gestört wird, beseitigte es das AraBAD-Operon als potenziellen Spielverderber. Die Mutanten können mit Arabinose nichts mehr anfangen, die Regulation von PBAD durch Arabinose via AraC bleibt jedoch erhalten. Damit Arabinose möglichst effizient in die Zellen gelangt und seine Signalfunktion erfüllen kann, exprimierten die Berliner den Transporter AraE von dem schwachen konstitutiven synthetischen Promotor ProA. Die Zellen sprachen danach über einen weiten Konzentrationsbereich auf Arabinose an. Wurde der GFP-Reporter vom Plasmid- und nicht von einem chromosomalen Konstrukt exprimiert, korrellierte die wachsende Arabinose-Konzentration (0,01 bis 0,1 Prozent) sehr genau mit den stärkeren GFP-Signalen.
Doppelt reguliert
Im nächsten Schritt platzierte Erhardts Mannschaft statt GFP einen künstlichen Transkriptionsfaktor (ATF) hinter den Arabinose-induzierbaren Promotor. ATF interagiert mit RNAP, bindet an die ihm zugewiesenen Bindestellen eines dahintergeschalteten synthetischen Promotors und treibt letzten Endes die Expression eines Reportergens an (siehe Bild). Der Witz dieses doppelt regulierten Systems besteht darin, maximale Kontrolle und Spezifität zu erlangen. Und vielleicht auch – so der Hintergedanke der Entwickler – höhere Expressionsraten. Als ATF-Komponenten verwendeten sie zunächst die DNA-Bindedomäne (DBD) von dCas9, die sie mit der Aktivierungsdomäne (AD) des bakteriellen Transkriptionsfaktors SoxS kombinierten, die sich bereits in Bakterien bewährt hatte (Nat Commun, 9(1):2489).
Eine scRNA führt den künstlichen Transkriptionsfaktor zum synthetischen Promotor. Dazu wird die scRNA mit einer Haarnadelstruktur fusioniert und von einem starken konstitutiven Promoter exprimiert. Die SoxS-AD bindet über ein fusioniertes Anhängsel an die Haarnadelstruktur und kommt so mit der DBD von dCas9 in Kontakt. Das Arabinose-induzierbare System besteht somit aus drei Kassetten: zwei sind für die Expression von dCas9 sowie von SoxS-AD zuständig und werden von PBAD angetrieben. Die dritte, von einem konstitutiven Promotor angekurbelte, Kassette steuert die Expression von scRNA. In Gegenwart von Arabinose werden SoxS-AD und dCas9-DBD exprimiert, wodurch die ATF-Komponenten zustande kommen.
Exotischer Faktor
dCas9 stammt von Streptococcus pyogenes und ist ziemlich exotisch für die gramnegativen Vertreter E. coli oder Salmonella. Es geht aber noch ausgefallener. Die Gruppe fragte sich, ob auch eine DBD von einem pflanzlichen Transkriptionsfaktor für die Genexpression in dem gramnegativen Bakterium funktionieren würde. Dazu fusionierte sie die bakterielle Aktivierungsdomäne SoxS-AD mit dem pflanzlichen Transkriptionsfaktor JUB1. Der zugehörige synthetische Promotor enthielt mehrere hintereinandergesetzte Kopien des JUB1-Bindemotivs. Auch dieser ATF wurde von Arabinose aktiviert und trieb die Expression seines Zielgens an.
Das neuartige Regulationssystem ist auch für das metabolische Engineering in gramnegativen Bakterien geeignet. E.-coli-Zellen produzierten mit dem pflanzenbasierten ATF und einem entsprechenden Konstrukt beta-Carotin. Salmonella modelte die Gruppe mit dem ATF-System zu einem Alkaloid-Detektor um.
Andrea Pitzschke
Naseri G. et al. (2022): A regulatory toolkit of arabinose-inducible artificial transcription factors for Gram-negative bacteria. bioRxiv, DOI: 10.1101/2022.11.30.518220
Bild: Naseri et al.