Tipp 236: Elektrophorese - SuperBuffer wirklich besser?
(13.09.2021) Liebe Laborjournal-Redaktion, bezugnehmend auf den in Laborjournal 5/2021 auf Seite 64 erschienenen Tipps-und-Tricks-Artikel einer Salzburger Gruppe zu einem angeblichen SuperBuffer möchte ich auf eine etwas tiefer liegende Problematik bei der Elektrophorese aufmerksam machen, die in dem Artikel nicht beachtet wird.
Die Grundlagen der DNA-Elektrophorese wurden bereits in den Neunzigerjahren ausführlich diskutiert und beschrieben. Der Tipps- und-Tricks-Artikel sowie das dazugehörige Paper (Gene 487(1): 72-4) zeigen mir jedoch, dass dieses Know-how entweder nicht in den molekularbiologischen Laboren ankam oder das Wissen verloren gegangen ist.
Bitte erlauben Sie mir, dazu ein wenig auszuholen.
Elektrophorese und Chromatographie sind nach wie vor die wichtigsten Trennmethoden für Makromoleküle, auch wenn sie von der Massenspektrometrie Konkurrenz bekommen haben. Beide Techniken können präparativ eingesetzt werden, was insbesondere bei der Chromatographie häufig der Fall ist. Ihre Hauptanwendung ist jedoch die Analytik. Selbstverständlich möchte man auch in der Lage sein, die „Güte“ einer analytischen Methode zu beschreiben. Für die Chromatographie wurde dafür die Zahl der theoretischen Böden (Platten) als Größe eingeführt, die ursprünglich aus der fraktionierten Destillation stammt.
Für die Elektrophorese ist dieser Parameter weniger sinnvoll, weshalb hier bevorzugt auf die Auflösung (Resolution) zurückgegriffen wird. Diese ist definiert als der Abstand zweier Banden, oder Peaks, geteilt durch die mittlere Bandenbreite (Peakbreite). Bei einem Flachgel sind Abstand und Breite ein Längenmaß, bei der Kapillarelektrophorese (oder bei der Chromatographie) ein Zeitmaß. Insgesamt ist die Auflösung jedoch dimensionslos. Im Fall von DNA ist es zusätzlich sinnvoll, diesen Wert auf die Länge des betrachteten Moleküls zu beziehen – also Basenpaare (bp) oder b). Wer es eilig hat, kann das Ganze noch durch die Laufzeit dividieren, dann erhält man die Trennleistung eines Systems.
Der Peakabstand wird vom Verhalten des Makromoleküls im Gel bestimmt, das recht komplex sein kann. Auch dies wurde in den Jahren von circa 1985 bis 1990 intensiv erörtert. Es lohnt sich jedoch, den Peakabstand durch die Wahl des geeigneten Trennmediums (Gel, Polymerlösung) und die Wahl des DNA-„Zustandes“ (Einzelstrang oder Doppelstrang) zu optimieren.
Die Bandenbreite (Peakbreite) wird ebenfalls von mehreren Parametern beeinflusst, die wichtigsten sind Diffusion und Konvektion (durch die Erwärmung). Hierbei kommt es zu einem Zielkonflikt: Erhöht man die elektrische Feldstärke, wird die Laufzeit zwar kürzer und die Diffusion zeitlich begrenzt, gleichzeitig erhöht sich jedoch die Stromstärke, wodurch sich das Gel stärker erwärmt, was wiederum zu einer höheren Konvektion führt. Dies gilt es also zu optimieren.
Der beste Weg dazu ist, den Querschnitt des Trennmediums (Gel, Kapillare) so gering wie möglich zu halten sowie die Leitfähigkeit des Puffers zu reduzieren. Hier ist die Kapillarelektrophorese klar im Vorteil. Mit geringen Durchmessern, zum Beispiel 50 Mikrometern, und einer guten Wärmeabfuhr lässt sich der Effekt der Konvektion fast völlig unterdrücken.
Wenn der Biochemiker den Begriff „Puffer mit geringer Leitfähigkeit“ hört, denkt er sofort an zwitterionische Puffer beziehungsweise an „Goods“-Puffer. Und tatsächlich wird in der DNA-Kapillarelektrophorese zum Beispiel der TAPS-Puffer seit etwa 25 Jahren erfolgreich eingesetzt. Zusätzlich verzichtet man auf Na2-EDTA, wodurch sich die Leitfähigkeit des Puffers deutlich verringert. Auf diese Weise wird die Peakbreite fast ausschließlich von der Diffusion vorgegeben – damit war die Frage nach der maximalen Auflösung gelöst.
Diese Grundlagen wurden wie gesagt in den Neunzigerjahren erarbeitet. Anlass dafür waren vor allem die großen Sequenzierprojekte, etwa das Humangenomprojekt. Der Flaschenhals bei der klassischen Sequenzierung nach Sanger ist nämlich die elektrophoretische Trennung, was vielleicht nur den wenigsten bewusst ist. Es war deshalb sehr wichtig, diese Methode besser zu verstehen und zu optimieren. Mit dem Aufkommen von Next-Generation-Sequencing-Methoden hat sich dies natürlich weitgehend erledigt. Es war schlussendlich der bessere Weg, neue Sequenzier-Methoden zu entwickeln, bei denen der elektrophoretische Trennschritt komplett wegfällt.
Die oben beschriebenen Prinzipien gelten natürlich genauso für die Flachbett-Elektrophorese (Agarose, Polyacrylamid). Vermutlich aus Kostengründen werden hier jedoch kaum zwitterionische Puffer eingesetzt. Man behilft sich mit klassischen Puffern (Tris-Borsäure oder Tris-Acetat), die so verdünnt eingesetzt werden, dass ihre Leitfähigkeit möglichst gering ist, sie aber gleichzeitig noch genügend Pufferkapazität aufweisen.
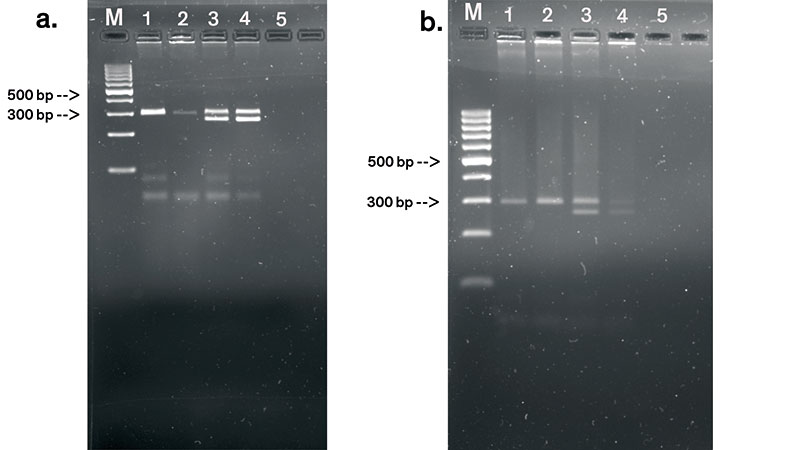
Ich habe mir einmal die Mühe gemacht, die Elektrophorese-Ergebnisse der Salzburger Gruppe genauer zu analysieren. Als Beispiel betrachte ich die Banden des Bandenpaars bei 300 bp und circa 400 bp (Bahn M), die etwa dieselbe Bandenbreite aufweisen. Das Nachmessen auf den Fotos mit einem Messschieber ist natürlich etwas „primitiv“, aber in Ermangelung eines Scans genau genug. Nach der Trennung mit dem SuperBuffer (Foto a) sind die beiden Fragmente 2,3 Millimeter voneinander entfernt, nach der Trennung mit TAE-Puffer (Foto b) jedoch 3,6 Millimeter, also 1,5-mal so weit. Die mittlere Bandenbreite beträgt im SuperBuffer 0,5 Millimeter, im TAE-Puffer 0,8 Millimeter, Letztere ist also 1,6-mal breiter. Die Auflösung ist somit dieselbe.
Der SuperBuffer ist also nur scheinbar besser.
Die Aussage: „Vor allem für das Publizieren von Ergebnissen sind aussagekräftige Bilder nötig, die scharfe, klar definierte Banden von DNA-Fragmenten zeigen“, ist Humbug. Das Gel soll dazu dienen, die Länge eines DNA-Fragments zu bestimmen oder zu überprüfen – und nicht dazu, einen Gutachter zu beeindrucken.
Ähnliches gilt auch für die Publikationen von Brody und Kern (Biotechniques 36(2): 214-6) sowie Zhang et al. (Gene 487(1): 72-4), die angeblich einen verbesserten auf Natriumborat basierenden Elektrophoresepuffer vorstellen. In keinem der beiden Paper wurde die Auflösung ermittelt oder wenigstens die Leitfähigkeit des Puffers gemessen. Die vermutete Verbesserung wurde nicht quantitativ erfasst und die entscheidende Literatur nicht herangezogen.
Für mich ist es unverständlich und erschreckend, dass die längst erarbeiteten Grundlagen einer so wichtigen Methode wie der Elektrophorese so wenig bekannt sind.
In der angefügten Literatur finden Sie einige grundlegenden Arbeiten zu diesem Thema. Darüber hinaus empfehle ich das im vergangenen Jahr im Wiley-Verlag erschienene Buch von Tarso B. Ledur Kist „Open and Toroidal Electrophoresis“, das eine sehr gute Zusammenfassung der Grundlagen praktisch sämtlicher elektrophoretischer Trennmethoden enthält.
Christoph Heller
Christoph Heller entwickelte mit seiner ehemaligen Gruppe am Max-Planck-Institut für molekulare Genetik in Berlin verschiedene Techniken für die Kapillarelektrophorese von DNA. Er veröffentlichte zu diesem Thema zahlreiche Artikel sowie Beiträge für Lehrbücher.
Referenzen
Separation of double-stranded and single-stranded DNA in polymer solutions:II. Separation, peak width and resolution (Christoph Heller, Electrophoresis 20, 1978-86)
Influence of electric field strength and capillary dimensions on the separation of DNA (Christoph Heller, Electrophoresis 21, 593-602)
Principles of DNA separation with capillary electrophoresis (Christoph Heller, Electrophoresis. 22(4): 629-43) )