Tipp 214: DNA-Extraktion mit Papier
Selbstgebaute Nukleinsäure-Extraktions-Platten und -Säulchen mit Filterpapier-Einsätzen funktionieren perfekt und kosten nicht viel.
Dass Zellulosefasern Nukleinsäuren binden, ist eigentlich ein ziemlich alter Hut. Schon vor mehr als zehn Jahren sicherte sich die Firma Cortex Biochem, die mittlerweile von Promega geschluckt wurde, ein US-Patent auf eine Extraktionsmethode von Nukleinsäuren und Proteinen mit Filterpapier aus Zellulose.
Spin-Platten oder -Säulchen, die Filterpapier statt Silika als Bindematerial für DNA nutzen, kann man aber auch sehr einfach selbst herstellen. Das hierzu nötige Filterpapier liegt in jedem Labor herum und lässt sich schnell in gewünschte Größen oder Formen schneiden. Wie man damit DNA-Extraktions-Platten für den Hochdurchsatz basteln kann, erklärt der Tomatenzüchter Dilip Panthee vom Mountain Horticultural Crops Research & Extension Center in North Carolina, USA, in Planta (246: 579-84).
Für die Zellulose-Spin-Platte benötigt man eine 96-Well-Platte, deren Wells ungefähr 500 Mikroliter fassen. In die Well-Böden bohrt man jeweils ein Loch mit einem Durchmesser von etwa einem Millimeter. Anschließend stanzt man mit einem Büro-Locher Scheiben mit circa acht Millimeter Durchmesser aus Whatman Grade-3-Filterpapier aus und legt sie auf die Böden. Eine Platte auf diese Weise zu präparieren, dauert kaum zehn Minuten – und sie kostet nur den Bruchteil einer kommerziellen DNA-Extraktions-Platte.
Universelle Methode
Panthee und sein Mitarbeiter Rui Shi verwendeten für ihre DNA-Extraktionsversuche mit den Platten Tomaten – aber auch viele andere Pflanzen, wie zum Beispiel Hopfen, Salat und Paprika. Die Pflanzenproben zerdepperten sie zunächst mit dem Kugelmühlenverfahren und lysierten die Zellen anschließend mit einem CTAB-Puffer. Danach platzierten die beiden die mit dem Filterpapier präparierten Spin-Platten auf einer ungelochten 96-Well-Mikroplatte, die als Sammelplatte diente, und füllten 125 µl des Lysat-Überstandes in die Wells der Spin-Platte. Sie zentrifugierten die Platten kurz ab, verwarfen den Durchfluss und wuschen die Ansätze nach dem gleichen Prinzip zweimal mit 150 µl Ethanol. Um das Ethanol gründlich zu entfernen, zentrifugierten Panthee und Shi die Filterpapier-Platte anschließend für 15 Minuten und trockneten sie zusätzliche zehn Minuten im Abzug. Zum Schluss setzten sie die Spin-Platte auf eine neue Sammelplatte, gaben 100 µl TE-Puffer in die Wells, inkubierten fünf Minuten bei Raumtemperatur und zentrifugierten die Platten danach eine Minute ab, um die DNA von dem Filterpapier zu eluieren.
Im Schnitt fanden die Zwei in den Wells der Sammelplatte fünf bis zehn Mikrogramm DNA, wenn sie 25 Milligramm Tomatenblätter als Ausgangsmaterial einsetzten. Die DNA konnten sie direkt als Template für eine PCR verwenden.
Die Filterpapier-Platten eignen sich für die DNA-Extraktion im Hochdurchsatz. Viele Gruppen arbeiten jedoch mit kleineren Ansätzen und verwenden die üblichen Silika-Säulchen. Auch hierfür haben sich Panthee und Co. eine Filterpapier-Version ausgedacht, die sie auf bioRxiv zur Diskussion stellen (doi: http://dx.doi.org/10.1101/392696).
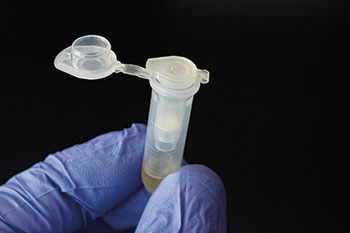
Die Säulchen kann man auf zwei Arten herstellen: Aus recycelten Silika-Säulchen oder selbstgebastelt aus PCR-Tubes und Pipettenspitzen. Die erste Variante ist ziemlich simpel. Die ausgedienten Silika-Säulchen werden zunächst zehn Minuten mit einer zehnprozentigen Natriumhypochlorit-Lösung (Bleiche) behandelt, mehrfach mit sterilisiertem Wasser gespült und danach getrocknet. Als Filterpapier dient Whatman-Grade-3-Papier, das man wie bei den Filter-Spin-Platten mit einem Papier-Locher ausstanzt.
Zwei Varianten
Ein oder zwei Filterpapier-Scheibchen setzt man in das Säulchen ein und schiebt sie mit einer Pipettenspitze auf den Boden der Silika-Membran. Nachdem man das Säulchen in das Sammel-Tube gesteckt hat, kann man das zusammengebaute Filterpapier-Säulchen autoklavieren und danach an der Luft trocknen.
Auch der Eigenbau der Filterpapier-Säulchen ist kein Hexenwerk. Dazu schneidet man den Boden von 0,5-µl-PCR-Tubes ab und setzt ein kurzes Stück aus dem oberen Teil einer 10 µl Pipettenspitze ein. Dieses dient als Halterung für die ausgestanzte Filterpapier-Scheibe, die man in das PCR-Tube einsetzt. Der Durchmesser des Papier-Scheibchens sollte etwas mehr als sechs Millimeter betragen. Für die DNA-Extraktion kann man die mit Silika-Säulchen mitgelieferten Lösungen und Puffer verwenden oder selbst zusammengestellte. Das Protokoll entspricht dem üblichen Ablauf für die DNA-Extraktion mit Spin-Säulchen.