Tipp 176: Cellfinder - Alles aus einer Hand
Sämtliche Daten zu Säugerzellen vereint in einer umfassenden Datenbank. Das wär doch was.
Wer Informationen zu einem Zelltyp sucht oder einfach nur wissen will, aus wie vielen verschiedenen Zelltypen der menschliche Körper besteht, muss sich meist durch einen riesigen Papierberg kämpfen oder die Daten aus vielen einzelnen Spezialdatenbanken zusammen klauben. Trotz des riesigen Ausstoßes immer neuer Daten zu Zellen, Zelllinien oder Geweben existieren bisher kaum Datenbanken, die das Datenmaterial aus den zahlreichen Einzelquellen auf einer Plattform vereinen.
Eine Gruppe um Andreas Kurtz vom Berlin-Brandenburg-Zentrum für Regenerative Medizin der Charité sowie Ulf Leser vom Institut für Computerwissenschaften an der Berliner Humbold-Universität, hat sich auf die Fahne geschrieben, dies zu ändern. Mitte letzten Jahres präsentierte sie die umfassende Zelldatenbank CellFinder, die Lebenswissenschaftlern und Medizinern den Zugang zu Zelldaten erleichtern soll. Die Berliner haben sich nicht weniger vorgenommen als alle relevanten Datenquellen in ihrem Zell-Repositorium zusammenzuführen. Biowissenschaftler können über das frei zugängliche Webportal www.cellfinder.de auf die in CellFinder gesammelten Daten zugreifen (Stachelscheid et al., NAR, 2014, Vol 42).
Neues Ontologie-Konzept
Für Bioinformatiker dürfte insbesondere das neue Ontologie-Konzept der Berliner Gruppe interessant sein, mit dem diese die Daten aus bestehenden Datenbanken sowie auch Neueinträge organisiert. In der Bioinformatik legen Ontologien fest, welche Vorraussetzungen die Mitglieder einer Gruppe, oder im Informatiker-Sprech einer Klasse, erfüllen müssen, um zu dieser zu gehören. Über Relationen oder Objekteigenschaften werden die einzelnen Klassen in Datenbanken schließlich miteinander verknüpft.
Für CellFinder entwickelte die Mannschaft um Kurtz und Leser die Ontologie CELDA (Cell: Expression, Localization, Development, Anatomy), die zum Beispiel bestimmt, wie Zellen in Zelltypen eingeteilt werden oder welche Vorraussetzungen für die Zugehörigkeit zu einzelnen Zelllinien gelten. Darüber hinaus integriert CELDA bereits bestehende Ontologien, die beispielsweise Entwicklungsstadien, die Genexpression in Zellen oder histologische und zytologische Daten von Zellen erfassen.
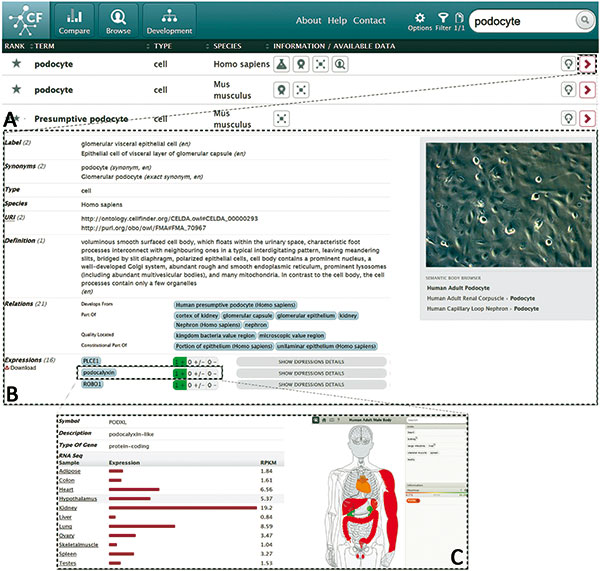
Nicht immer reichen Ontologien jedoch aus, um an Daten heranzukommen. In diesen Fällen führt auch für die Berliner Gruppe kein Weg daran vorbei, sie per Hand und Auge aus den unterschiedlichen Quellen herauszusuchen und in CellFinder einzupflegen. Gegenwärtig enthält die Datenbank Beschreibungen zu 2.294 Säugerzelltypen und 50.951 Zelllinien. Ergänzt werden diese durch mehr als 3.000 mikroskopische und anatomische Bilder sowie zahllose Expressionsprofile.
Der Nutzer kann über eine textbasierte Suchfunktion der CellFinder-Webapplikation nach Zellen suchen oder mit dem Semantic Body Browser beziehungsweise dem Developmental Tree der Applikation durch die Datenbank surfen. Im Semantic Body Browser sind verschiedene Ansichten von Mensch und Maus enthalten, die in weitere Ebenen unterteilt sind, die zusätzliche Details zeigen. Klickt man zum Beispiel auf die Lunge des Menschen, so öffnet sich eine Übersichtsseite, die neben generellen Informationen zur Lunge auch Daten zu den in der Lunge exprimierten Genen liefert. Die Browserfunktion des Developmental Tree stellt die Zellen und deren Entwicklungsstadien schließlich in einer Baumstruktur dar, mit der man den Differenzierungsgrad und das weitere Schicksal ausgesuchter Zellen analysieren kann.
Navigieren mit Körperbrowser
Übrigens, falls Sie tatsächlich wissen wollen, wie viele unterschiedliche Zelltypen im Menschen vorkommen: die CellFinder Datenbank führt 1.058 auf. Das sind wesentlich mehr als die etwa 200, die man meist in der Literatur findet. Es sind aber gerade mal halb so viel, wie von der Zelldatenbank CELLPEDIA vorgeschlagen (Hatano et al., Database, 2011, bar046). Die Arbeit dürfte den Entwicklern von Zelltypen-Ontologien also so schnell nicht ausgehen.
Harald Zähringer