Selenoproteine und Selenosom
von Juliet Merz (Laborjournal-Ausgabe 10, 2022)
(07.10.2022) Selen ist ein essenzieller Mikronährstoff für Mensch und Tier. Im Körper wird das Element vor allem in Proteine verbaut, die sogenannten Selenoproteine. Sie übernehmen viele unterschiedliche Funktionen, zum Beispiel katalysieren sie als Oxidoreduktasen chemische Reaktionen oder treten als Schilddrüsenhormone auf den Plan.
Die Entdeckung der Selenoproteine ist aber noch gar nicht so lange her. Nachdem Forschungsgruppen in den 1950er-Jahren Selen als Element mit einer biologischen Funktion sowohl in Bakterien als auch in Eukaryoten identifiziert hatten, wollten es Biologen, Chemiker und Mediziner genauer wissen: Wo taucht Selen in der Zelle beziehungsweise dem Organismus auf?
1973 landete ein Forscher-Trio vom Institut für Physiologische Chemie der Uni Tübingen den ersten gesicherten Treffer: Es hatte die Glutathion-Peroxidase aus Rinderblut mittels einer Neutronenaktivierungsanalyse untersucht und dadurch die Elementzusammensetzung quantitativ gemessen. Und tatsächlich: Das Enzym enthielt Selen (FEBS Letts 32: 132-4).
Auf welche Art und Weise das Element in Enzymen und anderen Proteinen jedoch verbaut ist, entschlüsselten drei Jahre später US-amerikanische Biochemiker (PNAS 73 (8): 2659-63). In einem bakteriellen Enzym fanden sie Selen in Form einer neuartigen Aminosäure – dem Selenocystein. Selenocystein ähnelt (wie der Name schon vermuten lässt) dem Cystein, bloß dass es anstelle eines Schwefelatoms ein Selen trägt. Über die Jahre hinweg entdeckten Forscher die außergewöhnliche Aminosäure dann auch in weiteren Enzymen sowohl von Prokaryoten als auch von Eukaryoten.
Allerdings warfen die Funde immer dieselbe Frage auf: Wie baut die Zelle Selenocystein überhaupt in Proteine ein? Denn klar war auch: Jedes Basentriplett ist bereits einer der klassischen zwanzig Aminosäuren zugeordnet oder beschreibt ein Start- beziehungsweise Stopp-Codon – für Selenocystein blieb also theoretisch gar kein Basentriplett übrig.
Den ersten Hinweis auf des Rätsels Lösung fanden 1986 parallel zwei Forschungsgruppen vom Beatson Institute for Cancer Research, UK, und der Universität München (EMBO J. 5: 1221-7; PNAS 83: 4560-4). Die beiden Teams hatten sich die Gensequenzen zweier Enzyme genauer angeschaut – einmal diejenige der bereits erwähnten Glutathion-Peroxidase der Maus wie auch diejenige der Formiat-Dehydrogenase H aus E. coli. Dabei waren sie auf eine verblüffende Eigenheit gestoßen: Inmitten der codierenden Gensequenzen fanden sie ein TGA(UGA)-Codon – also eigentlich das Signal für die sofortige Beendigung der Translation. Die UK-Gruppe zog schließlich den richtigen Schluss aus ihren Beobachtungen: UGA steuert den Einbau von Selenocystein in Proteine.
Rätsel gelöst
Aber warum beendet das Ribosom beim Stopp-Codon nicht die Translation, sondern baut stattdessen Selenocystein ein? Normalerweise läuft die Termination bei der Translation vereinfacht wie folgt ab: Wenn das Ribsosom über die mRNA gleitet und an ein Stopp-Codon gelangt (UGA, UAA oder UAG), pausiert es kurz, wodurch ein Release-Factor-Protein (RF) im Ribosom binden und die Translation beenden kann. Befindet sich ein UGA-Codon allerdings inmitten einer codierenden Sequenz, tritt eine Handvoll unterschiedlicher molekularer Mitspieler auf den Plan, die das Stopp-Codon in ein Selenocystein(Sec)-Codon umprogrammieren – das sogenannte Stop-to-Sec Recoding.
Welche Moleküle dabei mitmischen, ist bereits bekannt. Bislang war es aber ein Rätsel, wie die Beteiligten genau funktionieren und zusammenwirken. Ein Team der Berliner Charité und der University of Illinois in Chicago konnte jetzt mehr Licht ins Dunkel bringen (Science 376: 1338-43). Mittels Kryo-Elektronenmikroskopie beobachteten Erstautor Tarek Hilal et al. den ribosomalen Komplex, der sich um das UGA-Codon bildet – das Selenosom.
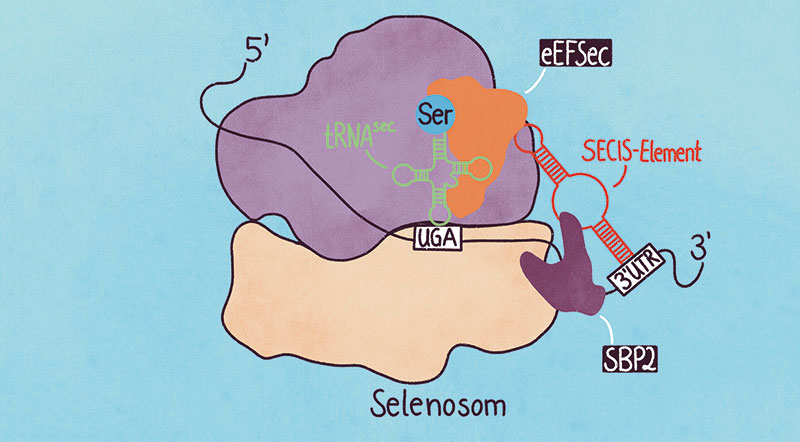
Beim Einbau von Selenocystein ist eine spezielle tRNA vonnöten, die mit der 21. Aminosäure beladen ist (tRNASec). Zusätzlich braucht das Ribosom noch einen Translationsfaktor: SelB in Prokaryoten, eEFSec in Eukaryoten. Bei Letzteren kommt noch ein Proteinfaktor dazu, das sogenannte SECIS-binding Protein 2 (SBP2).
Ein weiterer ganz entscheidener Mitspieler ist die Selenocystein-Insertionssequenz, das sogenannte SECIS-Element. Interessanterweise unterscheidet sich die Lage des Genabschnittes in Prokaryoten und Eukaryoten massiv. Während sich in Ersteren das SECIS-Element direkt an UGA anschließt, ist es bei Eukaryoten in der 3‘-untranslatierten Region der mRNA zu finden, also viele hundert Nukleotide entfernt.
Doch die Abgeschiedenheit des SECIS-Elements scheint bei Eukaryoten kein Problem zu sein – ganz im Gegenteil. Hilal und Co. konnten zeigen, dass die mRNA eine große Schleife bildet, sodass das UGA-Stopp-Codon und das SECIS-Element gleichzeitig am Ribosom gebunden sind (siehe Abbildung). „Das SECIS-Element wird dabei in einer bislang unbekannten Bindetasche am Ribosom verankert“, erklärt Seniorautor Christian Spahn in einer Pressemitteilung der Charité. Der Translationsfaktor eEFSec und der Proteinfaktor SBP2 interagieren derweil nicht direkt miteinander, sondern haben das SECIS-Element an gegenüberliegenden Seiten gebunden. Gleichzeitig interagiert eEFSec mit der tRNASec und stabilisiert den Selenosom-Komplex damit weiter.
Was anschließend bei der Translation von Selenoproteinen abläuft, bleibt allerdings noch ein Rätsel.