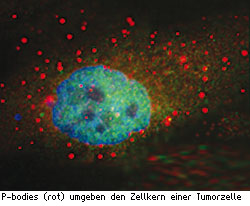
P-Bodies
von Petra Stöcker (Laborjournal-Ausgabe 01, 2006)
Das kurze Leben von mRNAs beginnt bei Polymerase II und endet nach Translation an den Polysomen - so dachte man lange Zeit. US-Forscher wiesen nun jedoch nach, dass sogenannte "processing bodies" (P-bodies) mRNAs auch zur späteren Translation parken können.
Das Leben der messenger-RNA (mRNA) ist im Normalfall kurz und anstrengend. Als Spross der eukaryotischen Polymerase II überbringt sie eifrig Arbeitsvorschriften von der DNA-Matrize zur Protein-produzierenden Maschinerie.
Lange waren Wissenschaftler sich einig, dass die mRNA mit dem Ende des Botengangs ihre Pflicht erledigt hat und im Zytoplasma umgehend klein gehackt wird. Seit kurzem kennt man den Schauplatz des Verdaus genauer: kleine, zelluläre Partikel mit dem unspektakulären Namen "P-bodies/GW-bodies".
Deren Entdeckung war eher ein Nebenprodukt bei der Untersuchung des mRNA-Abbaus, an der seit circa zehn Jahren mehrere Gruppen arbeiten. Darunter auch Roy Parkers Team an der University of Arizona in Tucson. Anhand der Bäckerhefe entwirrten sie die Reaktionskette, bei der zunächst der Poly-Adenosinmonophosphat-Rest am 3'-Ende gestutzt wird. Dem folgt die Abtrennung der methylierten 5'-terminalen Nukleotid-Kappe (Cap). Sind beide Enden angeknabbert, wird das lange Molekül schrittweise von vorne (5') nach hinten (3') fein säuberlich in seine Einzelteile zerlegt.
Die meisten daran beteiligten Enzyme konnte Parkers Labor identifizieren und charakterisieren. Seine Doktorandin Ujwal Sheth bearbeitete Ende der 90er Jahre hauptsächlich das Enzym Dhh1p, eine ATP-abhängige RNA-Helikase und Aktivator der 5'-Decapping-Reaktion. Bei ihren Klonierungs- und Positionierungsversuchen mit Hilfe von GFP (green fluorescent protein) blitzte ihr das untersuchte Enzym im Fluoreszenz-Mikroskop nur punktuell in bestimmten Bereichen entgegen. Auch alle weiteren Abbau-Enzyme, und schließlich eine synthetische mRNA strahlten als diskrete grüne Spots an den gleichen Stellen. Sheth taufte die entdeckten Partikel "processing bodies"("P-bodies", PBs) (Science 300, S. 805).
Auch Orte für RNA-Interferenz
Zahlreiche Experimente später drängte sich der Verdacht auf, dass die RNA-Moleküle innerhalb der P-bodies nicht zwangsläufig ihr drohendes Ende finden müssen - jedenfalls nicht sofort. Einige der in Hefe-P-bodies charakterisierten Enzyme finden sich in anderen Organismen in Granula, welche RNA zur späteren Verwendung aufbewahren. In der Tat kann mRNA aus den PBs auch wieder unversehrt auftauchen und problemlos für die Translation bereitstehen (Science 310, S. 486), was ihnen in Parkers Team augenzwinkernd die inoffizielle Bezeichnung "purgatory bodies" einbrachte.
Die ehrgeizigen Arbeiten rund um die Partikel konnten auch bei der noch relativ jungen und damit noch teilweise geheimnisumwobenen Disziplin der RNA-Interferenz (RNAi) etwas Klarheit schaffen. Kurze, doppelsträngige RNA-Moleküle (dsRNAs) sorgen hier für eine Blockade der Translation über den mRNA-Abbau durch Nukleasen, der demnach in P-bodies stattfindet. Den dafür entscheidenden Hinweis lieferte die Entdeckung, dass sich die Proteine Argonaut-1 und -2, Schlüsselkomponenten der Interferenz, bevorzugt in P-bodies konzentrieren.
P wie Parkplatz?
Die Fülle an Ergebnissen der letzten zwei Jahre aus unterschiedlichen Forschungsgruppen zeigt, dass P-bodies jedoch weit mehr als nur Häcksel-Station für mRNA und daran beteiligte Proteine vergleichbar auch in Säugetier-Zellen zu finden sind. Das "P" könnte auch als Abkürzung für "Parkplatz" stehen.
Dieser Eigenschaft sind Paul Anderson und Nancy Kedersha vom Harvard´s Brigham and Women´s Hospital in Boston auf den Fersen. In Stresssituationen, wie etwa Virusbefall, UV-Bestrahlung oder osmotischem Ungleichgewicht fährt die Zelle ihre Proteinproduktion auf ein Minimum zurück. Daran beteiligt sind bestimmte Stress-Granula, in die RNA eingelagert und so dem Translationsvorgang kurzerhand entzogen wird. Die Wissenschaftler zeigten, dass diese Partikel sich mit P-bodies kurzschließen, mehrere Proteine brüderlich teilen und die RNA untereinander sortieren, entweder zur Umformung oder zum Verdau (J. Cell Biol. 169, S.871).
Eine mögliche direkte Kontrolle der Protein-Synthese in Hefe präsentieren Roy Parker und Jeff Coller vom Howard Hughes Medical Institute, Department of Molecular and Cellular Biology, der University of Arizona (Cell 122, S. 875). Hefezellen, denen die P-body-Proteine Dhh1p und Pat1 - beides Aktivatoren der Decapping-Reaktion - fehlen, können ihre Proteinproduktion nicht mehr drosseln, die Zahl der P-bodies sinkt rapide. Im Gegensatz dazu wurde die Translation bei Überexpression beider Proteine merklich zurückgefahren. Die Partikel nahmen auffällig an Größe zu, das Zellwachstum war erheblich eingeschränkt. Parker vermutet ein Gleichgewicht innerhalb der Zelle zwischen Translation der mRNA an Polysomen und P-body-Formation.
Zwischenzeitlich mehren sich Hinweise über eine mögliche Verbindung zwischen P-bodies und einigen Erkrankungen. Bertrand Seraphins Team fand in humanen P-bodies das Protein RCK/p54, humanes Homolog zu Dhh1p, welchem eine Beteiligung an der Entstehung bestimmter Krebsarten zugeschrieben wird. Beispielsweise war dessen Konzentration zusammen mit der Anzahl an P-bodies in Brustkrebszellen deutlich erhöht.
Ignorieren lassen sich die unscheinbaren Zellkörperchen bei all dem nicht mehr. Möglich, dass sie in der ein oder anderen noch ungeklärten Frage gar zum Hot Spot aufsteigen.
Letzte Änderungen: 24.04.2006