Bioinformatik: Das öffentliche Mikrobiom - Metagenomik in der U-Bahn
Andrea Pitzschke, Laborjournal 04/2022
(12.04.2022) Jedes Jahr ziehen am 21. Juni mit Wattestäbchen bewaffnete Freiwillige los und nehmen im öffentlichen Raum von Städten Proben für das Konsortium „Metagenomics and Metadesign of Subways and Urban Biomes“ (MetaSUB).
Am weltweiten MetaSUB-Projekt beteiligt ist auch die Wiener Bioinformatikerin Alexandra Graf.
Die Aussage „Ich mache gerne Abstriche“ zeugt mitunter nicht von defensiver Schüchternheit, sondern eher von kompromissloser Wissbegier. Abstriche sind eine wichtige Ressource für die Metagenomik, die mit ihren Methoden das komplexe Sammelsurium an bekannten und unbekannten Organismen in einer Probe ans Licht bringt. Unvoreingenommen erfasst die Metagenomik (theoretisch) sämtliche DNA-Sequenzen und spürt dabei nicht nur Organismen auf, die kultivierbar sind oder bestimmte Marker tragen, wie zum Beispiel 16S-rRNA-Gene.
DNA-Extraktion, die Erstellung einer DNA-Bibliothek und die darauffolgende Sequenzierung gehören aber nur zum Vorspann der Metagenomik-Analyse, so aufwändig diese Methoden auch sein mögen. Mit den unzähligen ein paar hundert Nukleotide langen Sequenzhäppchen, die danach vorliegen, kann man noch nicht viel anfangen. Ohne Bioinformatik bleiben die darin enthaltenen Informationen verborgen.
Erst die Algorithmen der Bioinformatiker analysieren die Sequenzen, fügen Gene oder ganze Genome zusammen, durchforsten Datenbanken, stellen systematische Vergleiche an und ergründen Verwandtschaftsbeziehungen. Je besser die Datenbanken bestückt sind, desto mehr Aussagekraft hat jeder einzelne Datensatz. Mit der Größe und Qualität von Datenbanken wächst rückwirkend auch der Informationsgehalt früherer Dateneinträge. Es ist ein bisschen wie beim „Tatort“, wenn einem umtriebigen Kriminellen anhand seiner DNA jede Tat seiner langjährigen „Karriere“ nachgewiesen werden kann.
Mikrobiom in U-Bahnen
Ganz neue Möglichkeiten eröffnen Metagenom-Daten und bioinformatische Werkzeuge, die das internationale MetaSUB-Konsortium entwickelt hat (https://metasub.org/). Anhand systematisch gesammelter metagenomischer Fingerabdrücke, die Menschen im öffentlichen Raum hinterlassen haben, will MetaSUB die Welt genetisch kartieren. Grundgedanke dabei ist, dass das Metagenom eines Probenabstrichs dessen ortsabhängige mikrobiologische Zusammensetzung widerspiegelt. Das Mikrobiom, mit dem zum Beispiel U-Bahn-Fahrer permanent in Kontakt kommen und das sie weitergeben, ist in Tokio ein anderes als in Toronto – jede Großstadt hat ihre ureigene Signatur.
Zu diesem Ergebnis kam eine internationale Studie von MetaSUB, an der auch Forscher aus Deutschland, der Schweiz und Österreich mitwirkten (Cell 13: 3376-93.e17).
Die Bioinformatikerin und Co-Autorin des Cell-Papers Alexandra Graf von der Fachhochschule Campus Wien erzählt, wie es zu MetaSUB kam: „Die Initiative wurde von Christopher E. Mason von der Weill Cornell University in New York ins Leben gerufen. Wie er uns erzählte, kam er 2015 auf die Idee, als er mit seiner kleinen Tochter in der U-Bahn fuhr, die ihre Eiscreme im Stanitzel [Anm. d. Red.: Wienerisch für Eiswaffel] und auch auf ihren Händen abschleckte. Er wollte ganz einfach wissen, mit welchen unsichtbaren U-Bahn-Gästen sie denn da Kontakt hatte. Danach gab es bald die erste Pilotstudie in New York. 2016 wurden auch andere Städte eingeladen, bei dem Projekt mitzumachen.“
Über drei Jahre hinweg haben MetaSUB-Forscher weltweit am Swabbing Day (21. Juni) im öffentlichen Raum von sechzig Großstädten an Geländern, Ticketschaltern und Sitzbänken et cetera mit Wattestäbchen insgesamt 4.728 Proben nach einem vorab ausgearbeiteten, streng definierten Prozedere abgestrichen, aufbereitet und sequenziert.
Mit einer ganzen Choreographie bioinformatischer Algorithmen und Statistikanalysen versuchten sie danach, sinnvolle Informationen aus den gesammelten NGS-Daten zu extrahieren. Dazu wurden die circa 500 Nukleotide langen Sequenzen zunächst am Computer in 31nt-Fragmente zerstückelt (k-mere) und Gendatenbanken nach k-mer-Homologen abgesucht. Die MetaSUB-Crew nutzte hierfür die frei zugängliche Software KrakenUniq (github.com/fbreitwieser/krakenuniq).
Core- und Subcore-Mikrobiom
Mit der sogenannten MetaSUB Core Analysis Pipeline (CAP) nahm das Team die Abstrich-Sequenzen anschließend unter die Lupe. Damit sich die Entwicklungsarbeit an der Analyse-Pipeline für die gesamte Forschungsgemeinde auszahlt und niemand das Rad ein zweites Mal erfinden muss, steht sie detailliert und frei zugänglich auf GitHub zum Abruf bereit (github.com/MetaSUB/MetaSUB_CAP).
Die Ergebnisse der Analyse zeigen, dass ein gewisses Set an Mikroorganismen in allen Städten kursiert. In diesem Kern-Mikrobiom aus 31 Taxa geben Proteobacteria, Actinobacteria und Firmicutes den Ton an. Auch Akne-Erreger (Cutibacterium acnes) lauern überall. Neben dem Core-Mikrobiom, das in mehr als 97 Prozent aller Proben anzutreffen ist, existiert in 70 bis 97 Prozent der Proben ein Subcore-Mikrobiom mit 1.145 Taxa, das Saccharomyces cerevisae als einzigen Hefevertreter enthält. Vielleicht von bierbekleckerten oder mehlverstaubten Händen? Gut möglich. „Wir sehen viele Nahrungsmittel-assoziierte Bakterien“, erklärt Graf. Knapp 2.500 Taxa sind vergleichsweise selten und finden sich in weniger als einem Viertel der Proben. Das insgesamt aufgedeckte „urbane Panmikrobiom“ besteht aus 4.424 Taxa. Sein Muster weicht grundsätzlich ab vom Mikrobiom, das in menschlichen Exkrementen und Innereien logiert (Nature 486: 207-14).
Unter den Sequenzen gab es aber auch viele, die in Referenzdatenbanken bisher nicht vorkommen, interessanterweise besonders viele in Proben aus Europa. „Ich vermute darin einen Bias in den Datenbanken“, erklärt Graf – und führt weiter aus: „Historisch gibt es eher wenige Umweltmikrobiom-Projekte. Wenn, dann stammen sie meist aus sehr speziellen Umgebungen, etwa Minenabflüssen, heißen Quellen und anderen extremen Lebensräumen – zum Beispiel aus dem Meer, wie bei den Metagenom-Entdeckungsreisen von Craig Venter, oder aus Boden- und Wurzelproben der landwirtschaftlichen Produktion. Auch das humane Mikrobiom ist natürlich stark erforscht. Es kommt also darauf an, wo unser Forschungsfokus liegt, und der scheint nicht auf alltäglichen Umgebungen in Europa gelegen zu haben. Einen ähnlichen Effekt sieht man bei Genomen von höheren Tieren und Pflanzen, auch hier sind die Tiere und Pflanzen, die in Europa heimisch sind, schwächer vertreten.“
Biodiversitätsgradient
Die Anfangsinvestition in die Sammlung, Aufbereitung und Sequenzierung der Proben wirft auch im Nachhinein noch eine Wissensdividende ab. Und da jede neue, statistisch abgesicherte Beobachtung am Bildschirm eine plausible physische beziehungsweise biologische Erklärung verlangt, beflügeln sich Bioinformatiker und Biologen gegenseitig. In der MetaSUB-Studie zeigte sich, dass die Abstriche mit wachsender Entfernung vom Äquator weniger Taxa enthalten und in der nigerianischen Stadt Offa die größte Vielfalt herrscht. Warum ist das so? „Dieses Phänomen kennt man auch von den höheren Tieren und Pflanzen als Breitengradient der Diversität oder latitudinalen Biodiversitätsgradienten. Die Ursachen sind aber auch dort noch nicht klar. In den MetaSUB-Untersuchungen ist der Trend nur sehr leicht und die geographische Auflösung schlecht, aber es könnte sein, dass es ein ähnliches Phänomen wie im Makrobereich tatsächlich gibt.“
Die Mikrobiom-Signatur in öffentlichen Verkehrsmitteln, etwa in Offa, blieb über lange Zeiträume charakteristisch. Wurden vom selben Standort nach Jahren erneut Proben entnommen, ähnelten sich ihre Metagenome mehr als die Metagenome von verschiedenen Orten. Die variierende Ausstattung von U-Bahnen, etwa die Oberflächenmaterialien der Geländer, Griffe et cetera wären eine mögliche Erklärung. Oder liegt es vielleicht an der unterschiedlichen Bevölkerungsdichte? „Ja, im Moment scheint das einer der großen Einflussfaktoren zu sein“, meint Graf. „In Hongkong hat man auch gesehen, dass es einen Tagesrhythmus gibt. Während des Tages taucht viel humanes Mikrobiom auf, nachts verschwindet dieses wieder und ein anderes Profil kommt zum Vorschein.“
Urbanes Panresistom
Welche örtlichen oder zeitlichen Umstände das Mikrobiom beeinflussen, klärte die MetaSUB-Gemeinde mit einer Principle-Component-Analyse (PCA). Diese brachte ein eindeutiges Ergebnis: Der entscheidende Faktor ist das Klima. Von diesem hängt auch ab, welche Antibiotikaresistenzen in öffentlichen Verkehrsmitteln unterwegs sind. Das urbane Panresistom enthält ein beeindruckendes Spektrum von Resistenzen, die Antibiotika mit unterschiedlichen Wirkmechanismen lahmlegen oder umschiffen können.
Mithilfe der MegaRES-Ontology-and-Alignment-Software fanden die Forscher heraus, dass die meisten Proben Sequenzen von Resistenzgenen enthielten, oft auch mehrere (Nucleic Acids Res. 45: 574-80). Dem Analysetool liegen Sequenzen von achttausend Antibiotikaresistenz-Genen zugrunde, es wurde insbesondere für die effiziente Annotation von großen Sequenz-Datensätzen konzipiert.
Nur etwa ein Viertel der Abstriche enthielt keinen Hinweis auf Resistenzen. Der Anteil unbedenklicher Standorte könnte aber noch schrumpfen, wenn neue Resistenzgen-Sequenzen erkannt und in die Datenbank aufgenommen werden. Spiegelt das Resistom eines Standorts vielleicht den dortigen Umgang mit Antibiotika wider, und gibt es Hinweise, dass insbesondere Resistenzgene gegen Antibiotika kursieren, die in einem Land vornehmlich zum Einsatz kommen? „Ja, man sieht diesen Trend“, bestätigt Graf. „Wobei wir weniger Resistenzen gefunden haben als erwartet, was aber auch an der Sequenziertiefe liegen kann. Ein Resistenzgen mit der entsprechenden Mutation heißt auch noch nicht, dass der Organismus tatsächlich eine Resistenz aufweist – das muss im Labor noch ausgetestet werden. Oft schützen Gene, die Antibiotikaresistenzen erzeugen können, Mikroorganismen auch vor anderen Umweltstressoren, zum Beispiel Schwermetallen.“
In Bus oder Bahn die Finger abzulecken oder sich in den Augen zu reiben, scheint auch aus anderen Gründen keine gute Idee zu sein. Wer alle 60 Städte abfährt, die an der MetaSUB-Studie beteiligt waren, kommt mit fast 12.000 Virusspezies in Kontakt. Nur DNA-Viren wohlgemerkt, denn RNA-Viren gingen bei den MetaSUB-Protokollen als Schwarzfahrer durch. Für die Analyse der Virus-Sequenzen nutzte das Konsortium die Uncultivated-viral genome-mapping-Plattform des Joint Genome Institutes in Berkeley (Nucleic Acids Res. 47: 678-86). Den Schrecken der Virusbedrohung kann Alexandra Graf jedoch relativieren: „Viele der DNA-Viren, die wir gefunden haben, sind Bakteriophagen – also eigentlich Helfer gegen Bakterien.“
Die computerbasierte Metagenomik hat Suchtpotenzial. Wer kreativ und bioinformatisch angehaucht ist, kann aus den Datensätzen vermutlich noch so manches Muster ableiten. Und wie ist Alexandra Graf zur Metagenomik gekommen? „Mein Hintergrund ist vor allem die Biologie, auch ökologische Zusammenhänge haben mich schon immer interessiert. Nach dem Biologiestudium hat es mich dann aber in die IT verschlagen, die Verbindung dieser beiden Interessen im Rahmen der Bioinformatik schien ideal zu sein. Dass ich nun mit Mikroorganismen arbeite, hat sich durch den biotechnologischen Konnex ergeben. Aber ich muss sagen, dass ich sowohl die bioinformatische Arbeit wie auch das Metagenom-Thema absolut spannend finde. Es ist eine Art Detektivarbeit und ich bin immer wieder fasziniert, wie stark wir und unsere Welt von diesen kleinsten Lebewesen (und Viren) beeinflusst werden.“
Wie wirken sich Corona-Hygienemaßnahmen aus?
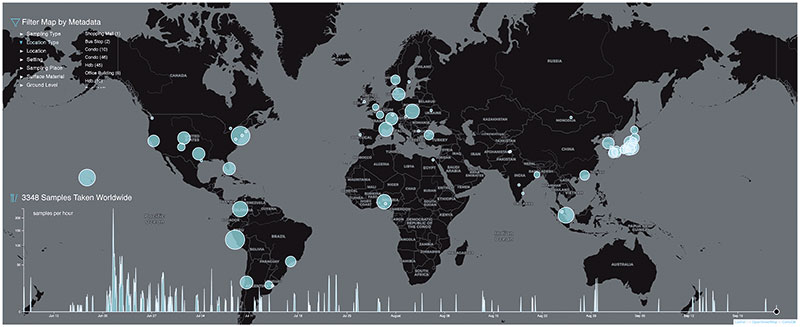
Mit dem Cell-Paper ist das MetaSUB-Konsortium ins Rampenlicht gerückt, einfach nur sonnen will man sich darin aber nicht. Ganz im Gegenteil, es wird weiter gesammelt und analysiert. Auf der interaktiven Plattform des Projekts, die Aufschluss zu Art und Oberflächenmaterial der Probe sowie Datum des Abstrichs gibt, sind derzeit fast 25.000 Proben verzeichnet. Auch 2020 und 2021 nahmen die MetaSUB-Forscher Proben. Da stellt sich natürlich die Frage, ob sich die Corona-Hygienemaßnahmen auch auf das Mikrobiom in Bus und Bahn ausgewirkt haben. „Das wissen wir leider noch nicht, die Frage ist aber sehr spannend“, so Graf. Durch die Pandemie wird sich die Antwort aber noch etwas in die Länge ziehen. „Die Sequenzierung läuft zentral in New York. Dort waren aber alle Kapazitäten durch das Infektionsmonitoring ausgelastet.“