Auf dem langen Weg zur Demokratisierung
Tiefgekühlte Elektronenmikroskopie
von Karin Hollricher, Laborjournal 11/2020
(11.11.2020) Weil die gelösten Strukturen zu verschwommen waren, stand die Kryo-Elektronenmikroskopie lange Zeit im Schatten der Röntgenstrukturanalyse. Das ist inzwischen passé. Neue Elektronendetektoren und verbesserte Software liefern so hochaufgelöste Bilder, dass immer mehr Strukturbiologen auf die Kryo-EM umsteigen.
Im Januar 2016 wählte Nature die Kryo-Elektronenmikroskopie (Kryo-EM) zur „Methode des Jahres“ – obwohl die Technik schon lange bekannt war. Die wesentlichen Grundlagen dafür wurden in den Siebzigerjahren gelegt. Jacques Dubochet entwickelte während seiner Zeit als Gruppenleiter am EMBL in Heidelberg die Vitrifizierung, mit der sich Proben so schnell einfrieren lassen, dass sich keine schädigenden Eiskristalle bilden. Joachim Frank schrieb Software am New York State Department of Health in Albany, New York, um aus vielen EM-Bildern dreidimensionale Strukturen zu rekonstruieren. Und Richard Henderson vom MRC Laboratory of Molecular Biology in Cambridge publizierte 1975 die erste Proteinstruktur aus dem gekühlten Transmissions-Elektronenmikroskop (TEM), damals mit einer Auflösung von acht Ångström. Eine beinahe atomare Auflösung gelang ihm 1990.
Vor drei Jahren wurde den drei Wissenschaftlern dafür der Chemie-Nobelpreis verliehen. Der Grund, die Technik 2017 so besonders zu würdigen, war der gewaltige Fortschritt in der Auflösung der Bilder. Diese „Resolution Revolution“ (Nature 343: 1443) war in erster Linie der Entwicklung neuer Detektoren zu verdanken, die schneller sind und rauschärmere Bilder liefern als die bis dahin verwendeten Kameras.
Strukturbiologen verwenden die Elektronenmikroskopie bei sehr tiefen Temperaturen (mindestens minus 150 Grad Celsius) auf zweierlei Weise: Die Kryo-Elektronenmikroskopie setzen sie zur hochauflösenden Strukturanalyse einzelner Moleküle ein. Schauen sie damit aber in eine Zelle hinein und bilden größere Komplexe oder Organellen im zellulären Kontext in situ ab, sprechen sie von Kryo-Elektronentomographie (Kryo-ET).
Verschiedene Blickwinkel
Da Elektronenmikroskope grundsätzlich nur zweidimensionale Projektionen liefern, rekonstruiert man die dreidimensionalen Strukturen mit Bildern aus verschiedenen Blickwinkeln. Bei der Kryo-ET kippt man dafür den gesamten Probenhalter und nimmt rund 60 Bilder aus verschiedenen Blickwinkeln auf. Die Kryo-EM kommt dagegen mit einem Bild aus, denn auf dem Probenhalter werden Moleküle ja in allen möglichen Orientierungen abgelagert und festgefroren – zumindest in der Theorie. Praktisch verhalten sich manche Proben anders, und immer macht einem der schlechte Kontrast einen Strich durch die „Ein-Bild“-Rechnung.
Kontrast ist ein essentielles Thema bei der Arbeit mit einem TEM. Egal, ob man sich für einzelne Moleküle oder eine 30 Mikrometer große Zelle interessiert: Das Bild aus dem TEM resultiert grundsätzlich aus der Differenz der Elektronendichte in den Proben und dem sie umgebenden Eis. Leider ist diese Differenz nicht besonders groß, die Bilder sind also sehr verrauscht. Deshalb kämpft der Strukturbiologe mit vielen Mitteln um ein möglichst hohes Signal-Rausch-Verhältnis. Die Option, einfach den Elektronenstrahl und somit das Signal zu verstärken, fällt flach, denn die Elektronen würden jede biologische Probe zerstören. Also gilt es, das Rauschen zu minimieren, was sowohl auf Seiten des Geräts mit Detektoren und Linsenkorrektoren, wie auch auf Seiten der Bildverarbeitung geschieht.
„Tatsächlich mitteln wir viele sehr verrauschte Bilder, bis ein Motiv scharf erscheint“, sagt Bettina Böttcher, Spezialistin für Kryo-EM am Lehrstuhl für Biochemie der Universität Würzburg. „Betreibt man aber die Bearbeitung zu intensiv oder auf der Basis falscher Referenzen, erhält man falsche Bilder – was man in der Szene als ‚Einstein-von-Rauschen‘-Effekt bezeichnet. Deshalb wurden statistische Faktoren in die Software eingeführt, die die gefundene Lösung mit ihrer Wahrscheinlichkeit, korrekt zu sein, gewichten. In der Realität ist das ein ziemlich komplizierter Prozess.“
Je weniger Bilder man mitteln muss, um das Signal vom Rauschen zu unterscheiden, desto geringer die Gefahr, falsche Bilder zu generieren. Am besten arbeitet man von Beginn an mit möglichst kontrastreichen Bildern. Hier kommen die neuen Sensoren ins Spiel. Sie detektieren die Elektronen direkt und produzieren weniger Rauschen als die zuvor verwendeten CCD- und CMOS-Kameras. In Letzteren werden die Elektronen von Szintillatoren in Photonen und danach wieder in Photoelektronen verwandelt. Direct Electron-Detektoren (DED), auch Direct Detection Device (DDD) genannt, sind nichts anderes als CMOS-Chips, die auch in den Kameras von Mobiltelefonen verbaut werden. Um sie im EM verwenden zu können, müssen sie jedoch größere Pixel haben und gegen die Elektronenstrahlen „abgehärtet“ werden. Der Markt wird von den Firmen GATAN, FEI Thermo Fisher und Direct Electron beherrscht, andere Hersteller gibt es aktuell nicht.
Der DED zählt jedes einzelne Elektron und generiert hierdurch nicht nur rauschärmere Bilder. Mit bis zu 1.500 Frames pro Sekunde ist er auch viel schneller als ein CCD. „In der Geschwindigkeit liegt die Magie der neuen Detektoren“, sagt Böttcher. „Nur so können einzelne Elektronen getrennt von anderen detektiert und die Gesamtbelichtungszeiten im Bereich einiger Sekunden gehalten werden.“
Auch lässt sich mit einem DED die zunehmende Schädigung der Probe von Bild zu Bild verfolgen. Die Schäden verursachen Rauschen, wodurch sich die Auflösung verschlechtert. Böttcher: „Wir können dieses Rauschen reduzieren, indem wir entsprechend der Schädigung die einzelnen Bilder bei der Rekonstruktion des Moleküls unterschiedlich gewichten. Das heißt, wir lassen die ersten Frames mehr zu der Struktur beitragen als die letzten. Das funktioniert schon mit unserem älteren, vergleichsweise langsamen Detektor, der nur 40 Frames pro Sekunde schafft.“
Weniger Rauschen
Einen Beitrag zum Bildrauschen liefern auch intakte Moleküle. Sie bewegen sich nämlich während der Messung, und zwar besonders stark beim ersten Kontakt mit dem Elektronenstrahl. Das lässt sich nachträglich minimieren, indem man die jeweilige Position der Moleküle in den ersten Frames bestimmt und entsprechend korrigiert.
Routinemäßig rechnet man mit einer Software möglichst viel störendes Rauschen heraus. Auf diese Weise erreichen erfahrene Strukturbiologen eine Auflösung von 3 Ångström. Damit kann man zwar Atome noch nicht sehen, aber man kann ein atomares Modell konstruieren, das eine Aussage über die Position einzelner Atome zulässt. Dabei fließen neben der berechneten 3D-Abbildung noch weitere Informationen ein, beispielsweise die Aminosäuresequenz sowie Bindungswinkel und -längen.
Manche Forscher geben sich mit 3 Ångström nicht zufrieden und treiben die Auflösungsgrenze immer weiter. Den Rekord von 1,25 Ångström halten aktuell die Gruppen von Holger Stark am Max-Planck-Institut für biophysikalische Chemie in Göttingen und von Sjors Scheres am Medical Research Council Laboratory of Molecular Biology in Cambridge (Nature, doi:10.1038/s41586-020-2833-4 und Nature, doi:10.1038/s41586-020-2829-0). Bei dieser Genauigkeit kann man sogar die kleinen Wasserstoffatome „sehen“.
Die große Herausforderung der Kryo-EM ist und bleibt allerdings die Probenpräparation. Man muss sein Molekül möglichst schonend und intakt isolieren, um es dann auf eine kleine Probenhalterung aufzubringen und in flüssigem Ethan in Windeseile einzufrieren. Vitrifizieren nennt man diesen Vorgang, bei dem das in der Probe enthaltene Wasser nicht kristallisiert, sondern glasartig erstarrt und dabei keine schädigenden Kristalle bildet. Die Eisschicht muss so dick sein, dass sie die Moleküle völlig umschließt. Größere Moleküle benötigen also dickere Eisschichten, die wiederum das Signal-Rausch-Verhältnis verschlechtern.
„Manche Proben lassen sich leider nicht intakt vitrifizieren. Das ist ein großes Problem“, sagt Böttcher. „Wir hatten schon Moleküle, die im EM vor dem Einfrieren sehr gut aussahen, aber danach war kein einziges intaktes Molekül mehr übrig, auf dem Grid sah es aus wie auf einem Schlachtfeld.“ Solche Geschichten kann wohl jeder erzählen, der sich mit Kryo-EM und Einzelpartikel-Analyse beschäftigt. Die Forscher hätten bisher keine Eigenschaft identifizieren können, die vorhersagen lässt, wie sich ein Molekül bei der Vitrifizierung verhalten wird, so Böttcher. Anderer Meinung ist Stark. Er glaubt, dass sich dieses Problem lösen lässt, wenn man auf jedwede Art von Chromatografie verzichtet (siehe hierzu das Interview mit Holger Stark auf Seite 48).
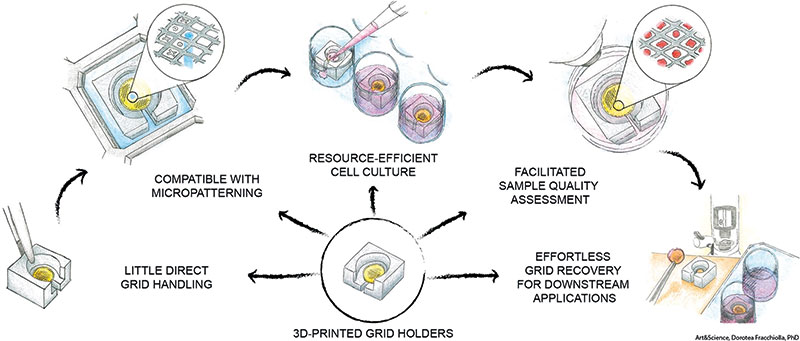
Schließlich muss sich der Strukturbiologe auch mit der Probenhalterung, dem Grid, beschäftigen. Dieses wenige Millimeter große, sehr empfindliche Gebilde besteht aus einem ringförmigen Rahmen und einer dazwischen aufgespannten Folie, auf der die Moleküle liegen. Es werden verschiedene Geometrien und Materialien verwendet. Gerne nimmt man dafür Gold, weil sich die Moleküle darauf weniger bewegen als auf anderem Material. „Diese Grids sind so weich, dass sie sich verbiegen, wenn man sie nur mit der Pinzette anfasst“, berichtet Florian Schur vom Institute of Science and Technology (IST) Austria in Klosterneuburg. Er und sein Team entwickelten deshalb einen Adapter, in den der Grid gelegt wird, damit man ihn während der Probenvorbereitung nicht mehr direkt berühren muss. Der Adapter kommt preiswert aus dem 3D-Drucker. Schur: „Wir kultivieren beispielsweise Zellen direkt auf den Grids in 6-Well-Platten. Weil die Grids oft aufschwimmen, müssen wir sie fixieren. Hier zeigt sich der große Vorteil des Adapters.“
Auch für kleinere Moleküle
Kryo-EM eignet sich sehr gut für Moleküle, die weder besonders groß noch sehr klein sind. Mit Molekulargewichten von weniger als 150 kDa tut man sich mit dieser Technik schwer. Noch, müsste man vielleicht sagen. Denn Radostin Danev entwickelte während seiner Zeit am Max-Planck-Institut für Biochemie in München eine sogenannte Volta-Phasenplatte. Damit konnten er und seine Kollegen die Struktur des nur 64 kDa großen menschlichen Hämoglobins mit bis zu 3,2 Ångström Auflösung rekonstruieren (Nat. Commun. 8: 16099). Im letzten Jahr gelang dies Forschern in La Jolla auch ohne eine Phasenplatte (Nat. Commun. 10: 1032).
Von besonders kleinen Molekülen kommen wir zu den mehrere Nanometer großen, komplexen Strukturen. Molekülkomplexe wie Ribosomen, Kernporen oder Nukleoli, Organellen und sogar das Zytoskelett lassen sich mit Kryo-Elektronentomografie (Kryo-ET) abbilden – sogar in ihrem zellulären Umfeld. So entdeckte beispielsweise das Team um Tomografie-Altmeister Wolfgang Baumeister vom Max-Planck-Institut für Biochemie in München, dass nukleäre Proteasomen an die Poren des Zellkerns andocken, wo sie als Türsteher fungieren und defekte Proteine abfangen können (PNAS 114: 13726).
Forscher aus Berlin, Heidelberg und Göttingen analysierten ein bakterielles Expressosom, das sowohl transkribiert als auch translatiert. Es besteht aus einem Ribosom, einer RNA-Polymerase und Transkriptions-Elongationsfaktoren. Die Forscher stellten anhand der Struktur fest, dass sich das Expressosom umbaut oder sogar ganz auseinanderfällt, wenn nur einer der beiden Prozesse unterbrochen wird (Science 369: 554).
Ganz aktuell sind die Elektronentomogramme der Spike-Proteine von SARS-CoV-2, die Forscher in Heidelberg, Frankfurt, Mainz und Langen veröffentlichten (Science 370: 203). 20 bis 40 Spikes sitzen auf der äußeren Hülle des Pathogens und bewerkstelligen die Bindung an die ACE2-Rezeptoren menschlicher Zellen. Die Proteine sind über drei Gelenke mit der Virushülle verbunden. Es ist denkbar, dass sie dank ihrer Flexibilität die Oberfläche einer Wirtszelle nach dem passenden Rezeptor regelrecht absuchen. Eine starke Glykosylierung schützt die Moleküle vor einem Angriff durch Antikörper.
„Kryo-Elektronentomografie schließt die Lücke zwischen Strukturbiologie und Zellbiologie“, sagt Martin Pilhofer vom Institut für Molekularbiologie und Biophysik an der ETH Zürich. Diese Technologie wurde in den letzten Jahren ebenfalls deutlich besser und anwenderfreundlicher. Auch sie profitiert von den neuen Detektoren. Zusätzlich bedurfte es aber der Entwicklung spezieller Tomografie-Software und schnellerer Grafikkarten, die mit den großen Datensätzen umgehen können. Die Zellen werden im vitrifizierten Eis hervorragend konserviert, die Tomogramme zeigen Schnappschüsse aus ihrem wahren Leben.
Allerdings ist eine Zelle zu dick für die direkte Mikroskopie. Zum Verschlanken verwendet man eine Methode namens Focused Ion Milling, kurz FIB. Dabei wird ein fokussierter Ionenstrahl wie ein Sandstrahler eingesetzt, der von zwei Seiten Schicht um Schicht von der Zelle abträgt, bis nur noch eine 200 bis 300 Nanometer dicke Lamelle übrig ist. Diese Kryo-FIBs kann man heute zwar kaufen, Forscher streben aber nach weiteren Verbesserungen, beispielsweise Automatisierung.
Eine automatische FIB bauten Pilhofer und sein Team auf. „Unsere FIB kann Zellen an verschiedenen Positionen auf dem Grid automatisch ansteuern und an jeder Zelle alle Ätzschritte nacheinander abarbeiten“, erklärt Pilhofer. „Das beschleunigt das Experiment deutlich und man muss auch nicht dauernd am Gerät sitzen.“
Gemittelte Sub-Tomogramme
Die Wissenschaftler untersuchen, wie Bakterien untereinander und mit anderen Organismen kommunizieren. Filamentöse Cyanobakterien beispielsweise sind zwar individuelle Zellen, bilden aber mehrzellige Strukturen. Über bisher wenig untersuchte Kanäle treten sie miteinander in Kontakt und tauschen Stoffe aus. Das Elektronentomogramm enthüllte nicht nur die etwa 50 Nanometer langen, verbindenden Kanäle, sondern auch unbekannte Strukturen an den Enden dieser Kanäle (Cell 178: 374). Aus sogenannten gemittelten SubTomogrammen ermittelten die Forscher eine höher aufgelöste Abbildung dieser Gebilde. Es zeigte sich, dass an beiden Öffnungen jedes Kanals in Höhe der Zellmembran Stopfen sitzen. Über diesen thronen im Zytoplasma Kappen, die wie Zahnräder mit fünf Zähnchen aussehen. „Wir konnten zeigen, dass ein gestresstes Bakterium den Kanal mit den Stopfen und Kappen schließen und die Kommunikation unterbrechen kann. So kann es verhindern, dass die ganze Kolonie geschädigt wird, wenn es verletzt oder mit einer toxischen Substanz kontaminiert wird,“ erklärt Pilhofer.
Die Mittelung von Sub-Tomogrammen ist ein Werkzeug, um aus einem weniger gut aufgelösten Bild eine hochaufgelöste Struktur zu rekonstruieren. Diese Arbeitsweise ist nicht neu, aber die Möglichkeiten dazu waren lange begrenzt. „Die Struktur entsteht durch die Mittelung von Ausschnitten der Bilder. Da ich für ein Tomogramm nur eine Probe messe, muss diese viele Kopien der betreffenden Struktur enthalten. Die Kunst ist, diese Kopien aufzuspüren, zu identifizieren und deren Orientierungen zu bestimmen“, sagt Schur. Als er noch in der Arbeitsgruppe von John Briggs am EMBL in Heidelberg war, entwickelte er die passende Software weiter und publizierte von einem viralen Gag-Protein die erste aus Sub-Tomogrammen entwickelte Struktur mit Subnanometer-Auflösung (J. Struct. Biol. 184: 394). Später konnte er am Beispiel eines viralen Capsid-Proteins zeigen, dass sich die Auflösung mit einem DED auf 3,9 Ångström verbessern ließ (Science 353: 506).
Die Lokalisierung spezifischer Moleküle oder einzelner Viren und Phagen in Zellen ist schwierig, denn die Moleküle werden für die EM nicht markiert. Die Lösung dieses Problems ist die Kombination von Fluoreszenzmikroskopie bei unter minus 140 Grad Celsius und Kryo-ET. Die Fluoreszenzsignale, die man von der bereits vitrifizierten Probe aufnimmt, verraten die Position der gesuchten Partikel, sodass man sie im EM gezielt anfahren kann. Für Kryo-ET und Kryo-EM sieht die Zukunft ziemlich rosig aus. Das dürfte auch FEI Thermo Fisher Scientific und Jeol freuen, die die hochleistungsfähigen 300-kV-Geräte herstellen. Zeiss stieg vor vielen Jahren aus dem Geschäft mit Transmissions-Elektronenmikroskopen aus. Vielleicht ärgert man sich in Oberkochen heute darüber.
Allerdings wird sich nicht jede Universität so ein Gerät leisten können. Die Mikroskope kosten nicht nur etliche Millionen Euro. Die tonnenschweren Apparate benötigen auch spezielle Räume mit verstärkten Böden und besonderer Abschirmung, die vor der hohen Spannung schützt. Obendrein schlägt der Unterhalt mit über 3.000 Euro täglich zu Buche. Daher setzt man in vielen Ländern auf Zentralisierung der Expertise und der Ausstattung. Am Simons Center in New York stehen beispielsweise sieben dieser Hochleistungsgeräte. Trotzdem muss man auf Messzeit warten, beklagten kürzlich Forscher in Science. Einer berichtete frustriert, dass er, nachdem er endlich Messzeit bekommen hatte, feststellen musste, dass seine Proben nicht viel taugten. Mit neuen Proben musste er sich wieder in die Warteschlange einreihen.
Cryo-EM für alle
Dieses Problem könnte allerdings mit der von einigen Wissenschaftlern geforderten „Demokratisierung“ der Kryo-EM gelöst werden. Sie glauben, dass auch weniger teure 100-kV-Geräte mit geringerer Leistungsfähigkeit aber spezieller Ausstattung schon viele wertvolle Beiträge zu strukturbiologischen Fragen liefern können. Mit diesen könnte man nicht nur die Qualität seiner Probe vor dem Auftritt im 300-kV-Mikroskop überprüfen – Richard Henderson und seine Kollegen in Cambridge sind davon überzeugt, dass diese Geräte auch brauchbare Daten generieren.
Glauben ist aber nicht wissen. Die Gruppe demonstrierte deshalb anhand von fünf bereits gut analysierten Proteinen mit Molekulargewichten von 64 kDa bis 4,5 MDa, dass man auch den kleineren Geräten Bilder entlocken kann, aus denen sich gute Strukturen rekonstruieren lassen (IUCrJ 6: 1086). Die Auflösung war bei dieser Studie im Wesentlichen durch den Detektor limitiert, der nicht schnell genug arbeitete und dessen Pixel zu klein waren.
In einer E-Mail an die Autorin schrieb Henderson: „I may have said previously that we are trying bribery, bullying and blackmail, but in the end it may be that it is persuasion, plus the weight of scientific proof, that will work.“ An diesem wissenschaftlichen Beweis arbeitet Henderson zusammen mit seinem Kollegen vom MRC Christopher Russo. „Bis zur Verwirklichung wird es aber“, so Henderson, „noch einige Jahre dauern.“
Last Changed: 11.11.2020