Special Mikrobiologie
Leben auf dem Chip
von Karin Hollricher, Laborjournal 11/2018
In Bakterienpopulationen gleicht nicht jede Mikrobe der anderen. Es gibt genetische Unterschiede – und phänotypische Ungleichheiten, deren Ursachen im Metabolismus der Zelle liegen. Diese metabolische Heterogenität finden viele Mikrobiologen äußerst spannend. Für die biotechnologische Produktion ist sie ein Problem.
Das Phänomen phänotypische Heterogenität in Bakterienpopulationen wurde erstmals von John Spudich und Daniel Koshland 1976 beschrieben (Nature 262: 467-71). Obwohl diese Entdeckung schon über vierzig Jahre zurückliegt, weiß man noch immer nicht viel darüber. Offensichtlich nutzt nicht jede Zelle alle physiologischen Synthese- und Abbauwege gleichmäßig, anscheinend gibt es funktional relevante metabolische Unterschiede zwischen den Zellen. Die Daten dazu sind aber – sagen wir mal – übersichtlich.
Wie kann man der Ursache einer Veränderung im metabolischen Phänotyp einer Population überhaupt auf die Spur kommen? „Um phänotypische Heterogenität zu analysieren, müssen wir auf Einzelzell-Experimente zurückgreifen. Zu diesem Zweck haben wir Mikrofluidik-Chips entwickelt, in denen wir kleine Kulturen aus einer einzelnen Zelle wachsen lassen und analysieren können“, sagt Dietrich Kohlheyer. Er ist kein Biologe, sondern Mechatronikingenieur und arbeitet am Institut für Bio- und Geowissenschaften am Forschungszentrum in Jülich.
Perfekt passend
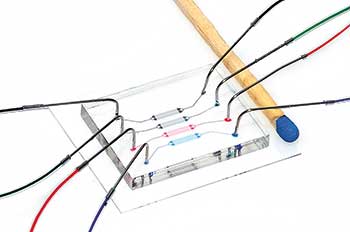
Der Herstellungsprozess eines Mikrofluidik-Chips beginnt am Computer. Wie soll der Chip aussehen, wie funktionieren? Mittels CAD (Computer-Aided Design) entwickeln die Forscher die Struktur des Chips. Das Modell von Kohlheyer und Co. soll neben einem Eingang, der etwa verschiedene Medien einschleust, auch einen Ausgang enthalten. Auf den geplanten Chip passen vier Versuchskanäle gleichzeitig. Das Herz des Mikrokulturgefäßes wird aus hunderten parallel angeordneten Kammern bestehen, deren Höhe den zu untersuchenden Mikroorganismen entspricht, also bei Bakterien in vielen Fällen gerade mal einen Mikrometer. Jedes Reaktionsgefäß – wenn man es so nennen will – soll etwa fünfzig mal fünfzig Mikrometer groß werden und einige wenige Pikoliter Flüssigkeit aufnehmen.
Dann geht es in den Reinraum. Im Schutzanzug und mit Maske ausgerüstet beschichten die Forscher etwa CD-große Silizium-Wafer mit einem zähflüssigen Photolack. Darauf legen sie eine Photomaske, um die erste Lage Mikrochip-Strukturen herzustellen. Der Wafer wird belichtet und der Photolack härtet unter kurzwelligem Licht zu einer stabilen Struktur aus. Alles, was nicht belichtet wurde, waschen die Biotechnologen in mehreren Schritten vom Wafer herunter. Danach fixieren sie die verbliebenen Strukturen und es kommt eine zweite Lage Mikro-Strukturen oben drauf.
Der strukturierte Wafer ist das Negativ, den die Forscher künftig zigfach für die Chipherstellung benutzen werden. Ist er fertig, können die Wissenschaftler die Reinraum-Klamotten ablegen und zurück ins gewöhnliche Labor gehen. Hier formen sie die Strukturen auf dem Wafer mit einem elastischen Silikon namens Polydimethylsiloxan (PDMS) ab. Das PDMS tragen sie flüssig auf, verfestigen es mit Wärme und ziehen es schließlich vom Wafer ab. Das gummiartige Gebilde zerschneiden sie mit dem Skalpell vorsichtig in einzelne Chips. An jedem stechen sie nun noch die Ein- und Ausgänge mit einer Nadel auf – dann kann der biologische Teil beginnen.
Pingelige Biotechnologen
Der Chip kommt auf ein Mikroskop, das automatisch im Zeitraffer aufnehmen kann. Medienreservoire und Abfall-Eppi werden angeschlossen, dann „schießt“ die Flüssigkeit mit einer Geschwindigkeit von 200 Nanoliter pro Minute durch die winzigen Kanäle, Schleusen und Kulturschalen.
„Wir sind mit der Herstellung der Medien sehr pingelig“, gibt Kohlheyer zu. „Wir haben nämlich mit Medien angefangen, welche die Mikrobiologen immer verwenden. Dabei mussten wir feststellen, dass die Kulturen damit im Chip nicht gut zurechtkamen. Die winzigen Kanäle und Kammern werden meist kontinuierlich mit frischem Medium versorgt, und so machen minimale Differenzen in der Zusammensetzung wie auch minimale Verunreinigungen bei den geringen Zellzahlen oft große Unterschiede. So fanden wir beispielsweise Mikropartikel, welche die Kanäle unserer Chips verstopft haben.“ Darum mischen sie die Kulturmedien lieber selber.
Nach dem ersten Spülen der Chips schleusen die Forscher die Bakterien ein, bis in jeder Kammer eine Zelle angekommen ist – oder zumindest in den meisten Reaktorgefäßen. Manche bleiben auch leer, in andere gelangt mehr als eine Zelle. Dort bleiben sie zwischen den Oberflächen hängen – die Höhe der Kulturkammern ist so gewählt, dass die Bakterien, wie bereits erwähnt, gerade so dazwischen passen. Bei zu hohen Flussraten können die Zellen natürlich einfach „durchflutschen“. Hier muss man die optimalen Einstellungen finden.
Anschließend wird wieder das Medium angeschlossen. Ist alles eingestellt und sind die Kulturgefäße mikroskopisch überprüft, bleiben die Zellen sich selbst überlassen, das Mikroskop beobachtet sie automatisch. Anhand der Aufnahmen können die Biotechnologen später das Verhalten der Bakterien und ihrer Nachkommen, ihre Morphologie oder die Intensität von Fluorophoren dokumentieren. Und sie können beschreiben, wie schnell die einzelnen Kolonien gewachsen sind sowie welche Größe die Zellen erreichen, bevor sie sich wieder teilen. Das Set-up ist so weit ausgereift, dass man weitergehende, kompliziertere Experimente machen kann. Kohlheyer möchte beispielsweise die Zellen komplexerer Mischkulturen einzeln kultivieren und anschließend getrennt wieder aus dem Chip herausholen.
Verwirrendes Wachstum
Wie verhalten sich die Bakterien nun in ihrem Nano-Ökosystem? Überraschend! Mikroskopische Einzelzell-Untersuchungen erlauben die räumliche und zeitliche Analyse kleinerer Populationen und bringen damit ein altes Postulat zum Wanken: die Wachstumskinetik von Jaques Monod.
Der französische Nobelpreisträger hatte Beobachtungen von Mikrobiologen in eine Theorie gefasst, wonach eine frisch angeimpfte Kultur zunächst einmal eine Lag-Phase, auch Latenzphase genannt, durchläuft, in der sich die Bakterien an die neue Umgebung gewöhnen, bevor sie in die Phase des exponentiellen Wachstums eintreten. „Im Mikrochip wachsen Bakterien oftmals unerwartet anders – ohne jegliche Lag-Phase“, erzählt Kohlheyer. „Die Wachstumskinetik von Monod basiert darauf, dass die Kulturen homogen sind und homogen wachsen.“ Das seien sie aber eben oft nicht und so verhielten sie sich auch nicht.
„In den Chips beobachten wir, dass manche Zellen sofort nach dem Beimpfen mit der Zellteilung beginnen, andere wachsen gar nicht oder erst viel später.“ Vermutlich wachsen in den großen Kulturen und Fermentern ebenfalls nicht alle Zellen sofort los, meint Kohlheyer.
„Die üblichen Analyseverfahren, wie zum Beispiel die Messung der optischen Dichte, können die anfänglich sehr geringen Zellzahlen überhaupt nicht auflösen. Somit beobachtet man oft nur eine scheinbare Lag-Phase, und zwar solange, bis die gesamte Population aus der Messungenauigkeit herauswächst. Derartige Heterogenitäten im Zellwachstum kann man mittlerweile in allen Wachstumsphasen von Bakterienkulturen aufdecken. In Bakterienpopulationen findet zudem häufig eine komplexe Arbeitsteilung statt. Die Zellen mögen genetisch identisch sein, physiologisch sind sie es aber nicht – und das sehen wir bei unseren Chip-Experimenten in Form von Wachstum oder genetisch integrierten Fluoreszenzreportern, welche sonst unsichtbare molekulare Mechanismen in ein Lichtsignal umwandeln.“
Außerdem wüchsen die Kulturen insgesamt schneller. „Vielleicht liegt das daran, dass wir ständig neues, unverbrauchtes Medium zuführen und Ausscheidungsprodukte abführen“, mutmaßt der Forscher.
In naher Zukunft wollen die Wissenschaftler ihre Apparatur so überarbeiten, dass sie auch die Umweltbedingungen verändern können, um erstens die Auswirkungen der dynamischen Änderungen zu beobachten – und zweitens auch mit komplexeren, beispielsweise anaeroben und thermophilen Organismen arbeiten zu können.
Zellen im Mikro-Inkubator
Die Jülicher Biotechnologen sind natürlich nicht die Einzigen, die Mikrofluidik-Chips für die Einzelzell-Analyse entwickeln. Es gibt bereits höchst unterschiedliche Designs. So konstruierten Forscher der Arbeitsgruppe von Andreas Schmid am Helmholtz-Zentrum für Umweltforschung (UFZ) in Leipzig den Envirostat für das kontaktfreie Positionieren einzelner Zellen im Mikro-Inkubator. Mehrere Elektroden werden an jeden Kulturbehälter, der nur wenige Pikoliter aufnehmen kann, angeschlossen und die Zelle darin mithilfe eines schwachen, inhomogenen, elektrischen Felds „festgehalten“ (Lab Chip 9: 576-85; Lab Chip 13: 397-408).
Bei der Bewertung der Ergebnisse aus der Einzelzell-Analytik sollte man jedoch Vorsicht walten lassen. Vereinzelten Bakterien fehlen die Nachbarn, die sie in natürlichen Populationen haben. Das kann sich wiederum auf den Phänotyp auswirken. Außerdem ist das PDMS nicht völlig inert, es kann beispielsweise kleine Moleküle binden: So braucht man sich nicht wundern, wenn der Inhibitor in der Kulturflasche oder auf der Petrischale funktioniert, aber nicht im Chip-Experiment.
Wie ist denn nun die Nachfrage nach solchen Einzelzell-Chips? Kohlheyer: „Die Industrie ist noch vorsichtig. Dabei könnten wir im Chip die Individualität darstellen, aber auch die Homogenität erhalten, weil jede Starterzelle die gleichen Bedingungen hat. Ein Ziel für industrielle Anwendungen wäre beispielsweise, robustere Produktionsstämme zu züchten oder herauszufinden, welche Bedingungen im Fermenter man wie ändern müsste, um die Heterogenität in der Kultur möglichst gering zu halten. Auf Seiten der Forschung sehen wir deutlich mehr Interesse, etwa bei Molekularbiologen, die Mechanismen für Anpassung an Umweltstress und Überleben untersuchen und versuchen herauszufinden, was eine einzelne, etwas anders tickende Zelle für die Population bedeutet.“
Prominentes Thema, wenn es um Anpassung und Überleben geht, sind natürlich Antibiotika-Resistenzen. Eine Infektionskrankheit, die nach einer Therapie wieder aufflammt, muss aber nicht unbedingt von genetisch resistenten Bakterien verursacht werden, sondern kann von Organismen hervorgerufen werden, welche die Therapie schlicht verschlafen haben und erst danach aktiv wurden. Solche Zellen heißen Persister. Die temporäre Unempfindlichkeit gegenüber gleich mehreren Antibiotika-Typen zeichnet sie aus. Schätzungsweise jede zehntausendste bis millionste Zelle einer Kolonie ist ein Persister. Weil diese Zelle sich genetisch aber nicht von den empfindlichen Zellen unterscheidet, führen Experimente an Populationen nicht weit. Kohlheyer ist überzeugt, dass Einzelzell-Experimente helfen können herauszufinden, warum Bakterien zu Persistern werden.
Last Changed: 08.11.2018