Gefaltete Netze analysieren Bilder von Zellen
Produktübersicht: High-Content-Screening-Systeme
High-Content-Screening-Systeme 
High-Content-Screening-Systeme bewerten in kurzer Zeit Abermillionen Bilder. Sinnvolle Ergebnisse kommen dabei nur mit intelligenten AuswerteAlgorithmen heraus.
Auf den ersten Blick sieht man vielen High-Content-Screening-Systemen nicht an, dass sich hinter ihrem unscheinbaren, eher an einen Scanner oder Laser-Drucker erinnernden Äußeren ein inverses Fluoreszenz-Mikroskop verbirgt. Wie am Fließband schießt es Bilder von Mikrotiterplatten, die ein Nanopositionierer auf dem Mikroskop-Tisch verschiebt, um die einzelnen Wells exakt in den Fokus des Objektivs zu rücken.
Das automatische Fluoreszenz-Mikroskop und das dazugehörige optische System aus Quecksilberlampe, LEDs, Lasern, empfindlicher CMOS- oder CCD-Kamera und raffiniertem Spiegelsystem sind aber für sich genommen nichts Besonderes: Der Knüller an HCS-Geräten sind die integrierten, zumeist auf sogenannten gefalteten (Convolutional) neuronalen Netzen (CNN) basierenden lernfähigen Bilderkennungsprogramme, die auch feinste Details erkennen und Millionen von Bilder in kürzester Zeit auswerten können.
Die Idee, die Struktur von Computer-Algorithmen und Software-Programmen ähnlich aufzubauen wie ein Gehirn, das aus zahllosen Nervenzellen beziehungsweise Neuronen besteht, die über Axone und Synapsen miteinander vernetzt sind, ist nicht neu. Bereits in den sechziger Jahren des zwanzigsten Jahrhunderts entwickelten Informatiker die grundlegenden Strukturen neuronaler Netze: Die kleinste Recheneinheit des Netzes sind miteinander verbundene „Neuronen”, die aus verschiedenen Richtungen Eingaben in Form von Zahlenwerten mit einer bestimmten Gewichtung empfangen und diese aufsummieren. Übersteigt die Eingabe-Summe einen Schwellenwert, der sich durch eine mathematische Operation einstellen lässt, „feuert” das aktivierte Neuron und gibt den Wert als Ausgabe-Signal an das nächste Neuron weiter (siehe hierzu auch den Hintergrund-Artikel von Mario Rembold in LJ 3/2019, Seite 18).
In frühen neuronalen Netzen waren sämtliche Neuronen miteinander verknüpft, was zu komplizierten Berechnungen führte, die selbst leistungsstarke Computer überforderten. Moderne neuronale Netze bestehen dagegen aus vielen versteckten Neuronen-Schichten. Die Ausgabe-Signale der einzelnen Lagen werden gesammelt und als Eingabe an die nächste Schicht weitergegeben. Diese sogenannten tiefen, neuronalen Netze beschleunigen die Übertragung der Daten und sind zudem auch lernfähig, weshalb sie auch als Deep-Learning-Neuronale-Netze bezeichnet werden.
Mit Bildpunkten gefüttert
Und was hat das Ganze mit der Bilderkennung in HCS-Geräten zu tun? In diesen bestehen die Daten für die Eingabeschicht des neuronalen Netzes aus den einzelnen Bildpunkten (Pixel) der von der Mikroskop-Kamera aufgenommenen digitalen Zell-Bilder. Für den Computer sind Pixel nichts anderes als Zahlenwerte zwischen 0 und 255, die für die Helligkeit des Bildpunktes stehen: 0 ist komplett dunkel, 255 maximal hell. Kombiniert man auf diese Weise Rot, Grün und Blau (RGB), lässt sich jede beliebige Farbe durch die entsprechenden Farbwerte für R, G und B ausdrücken. So codieren zum Beispiel die RGB-Werte 6, 250 und 7 einen bestimmten Grünton.
In digitalen Bildern sind die einzelnen Pixel rasterförmig in einer zweidimensionalen Matrix angeordnet und können so als Zahlenwerte in die Eingabeschicht des neuronalen Netzwerks übertragen werden. Jedes Neuron ist hierbei mit dem Farbwert eines Bildpixels verbunden. Dies hat zur Folge, dass schon kleine Bildgrößen riesige Datenmengen produzieren. So enthält zum Beispiel ein dreifarbiges Bild mit 1.000 x 1.000 Pixeln bereits drei Millionen gewichtete Werte, die von den Neuronen der Eingabeschicht verarbeitet werden müssen.
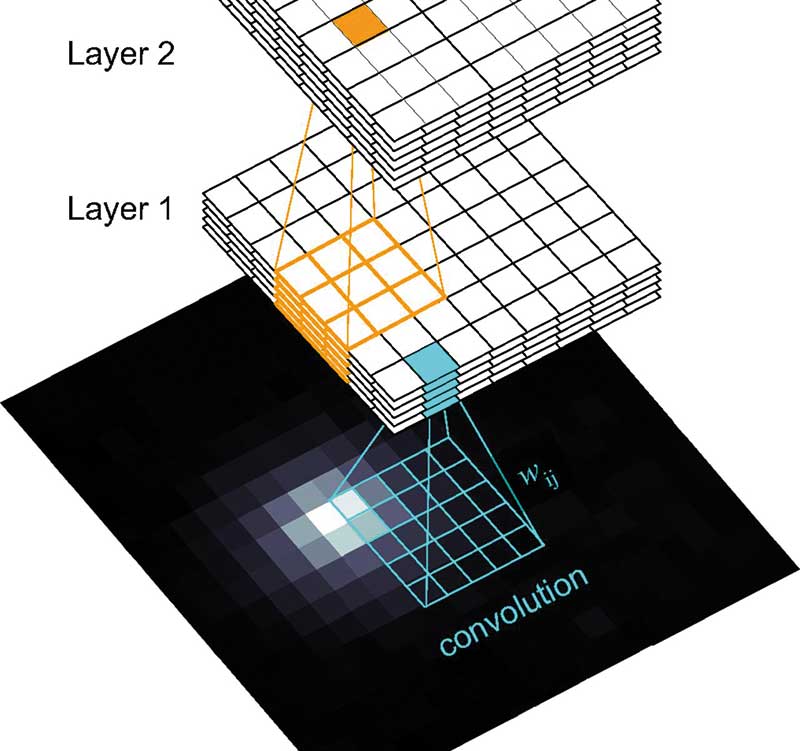
Japanische Informatiker erfanden deshalb in den achtziger Jahren einen genialen Trick: Sie reduzierten die Bilddaten, indem sie das Bild mit einer sogenannten Convolution-Matrix zusammenfalteten. Die Faltungsmatrix ist in der Regel eine simple Matrix mit ungerader Spalten und Zeilenzahl, üblich sind 5x5- oder 3x3-Matrizen, die wohlüberlegte Zahlenwerte enthalten: Etwa 0, 1, -1 oder auch größere zumeist einstellige positive oder negative Zahlen.
Die auch als Kernel bezeichnete Matrix wird Schritt für Schritt über entsprechende Pixelausschnitte des digitalen Bildes geschoben. Durch einfaches Multiplizieren der Matrixwerte mit den Farbwerten der Bildpixel und anschließendes Aufsummieren berechnet sie einen neuen vereinfachten Ausgabewert für den gescannten Bildausschnitt. Kernels reduzieren aber nicht nur die Datenmenge tiefer, gefalteter neuronaler Netze (DCNN) erheblich: Je nach gewählten Matrix-Werten verbessern sie auch die Bilderkennung und wirken zum Beispiel als Schärfe-, Maskierungs- oder Kantenerkennungs-Filter.
Wie man ein solches tiefes, gefaltetes neuronales Netz für die Erkennung charakteristischer Merkmale in Aufnahmen von Tumor-Organoiden einsetzt, beschreibt Michael Boutros Gruppe vom Deutschen Krebsforschungszentrum in Heidelberg in einem aktuellen Vorabdruck auf bioRxiv (doi.org/10.1101/660993).
Künstliche Mini-Tumore
Für ihre Phänotypisierungs-Experimente züchtete die Gruppe Organoide aus Darmkrebs-Zellen, die von endoskopischen Biopsien an Darmkrebs leidender Patienten stammten. Nachdem die Organoide eine gewünschte Größe erreicht hatten, wurden sie zerkleinert und als Zellsuspension gleichmäßig in die Wells einer 384-Mikrotiterplatte überführt. Die kleinen Mini-Tumore durften anschließend etwas weiterwachsen und wurden danach sukzessive mit mehr als 500 chemischen Verbindungen behandelt – darunter 63 klinisch relevante Krebs-Wirkstoffe sowie Chemikalien, die Kinasen attackieren oder in Zellwachstums-Signalwege eingreifen.
Die von den chemischen Verbindungen verursachten morphologischen Veränderungen der Organoide hielt die Kamera eines automatischen konfokalen HCS-Mikroskops fest, die mehr als drei Millionen Bilder der behandelten Tumor-Organoide schoss. Um dabei ein möglichst realistisches Bild zu erhalten, nahm sie Bilder von verschiedenen Fokus-Ebenen in der axialen Richtung (Z-Ebene) auf.
Für die anschließende Bildanalyse setzte Boutros Mannschaft zunächst ein Programm ein, das die Umrisse beziehungsweise Segmente der einzelnen Organoide anhand von Fluoreszenz-Intensitäten erkennen sollte. Diese Intensitäts-Segmentierung reichte jedoch nicht aus, um die Organoide zweifelsfrei zu identifizieren. Die Gruppe fütterte deshalb ein tiefes, gefaltetes neuronales Netz mit den unvollständigen Segmentierungs-Daten und trainierte es auf diese Weise, phänotypische Merkmale in den Tumor-Organoiden zu erkennen. Das DCNN spürte in den von neunzehn Patienten stammenden unbehandelten Tumor-Organoiden fast 500 phänotypische Merkmale auf, zu denen zum Beispiel unterschiedliche Organoid-Formen oder Texturen zählten.
Unterschiedliche Phänotypen
Interessant ist, dass zwischen den Tumor-Organoiden der einzelnen Patienten deutliche Unterschiede bestanden. Boutros Team konnte sie in sechs phänotypische Klassen einteilen, die sich durch ihren Organisationsgrad unterschieden. So waren einige eher rund und regelmäßig geformt, andere dagegen unregelmäßig und kompakt.
Um zunächst herauszufinden, welche Verbindungen der Substanz-Bibliothek für die Tumor-Organoide tödlich sind, behandelten die Wissenschaftler sie mit den entsprechenden Chemikalien. Für die Auswertung kombinierten die Heidelberger die Merkmal-Erkennung über das DCNN mit einem sogenannten Zufallswald-Algorithmus, einem maschinellen Lernalgorithmus, der üblicherweise für Klassifizierungen eingesetzt wird – in diesem Fall tot oder lebendig. Anschließend zeigten sie dem Analyse-System lebende und mit dem Zytostatikum Bortezomib getötete Organoide, wodurch es lernte, zwischen toten und quietschfidelen Tumor-Organoiden zu differenzieren.
Ursachenforschung
Auch für die Analyse von Experimenten mit der Substanz-Bibliothek, die zeigen sollten, welche phänotypischen Veränderungen die Wirkstoffe in den Tumor-Organoiden verursachten, setzte Boutros Team ergänzende maschinelle Lernalgorithmen ein. Mit ihrer Hilfe tauchte die Gruppe immer tiefer in die molekularen Mechanismen ein, die letztlich zu den verschiedenen Phänotypen der behandelten Tumor-Organoide führten.
Sehr faszinierende Arbeit – aber auch derart gespickt mit wilden Verfahren und Begriffen aus der Welt der Bioinformatiker, dass man als einfacher Biologe oder Biochemiker fast keine Chance mehr hat durchzusteigen. Sie zeigt zudem, dass es nicht damit getan ist, sich einfach ein HCS-Gerät zuzulegen, wenn man entsprechende Screenings durchführen will. Man muss auch wissen, wie man das Instrument mit entsprechenden Programmen und Algorithmen dazu bringt, verlässliche Ergebnisse zu liefern.
High-Content-Screening-Systeme im Überblick 
(Erstveröffentlichung: H. Zähringer, Laborjournal 10/2019, Stand: September 2019, alle Angaben ohne Gewähr)
Letzte Änderungen: 10.10.2019