Teuflisch schönes Licht
Methoden-Special: Neue Biolumineszenz-Systeme
Andrea Pitzschke
(08.12.2020) Biolumineszierende Organismen sind nicht nur aus optischen Gründen faszinierend. Sie liefern auch die Komponenten für zahlreiche Reportergen-Assays und Biolumineszenz-Imaging-Verfahren. Mit einem neuentdeckten Biolumineszenz-System aus Pilzen kann man sogar Pflanzen zum Leuchten bringen.
Verglichen mit populären Fluoreszenzproteinen wie dem Grün fluoreszierenden Protein (GFP) oder mCherry sehen Biolumineszenz-Systeme manchmal etwas blass aus. Das liegt vor allem an ihrem relativ schwachen Signal-zu-Hintergrund-Verhältnis. Die Biolumineszenz kann aber einen Trumpf ausspielen, der sie für zahlreiche biologische Assays und Techniken äußerst attraktiv macht: Sie benötigt zur Anregung keine Lichtstrahlen, die für Zellen gefährlich sind und zum Ausbleichen von Fluoreszenzproteinen führen können.
Biolumineszierende Organismen reichen von Bakterien über Glühwürmchen bis zu Pilzen. Ihre spezifischen Luciferine und Luciferasen liefern die Bausteine für Biolumineszenz-basierte Assays, die Biowissenschaftler rund um die Uhr für ihre Experimente einsetzen. Die Luciferase oxidiert das zugehörige Luciferin zu elektronisch angeregtem Oxyluciferin, das rasch in den stabileren Grundzustand übergeht und dabei Photonen aussendet. Bei Biolumineszenz-basierten Reporter-Assays platziert man das Luciferase-Gen stromabwärts des Zielgens, exprimiert das Konstrukt in einem passenden Zelltyp und gibt das Substrat Luciferin zu einem Zelllysat. Wird das Zielgen exprimiert, findet die Reaktion von Luciferase mit Luciferin statt und die entstehenden Lichtsignale werden registriert. Auch wenn man die Biolumineszenz einsetzt, um zum Beispiel Zellen für das Zell-Imaging zu markieren, muss Luciferin bei den gängigen Systemen von außen zugeführt werden.
Leuchtende Bakterien
Diesen Nachteil kann man mit dem Biolumineszenz-System von Leuchtbakterien umgehen. Bei diesen sitzen sämtliche Komponenten, die für die Biolumineszenz nötig sind, auf dem sogenannten luxCDABE-Operon. Die von luxAB codierte Luciferase katalysiert die Reaktion des Luciferins FMNH2 (reduziertes Flavin-Mononukleotid) mit Sauerstoff und einem Fettaldehyd zu FMN, Wasser und einer Fettsäure, bei der Biolumineszenzlicht entsteht. Anschließend überführt die von luxCDE codierte Fettsäure-Reduktase die Fettsäure unter Energieaufwand wieder in einen Aldehyd, während FMN durch eine Flavin-Reduktase zu FMNH2 reduziert wird.
Für viele Imaging-Techniken reicht die Helligkeit der bakteriellen Biolumineszenz jedoch nicht aus. Die Postdoktorandin Carola Gregor aus Stefan Hells Gruppe am Max-Planck-Institut für biophysikalische Chemie in Göttingen fand jedoch einen Weg, mit dem man das Licht in den Leuchtbakterien heller aufdrehen kann (PNAS 115: 962-67). Dazu erweiterte sie das luxCDABE-Operon des Bakteriums Photorhabdus luminescens mit dem frp-Gen des Wasserbakteriums Vibrio campbellii, das für eine Flavin-Reduktase codiert. Anschließend schleuste sie das aufgepeppte Operon in E. coli ein und optimierte es durch Zufallsmutagenese und entsprechende Screenings. Das hierdurch entstandene ilux-Operon (improved lux operon) erzeugte in E. coli eine siebenmal hellere Biolumineszenz als das luxCDABE-Operon, wodurch auch einzelne Zellen unter dem Mikroskop sichtbar waren.
Das ilux-Operon ist aber nicht nur auf E. coli beschränkt, sondern lässt sich auch an Säugerzellen adaptieren. Gregor und ihre Kollegen optimierten die Codons hierzu zunächst mit einem entsprechenden Software-Tool. Anschließend synthetisierten sie die Gene, klonierten sie separat in Expressions-Vektoren und cotransfizierten mit diesen HEK293-Zellen. Am hellsten leuchteten die Zellen mit diesem sogenannten co-Lux-System, wenn die Forscher einen dreifachen Überschuss der Fettsäure-Reduktase-codierenden luxC-, luxD- sowie luxE-Plasmide in die Zellen einschleusten. Die Synthese des Aldehyds scheint also der Geschwindigkeits-bestimmende Schritt der Reaktion zu sein. Wird das co-Lux-System effektiv in den Zellen exprimiert, ist die Lumineszenz ähnlich stark wie bei der Feuerfliegen-Luciferase.
Dank ihrer Farbvielfalt können fluoreszierende Reportermoleküle verschiedene Informationen über eine Probe liefern. Mit biolumineszierenden Reportern ist dies nicht so einfach möglich, obwohl ihr Biolumineszenz-Spektrum ebenfalls von Blau bis Rot reicht. Luciferasen reagieren zum einen oft mit fremden Luciferinen über Kreuz, zum anderen überlappen häufig auch ihre Spektren. Mit kommerziellen Systemen, die zwei Luciferase-Reporter enthalten, sind Analysen deshalb nur nacheinander möglich. Ein Beispiel hierfür ist das Dual-Luciferase-Reporter-System aus Firefly-Luciferase (Fluc) und Luciferin sowie Seefeder-Luciferase (Rluc) und Coelenterazin.
Wahlweise Grün oder Rot
Martha Grossels Team vom Connecticut College in New London, USA, schuf deshalb per Zufallsmutagenese zwei sehr ähnliche Fluc-Varianten: Eine setzt Firefly-Luciferin um, wodurch eine rote Biolumineszenz entsteht, die andere Benzothiophen-Luciferin, woraus eine grüne Biolumineszenz resultiert. Mit diesem Dual Analyte Reporter with Two Firefly Luciferase Substrates (DART)-Assay ist es möglich, zwei Biolumineszenz-Signale gleichzeitig zu messen (Sci. Rep. 8: 5990).
Besonders ausgeklügelt ist das Nanobit-Biolumineszenz-System, mit dem man Zellantworten auf Proteinebene untersuchen kann, etwa posttranslationale Modifikationen (Commun. Biol. 3: 8). Hierzu benötigt man neben dem Luciferase-Enzym zwei Primär-Antikörper gegen das Zielprotein: einen aus Kaninchen und einen aus der Maus. Die eingesetzte Luciferase stammt ursprünglich aus der Tiefsee-Garnele Oplophorus gracilirostris, wurde jedoch gentechnisch modifiziert und in ein System mit zwei getrennten Untereinheiten umgebaut. Die große Untereinheit (LgBiT) ist 18 kDa schwer, die kleine (SmBiT) 11 Aminosäuren lang. Eine enzymatisch aktive Luciferase entsteht nur, wenn sich 18-kDa-Domäne und 11-aa-Peptid nahe genug kommen. Die beiden Untereinheiten hängen an einem sekundären anti-Maus- beziehungsweise anti-Kaninchen-Antikörper.
Das Versuchs-Protokoll ist überschaubar und besteht im Wesentlichen darin, das Zelllysat mit den Primär- und Sekundärantikörpern zu inkubieren. Die Sekundärantikörper erkennen ihre jeweiligen Primärantikörper und nähern sich so weit an, dass sich die anhängenden Luciferase-Untereinheiten zur vollständigen Luciferase vereinen können.
Wie man das Biolumineszenz-Imaging verwenden kann, um biologische Prozesse in Versuchstieren über Wochen zu beobachten, zeigt Kathryn L. Pepples Gruppe an der University of Washington in Seattle (Sci Rep. 10: 11377). Sie untersuchte, wie sich unterschiedliche Immunzelltypen bei einer lokalen Infektion verhalten. Die dafür nötigen vier Mauslinien, die Luciferase von Neutrophil-, Myeloid-, T- oder B-Zell-spezifischen Promotern exprimierten, erhielt die Gruppe durch Kreuzung auf Basis des Cre/Lox-Systems. Die Luciferase brachte eine Mauslinie mit, die vor dem ROSA26-Luciferase-Transgen ein gefloxtes Transkriptions-Stoppcodon trug. Das Substrat Luciferin injizierten die Forscher in die Bauchhöhle der rekombinierten Tiere. Nach entsprechender Verletzung konnten sie verfolgen, wie sich die biolumineszierenden Immunzellen bei einer Regenbogenhautentzündung um das betroffene Auge konzentrierten, nicht aber um das unbehandelte Auge.
Auch die Krebsforschung könnte von Biolumineszenz-basierten Systemen profitieren, etwa bei Zytotoxizitäts-Tests in Zellkulturen. Forscher versuchten schon vor einigen Jahren, Luciferasen in diesen Tests einzusetzen – die damals verwendete Fluc war aber nicht stabil genug. Neuentdeckte Luciferasen aus Meerestieren wie Ruderfußkrebsen erzeugen jedoch nicht nur helleres Licht als Fluc, mit einer Halbwertszeit von mehreren Tagen sind sie auch ungleich stabiler. Darüber hinaus sind sie kleiner (ca. 20 kDa gegenüber 61 kDa) und obendrein ATP-unabhängig.
Freigelassene Luciferase
Preet M. Chaudharys Team von der University of Southern California, Los Angeles, konzentrierte sich für einen Zytotoxizitäts-Assay zunächst auf die aus dem Ruderfußkrebs Gaussia princeps stammende Luciferase (Gluc), die im natürlichen Umfeld sekretiert wird. Die US-amerikanischen Forscher vermuteten deshalb, dass Gluc in Zellen gefangen bleibt, wenn die Signalsequenz fehlt. Sobald die Zellen jedoch durch cytotoxische Substanzen angegriffen werden und die Membran durchlässig wird, müsste Gluc im Zellüberstand auftauchen.
Um diese Hypothese zu überprüfen, klonierte die Gruppe eine Gluc-Variante ohne Signalsequenz in verschiedene Expressionsvektoren und transfizierte mit diesen die Krebszelllinie 293FT. Anschließend behandelten Chaudharys Mitarbeiter die Zellen mit einer tödlichen Dosis Digitonin und führten mit dem Kulturüberstand einen Biolumineszenz-Assay durch. Tatsächlich war die Lumineszenz in diesem bis zu 5.000-mal höher als im Überstand der unbehandelten Kontrollen. Ermutigt von diesem Ergebnis klonierten die Kalifornier ein ganzes Potpourrie an Luciferasen aus verschiedenen Meerestieren in die Expressionsvektoren und führten mit diesen den gleichen Versuch durch. Auch mit diesen Luciferasen konnten die Forscher die zunehmende Biolumineszenz im Überstand nachweisen, wenngleich der Anstieg nicht so stark war wie mit der Gaussia-Luciferase. In Reminiszenz an den populären Strand El Matador State Beach an der kalifornischen Pazifik-Küste in Malibu nannten die Forscher ihren Zytotoxizitäts-Test Matador-Assay (Sci. Rep. 8:199).
Matador-Assay für CAR-T-Zellen
Den Matador-Assay könnte man zum Beispiel im Verlauf der CAR-T-Zell-Therapie einsetzen. Bei dieser werden T-Zellen aus Patientenblut gewonnen und ex vivo mit chimären Antigen-Rezeptoren (CAR) ausgestattet, die Tumorzellen erkennen. Die expandierten CAR-T-Zellen werden den Patienten während einer Chemotherapie wieder zurückgegeben und sollen die Tumorzellen attackieren und ausschalten. Mit dem Matador-Assay könnte man CAR-T-Zellen auf ihre Wirksamkeit hin überprüfen. Dabei nutzt man einen wesentlichen Vorteil des Assays aus: Die Signale können nur von den attackierten Zielzellen stammen, nicht aber von den T-Zellen. Viele übliche Zytotoxizitäts-Assays können nicht unterscheiden, ob der gefundene Marker für den Zelltod, etwa ein endogenes Protein oder Enzym, aus den Zielzellen oder den Effektorzellen stammt.
Die CAR-T-Zell-Therapie ist eine Gratwanderung. Die Rezeptoren auf den T-Zellen müssen stark genug an Epitope auf den zu tötenden Zellen binden, dürfen aber keine anderen Körperzellen angreifen. Der ebenfalls von Chaudharys Gruppe ausgearbeitete Malibu-Assay könnte das Screening von Kandidaten vereinfachen und bei der Suche nach perfekten Antikörper-Molekülen helfen (Sci. Rep. 10: 2318). Hierfür wird ein aufs Minimum reduzierter Antikörper, der zum Beispiel gegen das von B-Zellen exprimierte Oberflächenantigen CD19 gerichtet ist, mit einer Luciferase verknüpft. Die Luciferase (Nluc) dieses sogenannten Malibu-Glo-Reagenz stammt aus der Tiefseegarnele Oplophorus gracilorostris und enthält im Gegensatz zur Gluc des Matador-Assays das Sekretionssignal.
Wie gut der jeweilige Antikörper exprimiert wird, ist an der Biolumineszenz von 293F-Zellen zu erkennen, die mit dem Konstrukt transfiziert werden. Um die Bindeaffinität des Antikörpers beurteilen zu können, inkubiert man die Zielzellen 45 Minuten lang in einem Nluc-Überstand und misst nach einem kurzen Waschschritt und der Zugabe des Substrats Coelenterazin die an die Zellen gebundene Lumineszenz.
Mit dem Topanga-Assay der Kalifornier, der ebenfalls nach einem Strand benannt ist, kann man schließlich untersuchen, wie sich Variationen der Zielzellen auswirken, wenn die T-Zellen unverändert bleiben (Sci. Rep. 9: 1957). Dazu exprimiert man eine Auswahl potenzieller Antigene, beziehungsweise deren extrazelluläre Domänen, als Fusion mit einer Luciferase. Durch das beibehaltene Sekretionssignal gelangt das Fusionsprotein in den Kulturüberstand, der daraufhin mit CAR-T-Zellen inkubiert wird. Innerhalb einer halben Stunde kann man auf diese Weise Antigene aufspüren, die gut an die CAR-T-Zellen binden. Diese könnte man wiederum als Basis für die Entwicklung verbesserter Rezeptoren einsetzen.
Enges Wellenlängen-Fenster
Mit Chlorophyll fangen Pflanzen Sonnenenergie für die Photosynthese ein. Diese Fähigkeit steht Biolumineszenz-Anwendungen in Pflanzen jedoch im Weg, weil die Lichtsignale innnerhalb des Spektrums verschluckt werden, in denen Chlorophyll absorbiert – also zwischen 400 und 500 sowie 600 bis 700 Nanometern. Oft ist es für das Substrat auch schwierig, bis in das Gewebe vorzudringen, in dem die Luciferase exprimiert wird. Angesichts des happigen Preises von Luciferin und seiner toxischen Wirkung ist eine großzügige Dosierung keine Option.
Einen möglichen Weg aus diesem Dilemma fand ein großes internationales Team unter Federführung von Ilia V. Yampolsky vom Shemyakin-Ovchinnikov Institute of Bioorganic Chemistry in Moskau, dem neben russischen und brasilianischen Wissenschaftlern auch Fyodor Kondrashovs Gruppe vom IST-Austria in Klosterneuburg angehörte. Die Forscher entdeckten 2018 eine völlig neue, zu bisherigen Enzymen nicht homologe Luciferase (nnLuz) in dem brasilianischen Giftpilz Neonothopanus nambi.
Der Pilz stellt sein Leuchtsubstrat über den Kaffeesäure-Stoffwechselweg her. Hefezellen, die nnLuz und drei Enzyme des Substrat-Stoffwechselwegs exprimierten, leuchteten in Kaffeesäure-haltigem Medium. Mäuseembryonen, in die nnLuz-mRNA injiziert wurde, zeigten ebenfalls eine Lumineszenz, wenn sie mit dem passenden Luciferin versorgt wurden (PNAS 115(50): 12728-32).
Kaffeesäure (3,4-Dihydroxyzimtsäure), die Analog-Fotografen übrigens für Entwicklerflüssigkeit verwenden, entsteht über den Phenylpropanoidweg und ist der häufigste in pflanzlicher Nahrung vorkommende sekundäre Pflanzenstoff. Diese Schnittstelle von Pilz und Pflanze machte sich nicht nur die russische Gruppe zunutze, sondern auch eine US-amerikanische von der University of Minnesota mit Daniel Voytas als Seniorautor (Nature Biotechnol. 38: 944-46; Elife 9: e52786).
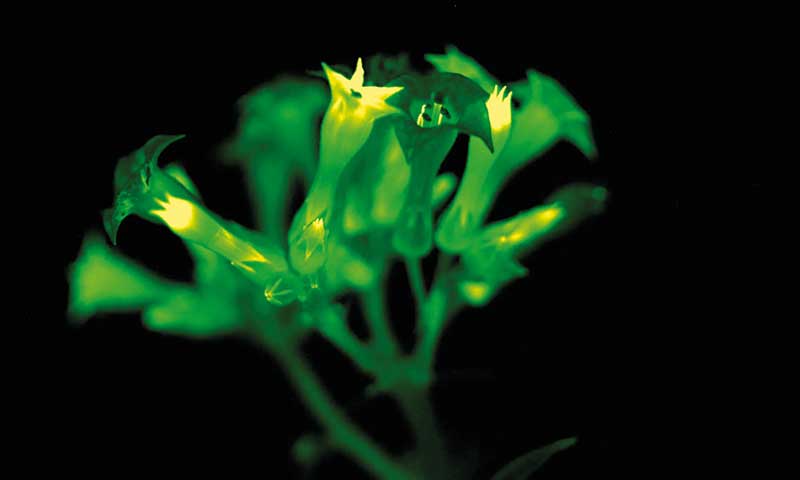
Die Forscher manipulierten den pflanzlichen Stoffwechsel mit vier Pilzgenen. Ziel war, das Biolumineszenz-Substrat ausgehend von endogener Kaffeesäure in Pflanzen entstehen zu lassen und mit der exprimierten nnLuz umzusetzen. Die Strategie ging tatsächlich auf. Transient mit Agrobacterium tumefaciens transformierte oder transgene Tabakpflanzen, die das Biolumineszenz-System exprimierten, emittierten Licht mit einer Wellenlänge von circa 525 Nanometern, das perfekt in die Lücke zwischen den Chlorophyll-Absorptionsspektren passte. Regionen, in denen viel Kaffeesäure vorliegt, zum Beispiel Wurzelspitzen, leuchteten heller. Auch Bedingungen, die die Kaffeesäureproduktion ankurbeln, verstärkten die Lumineszenz – etwa Hormonbehandlung, Trockenstress, Verwundung oder ein zusätzliches Synthese-Gen. Der Trick mit dem Substrat-Recycling in Leuchtbakterien funktioniert auch hier: Caffeylpyruvat-Hydrolase überführt das verbrauchte Leuchtsubstrat wieder in Kaffeesäure.
Während sich die russisch-österreichische Gruppe die Mechanismen des Biolumineszenz-Systems genauer anschaute, brachten die US-Kollegen transient transformierte Dahlien, Petunien und Rosen sowie Tomatenpflanzen zum Leuchten. Das System eignet sich offensichtlich für verschiedenste Pflanzenspezies und auch als Reporter für Genexpressions-Studien. Setzten die Forscher die Biolumineszenz-Gene beispielsweise hinter einen tagsüber aktiven diurnalen Promoter, leuchteten die Pflanzen täglich zum Beginn der Nacht besonders stark.
Louisa Gonzalez-Somermeyer, Doktorandin in Fyodor Kondrashovs Labor, hat am IST-Austria die molekularbiologischen Vorarbeiten zu dem Pilzsystem geleistet und bei einem Forschungsaufenthalt in Moskau die ersten Pflanzenexperimente durchgeführt. Ihr ehemaliger Kollege Karen Sarkisyan, der an der Entdeckung des Systems wesentlich beteiligt war, leitet inzwischen am Imperial College in London sein eigenes Labor. Zudem gründete er in Moskau das Start-up Planta, das mit der Pilzlumineszenz leuchtende Zierpflanzen herstellen will.
Gonzalez-Somermeyer berichtet, dass Pflanzen mit konstitutivem Promoter ihre volle Leuchtkraft jeweils im Tagesverlauf entwickeln. Stellt man sie ins Dunkle, behalten sie den circadianen Rhythmus zumindest für einen weiteren Tag bei. Sie könnte sich vorstellen, dass Pflanzenarten mit hohem Kaffeesäure-Spiegel besonderes Leuchtpotenzial haben. Die IST-Forscherin hält das neue Biolumineszenz-System aufgrund der hohen Sensitivität für besonders geeignet, um physiologische Veränderungen oder Variationen in der Genexpression in Echtzeit zu verfolgen. Nicht auf schädigendes Fluoreszenzlicht und Substratzugabe angewiesen zu sein und Pflanzen intakt lassen zu können, sei ein wesentlicher Vorteil. Über gerichtete Evolution oder Metabolic Engineering ließe sich das pilzliche Biolumineszenz-System vermutlich noch besser auf Gegebenheiten in Pflanzenzellen anpassen. Eine weitere Optimierungsstrategie wäre ihrer Ansicht nach, die Verfügbarkeit der Kaffeesäure zu erhöhen.
Gonzalez-Somermeyers Forschung an dem neuen Biolumineszenz-System geht jedenfalls weiter, wenn auch nicht am IST-Austria, sondern bei Planta in Russland.
Da Kaffeesäure im tierischen Stoffwechsel nicht vorkommt, ist das neue Biolumineszenz-System noch den Pflanzenforschern vorbehalten. Vorerst. Es fehlen nur zwei zusätzliche Enzyme, damit Tierzellen Tyrosin in Kaffeesäure umwandeln können. Mit einem euphorischen „Yes!“, bestätigt Gonzalez-Somermeyer, dass HEK293T-Zellen, die neben dem eigentlichen Gen-Repertoire mit einem Gen für die Produktion von Tyrosin-Ammonium-Lyase und Komponenten der 4-Hydroxyphenylacetate-3-Monooxygenase ausgestattet wurden, das erhoffte Lichtsignal abgaben.
Letzte Änderungen: 08.12.2020