Neue Protein-Labelling-Techniken
Proteine etikettieren mit Klick-Chemie
Henrik Müller
Proteine sind unsichtbar. Versteckt unter ihresgleichen, können molekular-, zell- und strukturbiologische Techniken nicht viel mit ihnen anfangen. Es sei denn, spezifische biochemische Etiketten holen die Proteine
aus ihrer Anonymität. Synthetische Biologen haben dafür ganz neue Möglichkeiten entwickelt.
„Für das Protein-Labelling hat sich über die letzten zehn Jahren in den Life Sciences eine komplett neue Chemie durchgesetzt“, erklärt Edward Lemke, biophysikalischer Chemiker und außerordentlicher Professor am Mainzer Institut für Molekulare Biologie.
Die Entwicklung der Labelling-Techniken verlief jedoch nicht geradlinig. Ziel erster selektiver Markierungsmethoden waren exponierte natürliche Aminosäurereste, wie Cystein-Seitenketten. Neben Cystein wurden vor allem die Seitenketten exponierter Lysine und aromatischer Aminosäuren ins Visier genommen (Chem. Soc. Rev. 45: 1691-1719).
So häufig diese Reste auf der Proteinoberfläche anzutreffen sind, so wenig erfüllen sie die Ansprüche von Biowissenschaftlern an ortsspezifische Markierungen. Denn ihre Erwartungen an perfekte Protein-Label sind hoch: Sie sollten an beliebige Positionen in Proteinen binden, egal ob pro- oder eukaryotisch, und gleichzeitig spezifisch für eine benutzerdefinierte Position sein – den Zellhintergrund dürfen sie jedoch nicht markieren. Zudem sollten die Markierungsmethoden schnell und quantitativ sein, ohne toxische Reagenzien zu benötigen oder zu erzeugen.
Lemke erläutert: „Ein Zellbiologe fasst konventionelle Labelling-Methoden meist nicht an. Denn er benötigt Techniken, die der Zelle keine Probleme bereiten.“
In diese nicht-invasive Zukunft weisen bioorthogonale Markierungsmethoden, die seit einigen Jahren en vogue sind. Bioorthogonal sind zum Beispiel künstliche chemische Reaktionen, die in Zellen stattfinden, ohne zelluläre Prozesse im Geringsten zu beeinträchtigen. Weder beeinflussen die Biomoleküle in der Zelle die Reaktion, noch löst die Reaktion selbst toxische Nebenwirkungen in der Zelle aus.
Der Inbegriff für Bioorthogonalität sind Reaktionen aus der Klick-Chemie, die mit hohen Ausbeuten sowie äußerst spezifisch ablaufen, und dabei mit Geschwindigkeitskonstanten von bis zu 106 M-1 s-1 fast Enzyme einholen. Da bioorthogonale Klick-Reaktionen die Abläufe in Zellen überhaupt nicht stören, sind sie perfekt für das Markieren von Proteinen geeignet. Populär sind die Bildung von Heterozyklen aus Carbonylen sowie Cycloadditionen zwischen Alkinen und Aziden. Azido-Gruppen sind dabei besonders vielfältig. Sie dienen oft direkt als Photo-Cross-Linker oder Infrarot-Sonde und können leicht zu Isopeptidbindungen, EPR- oder FRET-Sonden umgebaut werden.
Lemke ergänzt: „Was orthogonal ist, sehen Biologen anders als Chemiker. In vivo bleiben meist nur zwei Klick-Chemien übrig, da sie so toxisch für die Zelle sind wie Gummibärchen für den Menschen: Die Bertozzi-Chemie, also der gespannte Achtring mit Dreifachbindung plus Azid. Und die inverse Diels-Alder-Chemie. In der Regel ebenfalls ein Achtring mit trans-Doppelbindung plus Tetrazin. Alle Wissenschaftler, die im seitenspezifischen Labelling aktiv sind, arbeiten mit Bertozzi- oder inverser Diels-Alder-Chemie. Und da Diels-Alder eine Größenordnung schneller sein kann, kommt sie immer häufiger zum Einsatz. Ohne sie würde modernes Fluoreszenz-Labelling nicht funktionieren. Die Technik ist bahnbrechend.“
Kein Wunder, dass die Klick-Chemie in den letzten zehn Jahren zu einer riesigen Spielwiese für synthetische Biologen wurde. Lemke resümiert: „Ihre Magie ist, dass wir zwei Sachen so zusammenklicken können wie Legosteine, egal welche funktionellen Gruppen links und rechts dranhängen.“
Den Biophysikern um Heinz-Jürgen Steinhoff von der Universität Osnabrück missfiel beispielsweise, dass Cystein-Seitenketten paramagnetische NMR- und EPR-Experimente dirigieren. Zur Ortsmarkierung mit Nitroxid-Radikalen musste bisher Cystein durch Mutagenese eingebaut und dann in vitro mit Cystein-spezifischen Spin-Labeln modifiziert werden. Proteine mit nativen Cysteinen und In-vivo-Anwendungen schloss das meist aus.
Steinhoffs Mitarbeiter verglichen daher Klick-Reaktionen von Azid- und Alkin-funktionalisierten Nitroxiden mit zyklischen und linearen Lysin- und Phenylalanin-Derivaten. Ein waches Auge richteten sie auf die Internalisierung und Toxizität der Spin-Label in lebenden E. coli-Zellen. Am Ende markierten sie in vitro bis zu hundert Prozent ihrer Substrate binnen dreißig Minuten. In vivo machten ihnen jedoch gedrittelte Ausbeuten und die Redox-Sensitivität von Nitroxiden zu schaffen. Dennoch lädt ihre In-vivo-Machbarkeitsstudie zu leichteren Abstandsmessungen in Biomolekülen per NMR und EPR ein (J. Magn. Reson. 275: 38-45).
Modifizierte Aminosäuren
Für die Ortsmarkierung mit posttranslationalen Modifikationen (PTM) ist Dehydro-Alanin eine wertvolle Vorstufe. Viele bisherige Labelling-Verfahren beruhen auf kovalenten Bindungen zu Heteroatomen, taugen also nicht zum Aufbau des C-C-Gerüsts von Aminosäuren. Die Peptid-Chemiker um Ben Davis an der University of Oxford nutzten sp3-hybridisierte Kohlenstoffatome zur chemischen Mutagenese. Mit Halogenalkanen erzeugten sie relativ stabile Cα-Radikale von Dehydro-Alanin und bauten diese in alle möglichen Aminosäurereste ein: In fluorinierte, N- und O-glycosylierte, phosphorylierte, alkylierte und Isotopen-markierte sowie natürliche und unnatürliche Aminosäuren (Science 354: 6312). Noch mangelt es dem Reaktionsmechanismus aber an stereochemischer Kontrolle: Er erzeugt nicht nur L-Aminosäuren, sondern auch ihre Spiegelbildisomere.
Inzwischen existieren dank Klick-Chemie biochemische Klebeetiketten in allen erdenklichen Varianten: Mit photostabilen Fluoreszenz-Sonden können Forscher die zelluläre Lokalisation und Interaktion von Proteinen verfolgen – nicht nur in der höchstauflösenden Mikroskopie. Chemische Cross-Linker erlauben die Herstellung von Biosensoren, Protein-Mikroarrays und funktionalisierten Nanopartikeln. Gleichzeitig quervernetzen sie Proteine und ermöglichen so die strukturelle Massenspektrometrie. Synthetische PTMs erleichtern den Erkenntnisgewinn in Glykomik und Lipidomik. Ohne Isotopen-markierte Verbindungen wären NMR und EPR undenkbar. Polymere und cytotoxische Funktionsgruppen verbessern die Pharmakokinetik und Wirksamkeit therapeutischer Proteine und Antikörper-Wirkstoff-Konjugate. Ein Mangel an Protein-Labels herrscht offenbar nicht.
Vor ihrer Modifizierung müssen Aminosäuren oder ihre Vorstufen aber zunächst in das Peptidrückgrat integriert werden. Der Einbau erfolgt entweder durch die Erweiterung des genetischen Codes oder durch enzymatische Biokonjugation. Vor der Entwicklung von Code-Erweiterungen machte sich der gewitzte Forscher die Schlampigkeit mancher Aminoacyl-tRNA-Synthetasen (aaRS) zunutze, die auch nicht-kanonische Aminosäuren erkennen. Konkurrenz lassen sich die nativen Aminosäure-Substrate der aaRS aber nicht gerne bieten. Will der Bioforscher keine heterogene Mischung mit unmarkierten Proteinen riskieren, muss er auxotrophe Zell-Linien mit Strukturanaloga der Mangel-Aminosäure füttern. Das schränkt die einbaubaren Funktionsgruppen natürlich ein. Für die Montage, etwa von Seleno-Methionin, zur Phasierung kristallographischer Daten reicht es aber allemal aus.
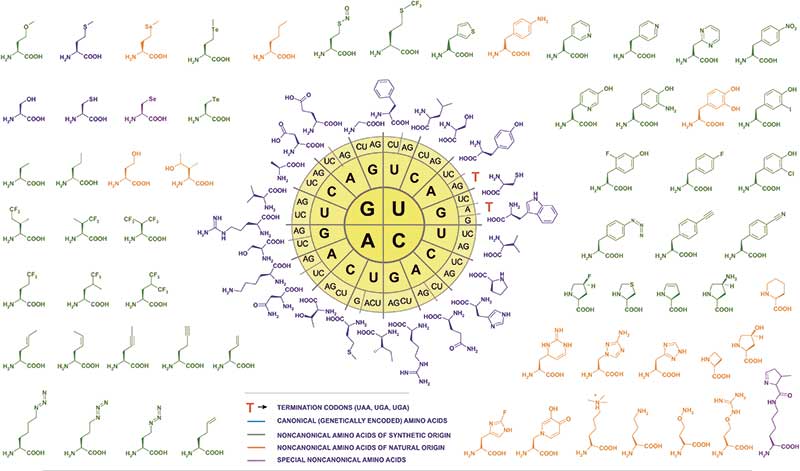
Eleganter ist der weiterentwickelte Ansatz, in dem meist ein Amber-Stopp-Codon als Sense-Codon uminterpretiert wird. Seit der Veröffentlichung vor 18 Jahren werden nicht-kanonische Aminosäuren fast ausschließlich mit der Amber-Suppression an gewünschten Positionen eingebaut. Die Methode nutzt ein bioorthogonales tRNA/aaRS-Paar, das ein UAG-Stopp-Codon ignoriert und in eine unnatürliche Aminosäure translatiert. Hierfür wird zum Beispiel in E. coli das Tyr-tRNA/TyrRS-Paar von Methanococcus jannaschii exprimiert, während E. colis Tyr-tRNA/TyrRS-Paar in Eukaryoten zum Einsatz kommt.
Amber-Suppresion
Zur ortsgerichteten Mutagenese eines Proteins muss, abgesehen vom tRNA/aaRS-Plasmid, nur die auserwählte DNA- oder mRNA-Position gegen das Amber-Codon ausgetauscht werden. Lemke dämpft jedoch übertriebene Erwartungen in Säugerzellen: „Dort gewinnt häufig die native Terminations-Maschinerie der Translation. Die besten mir bekannten Ausbeuten liegen bei fünfzig Prozent Unterdrückung eines Stopp-Codons. In der Regel sind es eher zwanzig Prozent“.
Lemkes Doktorand Christopher Reinkemeier benennt den Flaschenhals: „Wir kennen nur drei orthogonale tRNA-Synthetasen, die in Eukaryoten wirklich gut funktionieren. Am beliebtesten ist die Pyrrolysyl-tRNA-Synthetase aus dem Archaeon Methanosarcina mazei. Sie ist einzigartig, denn Säugerzellen verwenden kein Pyrrolysyl. Die Synthetase sieht für sie somit komplett fremd aus.“ Womit er die allem zugrundeliegende Schwierigkeit andeutet, weitere orthogonale tRNA/aaRS-Paare zu identifizieren. Lemke dazu: „Dummerweise ist dank der Evolution alles verwandt. Da im Säuger schon zwanzig tRNA-Synthetase-Paare rumschwimmen, ist es wie ein 3D-Puzzle, ein evolutiv entferntes Paar finden zu wollen, das keine Kreuz-Reaktivität mit diesen zeigt. Die Natur wollte halt nie mehr als 22 Aminosäuren codieren. Deshalb wird eher künstliche Proteinevolution zusätzliche Spezifität schaffen.“
Die Erweiterung des genetischen Codes erlaubt es inzwischen, die natürlichen Modifikationen Phospho-Serin, Acetyl-Lysin, Phospho- und sulfoniertes Tyrosin einzubauen. Die Palette unnatürlicher Bausteine dagegen reicht mit über zweihundert Aminosäuren von Fluoreszenz-Reportern über Photo-CrossLinker und 13C/15N/19F-NMR-Sonden bis zu bioorthogonalen Reaktionszentren. Es verwundert somit nicht, dass die Amber-Suppression mittlerweile nicht nur in Bakterien und Hefen eingesetzt wird, sondern auch in multizellulären Organismen wie C. elegans, D. melanogaster und A. thaliana.
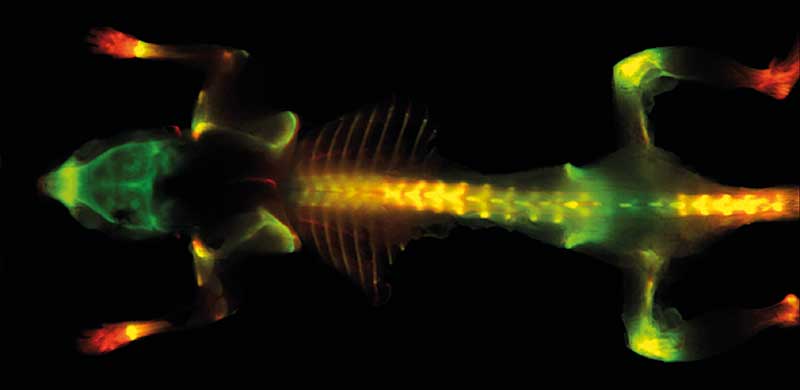
Die Molekularbiologen um Jason Chin vom MRC Laboratory of Molecular Biology der Universität Cambridge, UK, erweiterten die Technik schließlich auf Vertebraten. Sie injizierten auf adenoviralen Vektoren codiertes Grünfluoreszierendes Protein (GFP) mit einem Amber-Codon an Position 150 sowie Amber-spezifische Pyrrolysyl-tRNA/aaRS, in den Hypothalamus lebender Mäuse. Nach zwei Wochen beobachteten sie eine intracraniale GFP-Fluoreszenz, wenn sie nicht-kanonisches Lysin mithilfe einer osmotischen Mini-Pumpe durch das Mäusehirn strömen ließen oder es den Mäusen im Trinkwasser verabreichten (Nat. Chem. Biol. 12(10): 776-78).
Ausgereizte Code Expansion
Das Protein-Labelling ist buchstäblich in lebenden Säugern angekommen. Die Erweiterung des genetischen Codes von Mus musculus um NεAcetyl-Lysin durch transgene Integration eines entsprechenden tRNA/aaRS-Paars in das Mausgenom sorgte 2017 für noch mehr Aufsehen (Nat. Commun. 8: 14568).
Über die vielen Anwendungsmöglichkeiten der Genetic Code Expansion informiert ein Review von Heinz Neumann, Heisenberg-Fellow und Gruppenleiter für Angewandte Synthetische Biologie am Max-Planck-Institut für Molekulare Physiologie in Dortmund, und seiner Postdoktorandin Petra-Gisela Neumann-Staubitz (Curr. Opin. Struct. Biol. 38: 119-28). Neumann fasst zusammen: „Die Genetic Code Expansion ist nahezu ausgereizt. Jetzt müssen auf Seiten der Fluorophore mehr Anwendungsmöglichkeiten in Zellen entstehen.“
Bei drei Stopp-Codons wäre es natürlich Verschwendung, nur eines für die Erweiterung des genetischen Codes einzusetzen. Mit Plasmiden zur dualen Suppression können zusätzlich Ochre- oder Opal-Codons genutzt werden, um eine zweite bioorthogonale Aminosäure in ein Polypeptid einzuführen. Neumann warnt aber: „Mit jeder zusätzlichen Komponente wird es kombinatorisch komplexer, da alles mit allem orthogonal sein muss. Solch ein Vielkomponenten-System drückt die Proteinausbeute.“
Für FRET-basierte Anwendungen in Säugerzellen reicht es aber bereits aus. Über augenblickliche Herausforderungen multipler Suppression informiert ein Review von Sviatlana Smolskaya und Yaroslav Andreev von der Sechenov First Moscow State Medical University (Biomolecules 9: 255).
Neumann und Chin kreierten eine Erweiterung der Amber-Suppression in Form einer Translations-Maschinerie, die Quadruplett-Codons erkennt. Im Reagenzröhrchen evolvierten sie an der Translations-Terminierung beteiligte 16S-ribosomale-RNA. Ihre Suche nach Mutationen, die die Amber-Suppression begünstigen, führte sie zum Quadruplett-decodierenden E. coli-Ribosom RiboQ1 (Nat. Chem. 6(5): 393-403). Quadruplett-Genome geben Wissenschaftlern bis zu 256 freie Codons an die Hand, um bioorthogonale Funktionalitäten einzuführen.
Neumann relativiert aber: „Die ribosomale Einbaueffizienz ist der Flaschenhals. Denn Quadruplett-lesende-tRNAs stehen mit den endogenen Triplett-tRNAs in Konkurrenz. Oft wird auch die richtige Aminosäure eingebaut, das Leseraster aber nicht entsprechend verschoben. Ein weiteres Hindernis ist der Mangel an Stämmen, die orthogonale Ribosomen stabil im Genom integriert haben. Aktuell sehe ich deshalb nur begrenzte Anwendungen und biologische Fragestellungen für das System.“
Vielleicht werden ja nicht-kanonische Nukleotide oder eine Entfernung von Stopp-Codons im Zuge einer synthetischen Neucodierung ganzer bakterieller Genome diese Limits aufheben. Zumal Kosten von bis zu 35 Millionen Euro für Organismen wie Mycoplasma laboratorium, die durch komplett synthetische Genome kontrolliert werden, kein neues Limit darstellen.
Lemke kritisiert jedoch die Konzeption der gängigen Methoden: „Da wir jedes Stopp-Codon in der Zelle unterdrücken, erzeugen wir eine Menge ungewollt verlängerter Proteine. Und das stresst natürlich die zelluläre Physiologie. Für Zwecke reiner Überexpression in E.coli funktioniert das alles sehr gut. In der Zellbiologie finden orthogonale tRNA/aaRS-Systeme aber kaum Anwendung.“
Synthetisches Organell
Erklärtes Ziel von Lemkes Gruppe ist es daher, Proteine nicht-invasiv mit einem von ihnen entwickelten synthetischen Organell zu labeln (Science 363: 6434). Christopher Reinkemeier, Erstautor der Arbeit erklärt: „Wir minimieren die Amber-Unterdrückung fremder Proteine, indem wir ein orthogonales tRNA/aaRS-Paar in einem Designer-Organell vom Cytoplasma ausschließen. Das synthetische Organell erzeugen wir durch Phasenseparation und binden sie zusätzlich an Elemente des Cytoskeletts. Dann rekrutieren wir nur die gewünschte mRNA über spezifische RNA-Sekundärstrukturen zum Organell. Dank dieser doppelten Aufkonzentrierung plus Zugangsbeschränkung unterdrücken wir ganz selektiv nur das Amber-Codon unseres Zielproteins. Jetzt hoffen wir, in nächster Zeit ein Paper nachschieben zu können, in dem die Fluoreszenz zu sehen ist – mit hoher Auflösung, ohne jeglichen Hintergrund und im besten Fall in einer nachweislich glücklichen Zelle. In ein paar Jahren soll niemand mehr klobige GFPs benutzen müssen, sondern seitenspezifische Mini-Farbstoffe bevorzugen.“
Das zweite Verfahren, bioorthogonale Reaktionszentren in Proteinen aufzubauen, instrumentalisiert Enzyme. Ihre Regio- und Stereoselektivität macht sie für die Biokonjugation an kleine Moleküle und Polymere besonders attraktiv, vom In-vivo-Labelling bis zur Immobilisierung an Oberflächen. Außerdem offerieren Enzyme den direktesten Weg, ein Amber-supprimiertes Protein ein weiteres Mal zu modifizieren. Einziger Wermutstropfen, abgesehen von der notwendigen Co-Expression: Ihre Erkennungssequenzen müssen rekombinant an das Zielprotein fusioniert werden. Die enzymatische Biokonjugation ist somit auf N- und C-Termini und Schleifen flexibler Oberflächenreste begrenzt. Ein Review der Biochemiker um Mark Distefano von der University of Minnesota fasst die gängigsten Peptidasen, Transferasen, Ligasen und Oxidoreduktasen ausführlich zusammen (Chem. Soc. Rev. 47(24): 9106-36).
Eines der neuesten enzymatischen Verfahren entwickelte die Tubulis GmbH, eine Firmenausgründung der LMU München und des Leibniz-Forschungsinstituts für Molekulare Pharmakologie in Berlin. Tubulis‘ Forschungsleiter (CSO) Jonas Helma-Smets erklärt: „Mit der Tub-tag-Technologie machen wir die Tubulin-Tyrosin-Ligase (TTL), ein Enzym des Tubulin-Codes der Cytoskelett-Homöostase, erstmalig biotechnologisch nutzbar. Sie tyrosiniert den C-Terminus eines Proteins. Doch ihre 14-Aminosäuren-Erkennungssequenz ist zugleich ein Alleinstellungsmerkmal. Mit zwei Dritteln polaren Aminosäuren, hauptsächlich Glutamate, ist der Tub-tag extrem hydrophil und schafft so einen Puffereffekt gegen die Hydrophobizität vieler Wirkstoffmoleküle.“
Damit spielt Helma-Smets auf Tubulis‘ Entwicklungsfokus an. Chemotherapeutische Toxine sind meist hydrophob und verringern die Fähigkeit von Antikörper-Wirkstoff-Konjugaten, Krebszellen zu binden. „Außerdem akzeptiert die TTL eine ganze Reihe unnatürlicher Tyrosin-Derivate, die nachfolgend chemo-selektiv weiterkonjugiert werden können.“ Dazu zählen neben 3-Fluoro-Tyrosin zur 19F-NMR-Spektroskopie auch biotinylierte Aromaten und fluoreszierende Konstrukte wie Cumarin-funktionalisiertes Glycin. TTL weckt also nicht nur im krebstherapeutischen Biotech-Markt Interesse, sondern auch in der Einzelmolekül-Mikroskopie und -spektroskopie, die durch die Größe der eingesetzten Detektions-Moleküle limitiert sind.
Die Transpeptidase-Varianten und Homologen der Sortase A sind weitere Labelling-Werkzeuge, mit denen sich Biopolymere aber nicht nur markieren lassen: Mit ihnen kann man diese auch zyklisieren oder Proteindomänen selektiv mit NMR-tauglichen Isotopen versehen.
Ernsthafte Konkurrenz erhalten sie gegenwärtig von einer Endopeptidase aus dem Schmetterlingsblütler Clitoria ternatea namens Butelase 1. Im Gegensatz zu Sortasen benötigt sie nur eine kurze Erkennungssequenz von drei Aminosäureresten, hinterlässt also nur eine minimale Narbe im ligierten Polypeptid. Ihre außergewöhnliche Promiskuität erlaubt es sogar, Peptide aus D-Aminosäuren zu zyklisieren. Mit einer katalytischen Effizienz von bis zu 500.000 M-1 s-1 läuft sie Sortasen obendrein den Rang der schnellsten Peptid-Ligase ab. Bisher musste Butelase aufwendig aus C. ternatea-Samen gereinigt werden, weshalb sie sich nur zur Modifizierung der E. coli-Oberfläche mit Biotin- und Fluoreszenz-Sonden verdingen konnte. Im Juni 2019 beschrieben westaustralische Botaniker jedoch die Expression einer rekombinanten Butelase 1, welche die Endopeptidase in einer Funktionsverlust-Mutante von Arabidopsis thaliana erfolgreich ersetzte (Plant J. 98(6): 988-99).
Split-Inteine mit Turbo
Unterschiedliche Markierungsmuster benachbarter Peptidsequenzen erlauben auch intervenierende Proteine, sogenannte Inteine. In einem autokatalytischen Prozess schneiden sich diese aus einem Polypeptid heraus, die rechts und links verbleibenden Exteine fügen sie hierbei zusammen (Protein-Trans-Splicing). Im Fall von Split-Inteinen stammen die Extein-Fragmente von separat translatierten Proteinen. Manche Inteine, wie Gyrase A und die Split-Intein-Familie DnaE, sind auch zwanzig Jahre nach ihrer Entdeckung noch für Überraschungen gut. So entwickelte Tom Muirs Gruppe am Princeton Chemistry Laboratory, die DnaE-Variante Cfa mit „Beschleunigungs-Resten“, die wie ein Turbo für das Protein-Trans-Splicing funktionieren. Cfa ist aber nicht nur deutlich schneller als andere Inteine sondern auch noch bei 80 °C sowie in Gegenwart von 4 M Guanidiniumchlorid und 8 M Harnstoff aktiv (J. Am. Chem. Soc. 138(7): 2162-65). Das Protein-Splicing ist so nur noch eine Frage weniger Minuten.
Trotz der Vielfalt an Markierungsmethoden gibt es selten einen Königsweg. Falls die Mängel der oben genannten Techniken das eigene Forschungsprojekt zu sehr einschränken oder die Methoden zu aufwendig sind, lohnt sich vielleicht ein Blick auf alternative Techniken: Etwa die Festphasensynthese von Polypeptiden, die kürzer sind als fünfzig Aminosäuren, Fusions-Tags wie SNAP, CLIP und Halo, sequenzspezifische Chelatoren wie FlAsH-EDT2 und His-Tag-Sonden sowie konventionelle Antikörper, Nanobodies und autofluoreszierende Polypeptide.
Klar, auch diese Verfahren haben Nachteile, wie zum Beispiel die Reversibilität nicht-kovalenter Bindungen, ungenügende Ortsspezifität, räumlicher Abstand zum gewünschten Markierungsort oder eine Größenzunahme von dutzenden Kilodalton. In einem sind sich die Methodenentwickler jedoch sicher: Die zukünftige Arena aller Labelling-Verfahren sind lebende Zellen.
Letzte Änderungen: 10.10.2019