Lichtscheiben-Mikroskopie aufgeblähter Gehirne
Expansionsmikroskopie
Frederique Wieters
Mit aufwendigen höchstauflösenden Mikroskopen können Biowissenschaftler inzwischen Strukturen unterhalb der Auflösungsgrenze abbilden. Aber wieso den Spieß nicht einfach umdrehen und die Probe vergrößern, statt immer kompliziertere und teurere Mikroskope zu entwickeln?
Die Mikroskopie ist eines der wichtigsten Werkzeuge der biowissenschaftlichen Forschung, das aus keinem Labor mehr wegzudenken ist. Aufgrund des Abbe-Limits ist die Auflösung konventioneller Lichtmikroskope aber auf etwa 200 Nanometer begrenzt.
Um auch Strukturmerkmale jenseits der Auflösungsgrenze herkömmlicher Lichtmikroskope beobachten zu können, entwickelte der Physiker Stefan Hell die Stimulated Emission Depletion (STED)-Mikroskopie, für die er 2014 den Chemie-Nobelpreis erhielt. STED gehört zusammen mit der Structured Illumination (SIM)- sowie der Stochastic Optical Reconstruction-Mikroskopie (STORM), für die der US-Amerikaner Eric Betzig vom Janelia Research Campus in Ashburn, USA, ebenfalls den Nobelpreis erhielt, zu den sogenannten Superresolution-Mikroskopie-Verfahren (SRM).
Diese Methoden erreichen zum Teil eine Auflösung von wenigen Nanometern, die vorher nur mit der Elektronenmikroskopie (EM) möglich war. Bei der EM wird die hohe Auflösung jedoch mit einer komplizierten Vorbereitung der Proben und einem hohen apparativen Aufwand erkauft.
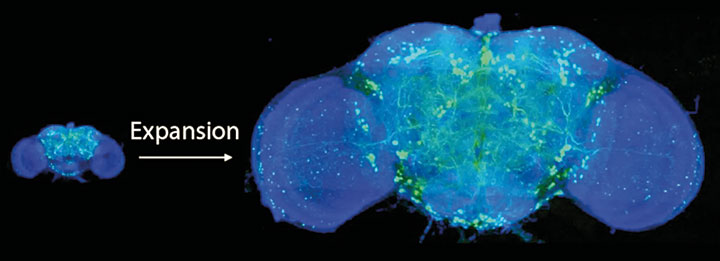
Was aber wäre, wenn man die zu untersuchende Probe, zum Beispiel Gehirnschnitte oder sogar ein ganzes Gehirn, einfach vergrößern und sie dann mit der Lichtmikroskopie betrachten würde? Das ist die Grundidee der von Edward Boydens Gruppe am Massachusetts Institute of Technology (MIT) entwickelten Expansionsmikroskopie (Science 347(6221):543-8).
Smarte Expansions-Gele
Bei der Expansionsmikroskopie (ExM) wird die Probe in Natriumacrylat und Acrylamid getränkt, die sich nach der Zugabe eines Polymerisations-Triggers miteinander vernetzen. Die Gel-Technik geht auf die Arbeit von Toyoichi Tanakas Gruppe am MIT zurück, die ab Mitte der siebziger Jahre synthetische Gele mit „smarten“ Eigenschaften entwickelte. In den achtziger Jahren adaptierten Christine Dreyer und Peter Hausen vom Max-Planck-Institut für Virusforschung in Tübingen diese Polyacrylamid-Hydrogele, um Proben besser färben und mikroskopieren zu können.
Die Expansionsmikroskopie folgt dem Gedanken, dass sich die biologische Information durch die isotrope, gleichmäßige Vergrößerung der Untersuchungs-Objekte nicht verändert. Im ersten Schritt werden die Biomoleküle in den Proben mit molekularen Ankern versehen, an denen sich die aufquellenden Hydrogele festhalten können. Anschließend taucht man die Probe in eine Monomer-Lösung, die mit einem Crosslinker vernetzt wird, wodurch die Anker an das entstehende Polymer-Netz binden. Danach homogenisiert man die vernetzte Probe (Hitze und Detergenzien oder enzymatischer Verdau) und wässert sie. Durch osmotische Kräfte dringt das Wasser in das Hydrogel ein, wodurch dieses aufquillt und zur Expansion der Probe führt (Nature Methods 16, 33-4).
Die behandelten Gewebeproben können sich bei diesem Prozess um das Hundertfache ihres Volumens beziehungsweise das Viereinhalbfache ihrer Größe in alle Raumrichtungen ausdehnen. Reguliert wird die Stärke der Expansion durch die Konzentration der zugegebenen Polymerisations-Trigger. Mit einer viereinhalbfachen linearen Expansion und einem Mikroskop mit einer Auflösung von 200 Nanometern erreicht man eine Auflösung von circa 45 Nanometern (~200 nm/4,5 = ~45 nm).
Mehrfach aufblähen
Es ist aber auch möglich, zwei oder sogar drei Expansionen hintereinander durchzuführen, wodurch sich die Auflösung weiter verbessert (Details zu den Expansionsprotokollen finden Sie unter expansionmicroscopy.org). Bei der ExM bleiben Antigene nach Ausdehnung der Polymere erhalten und können auch nachträglich mit Farbstoff-markierten Antikörpern angefärbt werden. Aufgrund der Expansion mit den wasserabsorbierenden Polymeren ist die Methode jedoch nicht für lebende Zellen geeignet.
Aber liefert die Expansionsmikroskopie auch tatsächlich originalgetreue Bilder der Zellstrukturen? Dieser Frage ging der Mikroskopie-Experte Markus Sauer vom Biozentrum der Julius-Maximilians-Universität Würzburg (JMU) zusammen mit Ed Boyden sowie Forschern aus Lausanne und Genf nach. Bislang vertraten viele Wissenschaftler die Meinung, dass die Expansion der Polymere ungleichmäßig verläuft und zu einer verzerrten Darstellung führt.
Keine Verzerrung der Realität
Das Forscherteam um Sauer zeigte jedoch am Beispiel von Zentriolen, dass die Ultrastruktur-Expansionsmikroskopie (U-ExM) tatsächlich die Realität abbildet (Nat. Methods 16(1):71-4). Die vom Biologen Theodor Boveri 1888 erstmals beschriebenen Zentriolen spielen eine entscheidende Rolle bei der Zellteilung. Das Team wählte sie für die U-ExM-Experimente, weil ihre Strukturen durch elektronenmikroskopische Aufnahmen gut beschrieben sind.
Die Gruppe konnte nachweisen, dass es bei der U-ExM zu keiner unregelmäßigen, anisotropen Verschiebung der Zentriolen oder anderer Zellkompartimente kommt, wenn man diese mit Paraformaldehyd oder Glutaraldehyd fixiert.
ExM-Proben bestehen zu einem Großteil aus eingelagertem Wasser und sind transparent. Sie sind daher auch für die Lichtblatt- oder Lichtscheiben-Mikroskopie (LSM) geeignet. Bei der LSM wird die Probe nicht punktuell beleuchtet, sondern durch eine Lichtscheibe, die mehrere Fluorophore gleichzeitig anregt, wodurch größere Gewebe schnell und Zell-schonend mikroskopiert werden können. Um eine transparente Probe zu erhalten, wird die LSM meist mit sogenannten Clearing-Methoden (CLARITY, CUBIC oder iDISCO) kombiniert, mit denen Lipide entfernt werden. Das hat den Vorteil, dass die Probe nicht kryostatisch geschnitten werden muss, sondern in einem Stück mikroskopiert werden kann was die Bildung von Artefakten beziehungsweise Veränderungen anatomischer Strukturen minimiert.
Perfekt ergänzt mit Lichtscheiben-Mikroskopie
Ein Team um Ed Boyden und Eric Betzig kombinierte die ExM mit der von Betzig entwickelten Lattice Light Sheet-Mikroskopie (LLSM) und visualisierte mit der neuen ExLLSM-Technik komplette Drosophila-Gehirne oder den Kortex von Maus-Gehirnen (Science 363, 6424, eaau8302). Die Gruppe untersuchte mit ExLLSM unter anderem dendritische Dornfortsätze (Spines), die Distanzen zwischen den prä- und postsynaptischen Proteinen Bassoon und Homer1 sowie Zellschicht-spezifische Variationen der axonalen Myelinisierung. Da die Gewebe als Ganzes visualisiert wurden, konnten die Forscher zum Beispiel alle dopaminergen Neuronen eines Drosophila-Gehirns und deren Verbindungen zwischen den einzelnen Hirnregionen darstellen.
Die ExLLSM kombiniert die hohe Geschwindigkeit der LSM mit der Nanometer-genauen Auflösung der ExM. Hinzu kommt, dass man Proteine mit spezifischen Fluoreszenzfarbstoffen exakt markieren kann. Die Kombination aus Expansions- und Lichtscheiben-Mikroskopie füllt damit die Lücke zwischen konventionellen optischen Methoden und der Elektronenmikroskopie.
Betzig und Boyden gehen davon aus, dass mit einer zehnfachen Expansion schon bald Protein-spezifische Analysen von gesamten Gehirnen, mit einer Auflösung von 25 Nanometern oder besser möglich sind. Die ExLLSM-Technik wäre dann auch dazu geeignet, die neuronale Entwicklung oder die Korrelation zwischen Verhalten und neuronaler Aktivität auf Nanometer-Ebene zu beobachten.
Letzte Änderungen: 08.05.2019