Funktionelle Genetik im XXL-Format
Spezial: High-Content-Screening und -Analyse
Karin Hollricher
Die automatisierte, mikroskopische Analyse zellulärer Phänotypen im Hochdurchsatz, das High-Content-Screening, verzeichnet Erfolge. Eine Sammlung positiver Nachrichten.
Zikaviren verursachen schwerste neurologische Schäden. Es gibt jedoch derzeit kein Medikament gegen die Infektion. Aber man darf hoffen: die Viren haben offensichtlich Achillesfersen. Die kennen Forscher zwar noch nicht, doch sie identifizierten Moleküle, die die Vermehrung der gefährlichen Viren unterbinden. Eines verhindert Zika-Infektionen nicht nur in aus Stammzellen differenzierten neuronalen Zellen und Organoiden, sondern auch in den Gehirnen von Mäusen (Cell Stem Cell 21: 274). Obwohl das Molekül aus der wunderschönen, aber sehr giftigen rosenroten Spinnenlilie Lycoris radiata stammt, ist es für den Menschen nicht toxisch und von der FDA bereits als pharmakologische Substanz zugelassen.
Gefunden haben die Forscher das Molekül mit einer Methode namens High-Content-Screening (HCS); mitunter firmiert sie auch unter HCA (High-Content-Analyse) oder HCI (High-Content-Imaging). High-Content-Screenings haben sich in den letzten fünf Jahren vor allem in der Grundlagenforschung einige Freunde gemacht, weil man damit neue Genfunktionen entdecken und qualitativ, quantitativ und zeitlich aufgelöst beschreiben kann. In relativ kurzer Zeit lässt sich darstellen, wie Zellen unter dem Einfluss einer Behandlung phänotypisch reagieren, seien dies Medikamente, kleine Moleküle oder das An- oder Ausschalten von Genen.
Suche nach Phänotypen
Offensichtliche Phänotypen sind schon seit 150 Jahren ein wichtiges Werkzeug der klassischen Genetik: Im 19. Jahrhundert experimentierte Gregor Mendel, in völliger Unkenntnis von Genen und DNA, mit verschrumpelten, gelben Erbsen. Im frühen 20. Jahrhundert schloss Thomas Morgan Hunt aus seinen Arbeiten mit Drosophila, dass Phänotypen nicht nur vererbbar sondern auch an Chromosomen gekoppelt sind. Der Zusammenhang von Phänotypen und Genen, als funktionelle Genetik bezeichnet, bescherte der Biologie eine Fülle spannender Erkenntnisse. HCS ist funktionelle Genetik, nur eben im großen Stil.
Drei Dinge braucht man für ein High-Content-Screening:
- Verschiedene und in unterschiedlichen Farben fluoreszierende Moleküle, mit denen man gleichzeitig mehrere Zellbestandteile wie Kern, Membran, Golgi-Apparat, ER und Zellskelett markieren und sichtbar machen kann.
- Ein automatisiertes Mikroskop, das in Eigenregie 384-Well-Platten unter das Objektiv legt und von jeder Vertiefung ein Bild macht.
- Und schließlich eine ausgefuchste Software, die in diesen niedrig-aufgelösten Bildern zunächst einzelne Zellen und sogar Organellen beziehungsweise Kompartimente identifizieren und damit den zellulären Phänotyp kategorisieren kann.
Eine Roboter-gesteuerte Plattform, die die Vorbereitung der Zellen bewerkstelligt, ist nicht unbedingt nötig, vereinfacht aber die Arbeit und standardisiert das Verfahren.
Der Einsatz der Mikroskopie und die Abfrage vieler zellulärer Parameter unterscheidet das HCS vom Hochdurchsatz-Screening, welches typischerweise das Verhalten nur eines Moleküls, zum Beispiel eines Enzyms oder eines Rezeptors, in der Anwesenheit verschiedener Moleküle überprüft.
Automatisierte Screens
Das erste High-Content-Screening mit einer ersten Imaging-Plattform publizierten vor elf Jahren Kenneth Giuliano, Lansing Taylor und Kollegen (J. Biomol. Screen. 2: 249). Sie diagnostizierten, dass die Genomforschung zwar sehr viele Daten zur Verfügung stelle, aber die Analyse der biologischen Funktion eines Gens oder Moleküls sehr viel Zeit in Anspruch nehme und demzufolge ein Engpass in der Pharmaforschung sei. Als Lösung für dieses Problem schlugen sie vor, Zellen für automatisierte sekundäre Screens einzusetzen, weil diese als „Einheit des Lebens“ alle wichtigen Funktionen ausführten und man Veränderungen der Zellkomponenten bestimmen könne, selbst wenn man das Target nicht kenne.
„Das Konzept ist, jede Zelle wie ein Well zu behandeln, das räumliche und zeitliche Informationen über das Verhalten der markierten Bestandteile enthält“, schrieben die Forscher 1997. Und weiter: „...Automatisierung ist der Schlüssel zur Steigerung der Produktivität.“ Sie stellten zwei Konzeptstudien vor. Zum einen beobachteten sie das Überleben beziehungsweise die Apoptose von Zellen nach einer Behandlung mit dem Anti-Krebsmittel Paclitaxel. Zum anderen dokumentierten sie die Endozytose des Glucocorticoid-Rezeptors, nachdem er einen passenden Liganden gebunden hatte. Dafür benutzten sie jeweils nur zwei Farbstoffe, um die Phänotypen voneinander zu unterscheiden.
Zu selten Multi-Parametrisch
High-Content meint allerdings „viel Inhalt“. Es geht also um mehr als nur ein oder zwei Parameter. Doch die meisten Studien sind bisher eher einfach gestrickt, wie vor vier Jahren Shantana Singh und Kollegen berichteten, die für die gemeinsam vom Broad Institute und der Harvard University betriebenen Imaging Platform arbeiten. Sie schrieben: „Angesichts der Fähigkeit des HCS, multi-parametrische Readouts zu liefern, sind wir überrascht, dass 60 bis 80 Prozent der Artikel nur eine oder zwei Eigenschaften der Zellen berücksichtigt haben.“ Aber es gebe inzwischen Fortschritte, meint Michael Boutros vom DKFZ, dort Sprecher des Forschungsschwerpunkts funktionelle und strukturelle Genomforschung. Boutros und seine Mitarbeiter nutzen HCS, um Signalwege, Genfunktionen und zelluläre Netzwerke zu erfassen. Dazu testeten sie Kombinationen verschiedener RNAi-Moleküle an Drosophila-Zellen. Unter 70.000 RNAi-Paaren fanden sie über 600, die die Phänotypen der Zellen quantitativ beeinträchtigten (Nat. Methods 8: 341).
Außerdem interessierte Boutros‘ Team, welche Wirkungen eine Kombination mehrerer pharmakologischer Substanzen auf Krebszellen hat und inwieweit diese von der individuellen genetischen Ausstattung der Zelllinien abhängt. Dabei entdeckten die Forscher Überraschendes: Beispielsweise reagiert das Proteasom auf einen EGFR-Inhibitor und MEK-Inhibitoren verstärken den Effekt eines Medikaments, das Alkoholkranken helfen soll, abstinent zu bleiben (Mol. Syst. Biol. 11: 846). „Ich habe den Eindruck“, so Boutros, „dass die multi-parametrischen High-Content-Screenings zunehmen.“
Ein Beispiel – das Cell Painting – hier im Detail, weil es so schön ist. Letztes Jahr veröffentlichten Forscher ein Protokoll für ein HCS mit sechs fluoreszierenden Farbstoffen, womit sie acht zelluläre Organellen beziehungsweise Kompartimente markierten und morphologische Charakteristika wie die Größe, Form, Textur und Intensität der Färbungen nach der Inkubation mit verschiedenen kleinen Testmolekülen und/oder siRNA-Transfektion analysierten (Nat. Protoc. 11: 1757-74).
Das gesamte Prozedere machten sie mit Zelllinien, primären Zellen und einer Co-Kultur von primären Hepatozyten und Fibroblasten. Insgesamt untersuchten sie 13 verschiedene Kulturen in 384-Well-Platten mit schwarzem Boden (geringere Eigenfluoreszenz) mit einem Weitfeld-Mikroskop bei 20-facher Vergrößerung und auf fünf Kanälen (mit fünf Filtern). Zwar seien konfokale Mikroskope schneller und lieferten kontrastreichere Bilder. Doch sie seien teurer und hätten nur eine begrenzte Auswahl von Anregungswellenlängen, argumentierten die Forscher.
In den lebenden Zellen markierten sie zunächst die Mitochondrien mit MitoTracker DeepRed. Anschließend fixierten sie die Zellen mit Formaldehyd, machten die Membranen mit Triton X-100 durchlässig, um dann die Zellkerne mit Hoechst 33 342 und die Nucleoli sowie cytoplasmatische RNA mit grün fluoreszierendem SYTO14 zu färben. Das ER wurde mit ConcanavalinA, Golgi und Plasmamembran mit Weizenkeim-Agglutinin und das Zytoskelett mit Phalloidin markiert, wobei jedes der genannten Moleküle mit einem AlexaFluor-Farbstoff versehen war. Die Auswertung erfolgte mit der hauseigenen, Open-Source Software CellProfiler.
Schnelles Verfahren
Ein solches HCS ist ziemlich fix. „Die Zellkultur und die Bildaufnahme dauern zwei Wochen; die Extraktion und Analyse der Daten nochmals ein bis zwei Wochen“, heißt es in dem Artikel. Okay, da waren auch HCS-Experten am Werk, und zwar von der Imaging Platform in Cambridge (USA) und der in Salt Lake City ansässigen Firma Recursion Pharmaceuticals. Letztere stellt sich als „Truly Tech-First Life Sciences“ Firma vor, die sich auf Test-Design, paralleles Screening sowie Auswertung zellulärer Phänotypen spezialisiert hat.
Nachdem das Cell Painting so prima funktioniert hatte, testeten die Forscher vom Broad Institute dann auch gleich mal ein paar Substanzen. Sie dachten sich wohl, wenn schon, denn schon. Aus der Analyse von über 30.000 kleinen Molekülen resultierten 919.265 Aufnahmen aus verschiedenen Perspektiven, die man jetzt als Schwarz-Weiß-Bilder im TIF-Format in den öffentlichen Datenbanken „The Cell Image Library“ und „Image Data Resource“ finden kann. Die Arbeit wurde im letzten Jahr – wo sonst – in GigaScience (6: 1-5) publiziert.
Nachdem man eine große Auswahl an Fluoreszenzfarbstoffen und -markierungen sowie automatisierten Mikroskopie-Plattformen kaufen kann, hängt der erfolgreiche HCS-Einsatz von zwei Faktoren ab: Man muss einen richtig guten Test haben und eine mächtige Software für die Bildanalyse.
Eine für die Entwicklung neuer Analysen entscheidende Technologie ist CRISPR/Cas9. Mit dieser Technik lassen sich endogene Reporter zur gezielten Markierung von Proteinen recht einfach einbringen und somit neue Testmodelle entwickeln. „Dies war bisher über homologe Rekombination fast nur in embryonalen Stammzellen von Mäuse möglich, aber durch CRISPR/Cas9 lässt sich dies nun in fast allen Zellmodellen durchführen“, erklärt Boutros.
Automatisierte Bildauswertung
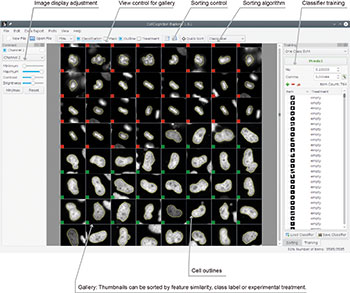
Der zweite Knackpunkt ist die automatisierte Bildauswertung und der Umgang mit sehr großen Datenmengen. Wir sprechen hier von etlichen Terabyte. Man kann Software zur Bildanalyse kaufen, es gibt aber auch Open Source-Programme. Was auch immer man benutzt, ein Bildverarbeitungsprogramm muss trainiert werden, um Phänotypen zu klassifizieren, die man bei dem jeweiligen Assay analysieren will. Prinzipiell gibt es dafür zwei Vorgehensweisen: Entweder trainiert der Benutzer sein Programm anhand von Bildern mit den möglichen und den gesuchten Phänotypen. Dieses so genannte Supervised Machine Learning nutzt man beispielsweise auch, wenn man sein iPhoto-Programm trainiert, bestimmte Personen auf Fotos zu erkennen.
Der zweite Weg zur Bildanalyse heißt Deep Learning. „Wenn man noch gar nicht weiß, wie die Zellen bei einem Screening reagieren werden, man also den Phänotyp nicht kennt und keine Trainingsbilder hat, kann man Deep Learning einsetzen“, sagt Daniel Gerlich vom Institut für Molekulare Biotechnologie in Wien. „Das ist eine statistische Methode, bei der die Software nach Andersartigen, nach ‚Outliern‘ sucht. Der Computer lernt also nicht, wonach ich suche, sondern wonach ich nicht suche. Er erlernt somit das Normale und identifiziert anhand dessen das Andere.“
Gerlich und seine Kollegen führten eine Konzeptstudie durch, mit der sie überprüfen wollten, ob und welche der beiden Verfahren, Supervised Machine Learning und Deep Learning, besser ist oder mehr Erkenntnis bringt (Mol.Biol. Cell. 28: 3428-36). Die Software CellCognition Explorer, die die Forscher auf der Basis vorhandener Programme entwickelten, ließen sie auf einen RNAi-Screen los, bei dem sie nach morphologischen Phänotypen des markierten Chromatin-Markers Histon2B suchten. Als Sparring-Partner stand das Programm CellCognition im Ring. Das Resultat: „Unser Deep Learning-Algorithmus fand viele Phänotypen, ohne dass wir diese vorher klassifizieren mussten. Im direkten Vergleich beider Verfahren sind wir zu ganz ähnlichen Ergebnissen gekommen. Welches besser ist, hängt von der Art des Screens ab und davon, was man schon weiß beziehungsweise herausfinden möchte“, so Gerlich.
Deep Learning mit Vorteil
Dass Deep Learning durchaus besser sein kann, zeigten Oren Kraus, Ben Grys und Kollegen von der University of Toronto (Mol. Syst. Biol. 13: 924). Sie verglichen den traditionellen Machine Learning-Ansatz ensLOC mit DeepLoc, einer auf Deep Learning basierten Software. Versuchskaninchen waren Hefezellen, die jeweils ein GFP-markiertes Protein enthielten. Insgesamt beherbergten die Hefen 15 verschiedene Proteine, in 15 verschiedenen Kompartimenten oder Umgebungen.
Die Programme sollten die Proteine aus den Bildern lokalisieren und den Ort zuordnen. Dabei zeigte sich, dass ensLOC mehr Fehler machte als das Deep Learning-Programm. So lokalisierte es 35 Prozent der Vakuolen-Proteine fälschlicherweise im Zellkern, DeepLoc nur knapp 20 Prozent. Für die Entwicklung neuer, leistungsstärkerer Programme sind Mathematiker und Informatiker gefragt, die im Kontext biologischer Fragestellungen denken. Die seien Mangelware, und somit werde die Datenanalyse auf längere Sicht ein Engpass beim Einsatz von HCS sein, denkt Marc Bickle, Leiter der Screening Facility am Max-Planck-Institut für Molekulare Zellbiologie und Genetik in Dresden.
Je komplizierter ein Screen ist, desto komplexer sind die Phänotypen und desto intelligenter müssen auch die Programme sein. Und die Phänotypen werden auf jeden Fall komplexer werden, denn nach der zweidimensionalen Zellkultur werden die verwendeten Untersuchungsobjekte nun dreidimensional. Gemeint sind Organoide, die beispielsweise aus Patientenzellen gezogen wurden, und ganze Tiere, vornehmlich Zebrafischlarven. Mithilfe Letzterer fand eine Gruppe Dresdner Forscher, zu denen auch Bickle gehörte, heraus, dass die als Anti-Epileptikum verwendete Valproinsäure eine Histon-Deacetylase inhibiert und dies die Vermehrung hämatopoetischer Stammzellen in vitro und in vivo aktiviert, was sich positiv auf die Überlebens- und Differenzierungsfähigkeit transplantierter Stammzellen auswirkt (Sci. Rep. 7: 12084). Bickle: „Ohne High-Content Screening hätten wir das vermutlich überhaupt nicht entdeckt.“ Möglicherweise wird sich Valproinsäure in Zukunft als sinnvolle Unterstützung bei Knochenmarktransplantationen oder Gentherapien erweisen.
Auch Organoide, die Schnittmengen von 2D-Zellkulturen und lebenden Tieren, sind bei weitem „lebensähnlicher“ als gewöhnliche Zellkulturen (siehe dazu das LJ-Editorial Vom Organmodell zum Organersatz). Gleichzeitig kann man auf Tierversuche verzichten, zunächst jedenfalls. „Die gleichzeitige Evolution von Organoid-Modellen und bildgebenden Technologien verändern die Lebenswissenschaften“, sind die Organoid-Spezialisten Anne Rios (Walter and Eliza Hall Institute, Melbourne, Australien) und Hans Clevers (Universität Utrecht, Niederlande) überzeugt und prophezeihen der Kombination eine „leuchtende Zukunft“ (Nat. Meth. 15: 24-6).
„High-Content Screening hat sich als sehr fruchtbar für die Grundlagenforschung entpuppt“, resümiert Boutros. Wie alle Grundlagenforscher schaut er erstmal auf alle Phänotypen und fragt sich: „Was ist eigentlich alles passiert?“ Dann macht er sich daran, neue Phänomene, Funktionen und Signalwege zu entdecken.
Reservierte Pharmaforschung
Was ist mit der Pharmaforschung? Die fokussiert sich branchenbedingt darauf, herauszufinden, ob und welche Moleküle Unterschiede zwischen Krank und Gesund verursachen beziehungsweise, aus Krank wieder Gesund machen. Ein solches Experiment, wenn es denn zellbasiert und phänotypisch ist, nennen Pharmaforscher dann folgerichtig Phenotypic Drug Discovery (PDD). Ist HCS, pardon PDD, in der Medikamentenforschung angekommen? Ein bisschen, wäre wohl die richtige Antwort. Einer Studie aus dem Jahr 2011 zufolge, wurden zwischen 1999 und 2008 fünfzig neue sogenannte First-in-Class-Moleküle in klinischen Tests evaluiert, von denen 28 aus phänotypischen Tests stammten, nur 17 aus so genannten Target-Based Screens (Nat. Drug. Discov. 10: 507-19). Unter Target-based Screen versteht man die Suche nach Molekülen, die ein potentielles, für eine Krankheit bedeutsames Protein beeinflussen. Dafür muss man den Kandidaten bereits kennen, also auch eine Hypothese haben. Dieses Vorgehen war insbesondere bei der Suche nach neuen Medikamenten gegen Krebs erfolgreich und führte zu verschiedenen Kinaseinhibitoren.
Im Gegensatz zu diesen Studien ist es für die zellbasierten, phänotypischen Hochdurchsatz-Analysen nicht nötig, bereits Kandidaten identifiziert zu haben. Es ist ein agnostischer, hypothesenfreier Ansatz, der kein spezielles Molekül im Visier hat. Das hat den Vorteil, dass man Substanzen mit so genannten new Mechanisms-of-Action finden kann. Der Pharmaforscher spricht von nMoA. Statt also beispielsweise gezielt einen weiteren Kinaseinhibitor oder Apoptose-Aktivator zu suchen, könnte man mehr über eine Krankheit, ihre zugrunde liegenden molekularen Prozesse und damit auch neue Ziele für neuartige Wirkstoffe entdecken, wenn man die vorhandenen chemischen Substanzbibliotheken auf ihre phänotypische Wirkung hin testet.
Bessere Ursachenbekämpfung
Nehmen wir zum Beispiel Arthritis. Diese Allerwelts-Erkrankung wird derzeit vor allem mit Schmerzmitteln in Schach gehalten. Es existiert jedoch kein pharmakologischer Wirkstoff, der das reduzierte Knorpelgewebe wieder aufbaut. Daher begaben sich Forscher der Novartis Research Foundation in San Diego auf die Suche nach einem Mittel, das tatsächlich die Ursache der Erkrankung beseitigen könnte. In einem phänotypischen Screen mit mesenchymalen Stammzellen, den Vorläufern der Knorpelzellen, war ein Molekül namens Kartogenin der Top-Hit. Es induzierte die Expression verschiedener typischer Knorpelzellen-Marker. Auch in Tiermodellen steigerte Kartogenin die Regeneration von Knorpelzellen (Science 336: 717-21). Die anschließenden zellbiologischen Untersuchungen enttarnten den nMoA des Kartogenins: Es inhibiert die Bindung des Aktin-bindenden Moleküls Filamin A an den Transkriptionsfaktor CBFß, sodass letzterer vom Zytosol in den Zellkern wandern kann und dort mithilfe des Faktors RUNX die Expression der knorpelspezifischen Gene ankurbelt.
Diese Studie ist kein HCS im eigentlichen Sinn, die Autoren nennen sie selber einen „Image-based High-Throughput Screen“. Denn die Forscher analysierten nur eine spezielle, für die Induktion der Chondrogenese typische Veränderung. Dies geschah allerdings im 384-Well-Format. Die Auswertung der Bilder war, auch wenn das nicht ganz genau aus der Beschreibung hervorgeht, vermutlich nicht automatisiert. Sie war auch sehr einfach, beruhte nämlich schlicht auf einer Farbreaktion. Nichtsdestotrotz geht aus der Studie hervor, dass phänotypische Analysen ganz neue Arten von Mechanismen und Zielmoleküle hervorbringen können.
Wie geht‘s weiter mit HCS?
Hochrangige Vertreter verschiedener Pharmafirmen sind sich einig, dass PDD beziehungsweise HCS eine echte Herausforderung für die Medikamentenentwicklung ist. Sie sei allerdings dem bewährten Target-based Screen, aus unternehmerischer Sicht heraus unterlegen. Es sei aber gleichzeitig auch eine wirksame Strategie, wenn es um Krankheiten gehe, über die man noch wenig wisse. Hier könne PDD/HCS „[...] wirklich neue Therapeutika für bisher unbefriedigte medizinische Bedürfnisse liefern“ (Nat. Rev. Drug. Disc. 16: 531-43).
Und nun? Fast jede neue Technologie durchläuft den so genannten Gartner Hype-Zyklus, der auf einem X-Y-Diagramm aussieht wie eine gedämpfte Schwingung.
Er besagt, dass erst einmal völlig überzogene Erwartungen an das Potenzial einer neuen Technologie entstehen, die dann rasant ins Tal der Enttäuschungen fallen. Über den dann wieder ansteigenden Pfad der Erleuchtung erreichen die Erwartungen schließlich ein Plateau der Produktivität: Jetzt sind Vor- und Nachteile einer Technologie allgemein bekannt und akzeptiert, die Technologie kann ihrem Potential entsprechend eingesetzt werden.
Nicht nur erneuerbare Energien und Augmented Reality, auch Gentherapie, Genomforschung und all die anderen Omiken, Krebsforschung, Drug Design und Screening, RNAi, CRISPR/Cas9 befinden sich in verschiedenen Abschnitten dieses Zyklus. Viele Medikamentenentwicklungen sitzen im Tal der Enttäuschung fest. Die Gentherapie, die nach dem Tod von Jessy Gelsinger tief in diesem Tal versackt war, ist dank RNAi und neuer viraler Vektoren jetzt auf dem Pfad der Erleuchtung. Die Genomik, die mit der Deklaration von Bill Clinton zum Humangenom einen sagenhaften Gipfel erklommen hatte, ist auf dem Plateau der Produktivität angekommen.
Auch dem High-Content Screening werden bei allen Lorbeeren, die es schon einheimsen konnte, die Tiefen des Gartner-Zyklus sicher nicht erspart bleiben. Aber die bisherigen Daten weisen darauf hin, dass diese Methode sicherlich produktiv eingesetzt werden kann, selbst in der Medikamentenforschung – wenn man die passenden Assays und die richtige Datenverarbeitung hat.
Letzte Änderungen: 04.02.2018