Scheibchenweise statt am Stück
LSFM in Zell- und Entwicklungsbiologie
Isabell Smyrek, Katharina Hötte, Frederic Strobl und Alexander Schmitz
Die Lichtscheibenfluoreszenz-Mikroskopie schont die Zellen und liefert hochaufgelöste Bilder. Ihre Vorteile kommen insbesondere bei Langzeitaufnahmen von lebenden Proben zum Tragen.
„Mehr Licht!“ – so lauteten angeblich die letzten Worte des größten deutschen Dichters und Denkers Johann Wolfgang Goethe an einen seiner treuen Diener. Im Hinblick auf die Fluoreszenzmikroskopie ist dies allerdings kein guter Gedanke. Statt die ganze Probe zu beleuchten und aufzunehmen, wie bei der klassischen Fluoreszenzmikroskopie, ist es für die Zellen schonender die Aufnahmen „scheibchenweise“ durchzuführen. An diesem Grundsatz orientiert sich die Lichtscheibenfluoreszenz-Mikroskopie (LSFM), die in der Sprache der Jedi-Ritter ausgedrückt, die "Macht der dunklen Seite" ausnutzt.
Nachdem Ernst Stelzer in seinem Essay in der letzten Laborjournal-Ausgabe (LJ 7-8, S. 46) die Theorie der LSFM beleuchtete, beschreiben Mitarbeiter seiner Gruppe am Buchmann Institut für Molekulare Lebenswissenschaften in Frankfurt, im Folgenden einige praktische Anwendungen.
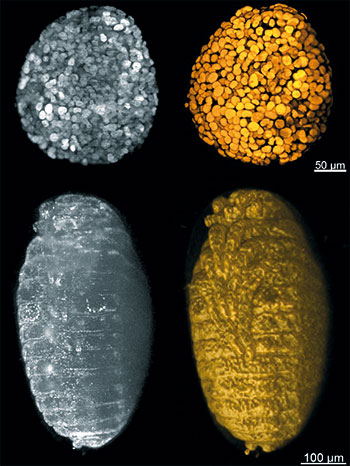
Die Stärken der noch jungen LSFM liegen vor allem in der Langzeitbeobachtung von dreidimensionalen (3D), lebenden Proben. So finden sich immer mehr Publikationen aus der Zell- und Entwicklungsbiologie, in denen die Forscher die Vorteile der LSFM nutzen. In der 3D-Zellbiologie verwendet man häufig Sphäroide (Aggregate interagierender Zellen) als Modell. Anders als Zysten, die in einer extrazellulären Matrix durch multiple Teilungen einer einzelnen Zelle ein dreidimensionales und innen meist hohles Gebilde erzeugen, werden Sphäroide von Anfang an aus mehreren Zellen geformt, die in einer nicht-adhäsiven Umgebung Zell-Zell-Kontakte ausbilden können.
Im Prinzip sind die meisten Zelltypen (zum Beispiel Primärzellen, Tumorzelllinien und „herkömmliche“ Zelllinien) zur Bildung von Sphäroiden in der Lage. In der Praxis existieren jedoch unterschiedliche Methoden zur Erzeugung von Sphäroiden. Bei der Tropfen-Methode („hanging drop“) pipettiert man zuvor vereinzelte Zellen in einem Tropfen Medium auf eine nicht-adhäsive Fläche, die man dann kopfüber im Inkubator platziert. Durch die Schwerkraft sammeln sich die Zellen am unteren Teil des Tropfens und bilden ein Aggregat. Eine andere Methode nutzt das Entstehen von Sphäroiden in nicht-adhäsiven Mikrotiterplatten mit U-förmigen Näpfchen. Die Zellsuspension wird hierzu in die Vertiefungen pipettiert („liquid overlay“), in denen die Zellen Sphäroide formen.
Zellforscher verwenden Sphäroide als Tumormodell. Sie finden aber auch immer mehr Anklang in der Wirkstoffforschung, da sie den physiologischen Zustand eher repräsentieren als ein zweidimensionaler „Zellrasen“, der auf einer harten Plastikoberfläche wächst.
Sanfter Blick ins Innere der Zelle
Für die Tumorforschung ist eine detaillierte Beobachtung von Sphäroiden unerlässlich. Die LSFM ist hierfür hervorragend geeignet, da sie Endpunktaufnahmen mit guter Qualität und hoher Eindringtiefe liefert, die einen Blick ins Innere der Sphäroide gestatten. Lebende Sphäroide lassen sich mit LSFM auch über längere Zeiträume hinweg beobachten. Hierbei störende Effekte wie Phototoxizität und das Bleichen der Fluorophore sind bei der LSFM auf ein Minimum reduziert.
Und wie bekommt man die Zellkugel beziehungsweise den Sphäroid in das LSFM? Hier bieten sich je nach Fragestellung verschiedene Arten der Präparation an. Bei der klassischen Methode präpariert der Experimentator die Probe in einer Agarosesäule, die er dann in die LSFM-Probenkammer einsetzt. Hierfür wird der Sphäroid zunächst in flüssige Agarose überführt (Achtung – nicht zu heiß!) und dann in eine Glaskapillare aufgezogen. Nachdem die Agarose ausgehärtet ist, schneidet man die Glaskapillare so zurecht, dass sie auf den Probenhalter im Mikroskop gesteckt werden kann. Ein dünner Stift an der Oberseite des Probenhalters wird in die Kapillare geführt und drückt den Teil der Agarosesäule, in dem sich der Sphäroid befindet, am anderen Ende heraus.
Die polymerisierte Agarose fungiert als engmaschiges Netz, das für Stabilität sorgt und den Sphäroid während der Aufnahme in seiner Position hält. Zusätzlich erlaubt die Agarose den Austausch von Gasen und Nährstoffen mit dem in der Probenkammer befindlichen Medium, um den Sphäroid bei Langzeit-Lebendaufnahmen optimal zu versorgen.
Kurzum, durch die reduzierte Phototoxizität, das verminderte Bleichen der Fluorophore, sowie die hohe Geschwindigkeit des Mikroskops ist die LSFM perfekt dazu geeignet, Sphäroide über einen längeren Zeitraum zu untersuchen.
Dreidimensionale Integrität bleibt erhalten
Auch Entwicklungsbiologen haben inzwischen die Vorteile der LSFM für sich entdeckt. Im Laufe des letzten Jahrzehnts etablierten sie mehrere Techniken, um Embryonen von Modellorganismen im LSFM zu untersuchen. Einer der wichtigsten Faktoren ist hierbei die Bewahrung der dreidimensionalen Integrität der Probe. Ähnlich wie bei Sphäroiden können Embryonen in Agarosesäulen eingebettet werden, was für einige Organismen (etwa Drosophila melanogaster) problemlos funktioniert.
Bei Tribolium castaneum, dem Rotbraunen Reismehlkäfer, verwenden die Forscher jedoch eine andere Technik: Der Embryo wird anterior, also mit der Vorderseite, auf die Spitze einer Agarose-Halbkugel „geklebt“. Mit PEI-Röhrchen ist es möglich, Zebrafisch-Embryonen in sehr niedrig konzentrierter Agarose einzubetten und dadurch den Einfluss auf das Größenwachstum zu minimieren. Auch Mäuseembryonen beobachteten Forscher schon mit dem LSFM, allerdings verwendeten sie hierfür einen Acrylstab mit kleinen „Taschen“, in denen die Embryos platziert waren.
All diese Techniken für die Aufnahme im LSFM haben eines gemeinsam: Im Gegensatz zur Anpassung der Probe an das Mikroskop in der konventionellen Mikroskopie geht man bei der LSFM den umgekehrten Weg und passt die Präparation und das Mikroskop an die Probe an.
Und wie bekommt man den Embryo zum „Leuchten“? Hier haben sich drei grundlegende Techniken durchgesetzt, die bei den meisten Modellorganismen der Entwicklungsbiologie geläufig und mit der LSFM kompatibel sind: (1) die Injektion/Applikation von fluoreszierenden Farbstoffen, (2) die Injektion/Applikation von mRNA, die für Fluoreszenzproteine kodiert, und (3) die Herstellung von transgenen Organismen, die Fluoreszenzproteine exprimieren.
Die ersten beiden Methoden lassen sich verhältnismäßig schnell umsetzen, erfordern aber eine Manipulation der Probe. Expressionsort und Zeitfenster kann man mit ihnen nur bedingt beeinflussen. Beim letztgenannten Verfahren muss man im Vorfeld mehr Zeit und Arbeit investieren. Dafür kann man die Probe ohne weitere Vorbereitung in das Mikroskop einsetzen und mit einem entsprechenden Promotor Expressionsort und Zeitfenster festlegen.
Bei der Arbeit mit lebenden, sich über mehrere Stunden, Tage oder sogar Wochen entwickelnden Proben ist es äußerst wichtig, den Einfluss der Beobachtungsmethode auf die Probe zu minimieren. Stringente Qualitätsstandards sind für die Experimente deshalb unabdingbar.
Bei der Aufnahme von Insektenembryonen wird der Embryo nach der Aufnahme unter physiologischen Bedingungen weiter inkubiert. Anschließend überprüft man, ob er schlüpft, sich zum gesunden adulten Tier entwickelt und Nachkommen zeugen kann. Nur so ist gewährleistet, dass durch den Energieeintrag während der Aufnahme keine Schäden am untersuchten Objekt hervorgerufen wurden und die Aufnahme die Wildtyp-Entwicklung abbildet.
Eine große Herausforderung ist die Visualisierung und Verarbeitung der erhaltenen multi-dimensionalen LSFM-Daten. Pro Aufnahme kommen schnell mehrere 100 GigaByte bis einige TeraByte zusammen. Aus der Beobachtung der Embryogenese von Tribolium castaneum über einen Zeitraum von fünf Tagen resultieren zum Beispiel ca. 1 TeraByte Bilddaten.
Der Experimentator muss deshalb einige Punkte beachten: (1) Aufgrund der Menge lassen sich die Daten nur begrenzt über längere Zeiträume abspeichern. (2) Komplexität und Umfang der Daten machen aus der Analyse eines untersuchten Prozesses das sprichwörtliche Suchen nach der „Stecknadel im Heuhaufen“. (3) Die Leistung vieler Rechensysteme reicht häufig nicht aus, um eine Verarbeitung in einem vernünftigen zeitlichen Rahmen zu ermöglichen. (4) Für die Extraktion der relevanten Information ist eine Kette („pipeline“) spezialisierter Programme nötig. Die qualitative und quantitative Auswertung der Daten ist deshalb sehr komplex und zeitaufwendig. Schnelle, automatisierte Verarbeitungsabläufe und Programme sind für die LSFM daher essentiell.
Und was macht man mit den Daten? Schön, dass es hier auch gute Nachrichten gibt! Die Entwicklung leistungsfähiger Datenverarbeitungs-Systeme schreitet stetig voran. Schnelle, mehrkernige Prozessoren, große Arbeits- und Festplattenspeicher, flotte Flashspeicher und die Möglichkeit, auf parallel arbeitenden Grafikkarten zu rechnen, gehören heute zum Standard vieler Arbeitsgruppen. Nur so ist die Verarbeitung sehr großer (Tera-Byte)Datensätze möglich. Zudem existieren häufig arbeitsgruppenübergreifende Computer-Cluster, die deutlich leichter zu nutzen sind als noch vor einigen Jahren.
Für die Visualisierung kann der Anwender auf verschiedene kommerzielle, hochspezialisierte Programme zurückgreifen (zum Beispiel Amira, Arivis oder Imaris), die es erlauben, Terabyte-große 3D-Datensätze als Funktion der Zeit darzustellen beziehungsweise in ein Bild zu übersetzen (Rendering). Hierdurch ist ein fundierter Einblick in die Daten möglich, um sie auch für Nichtexperten verständlich aufbereiten zu können. Mittlerweile existieren auch viele frei zugänglicher Programme für die Bildverarbeitung. Hierzu zählen die „Open Source“ Programme Fiji, Icy, Cell Profiler oder Ilastik. Diese bieten Basis-Funktionen, aber auch spezialisierte Plugins für einige der gängigsten Verarbeitungsschritte an.
Umfangreiche Datenanalyse
Soll die Bildverarbeitung in eine Analyse-Pipeline eingebettet werden, bedarf es jedoch meist umfangreicherer Programme. Hier bieten kommerzielle Plattformen wie Mathematica oder Matlab neben zahlreichen Werkzeugen für die Bildverarbeitung auch Funktionen für die weiterführende statistische Auswertung und Analyse.
Die Qualität der LSFM-Aufnahmen ermöglicht eine umfangreiche quantitative Datenanalyse. So lässt sich zum Beispiel eine Segmentierung auf zellulärer und subzellulärer Ebene durchführen. Anschließend können Merkmale von Zellen und Organellen extrahiert werden. Dank der hohen zeitlichen Auflösung ist es möglich Zellen und Strukturen über längere Zeiträume zu verfolgen („tracking“) und zelluläre Stammbäume („lineage“) zu rekonstruieren.
Allerdings erfordert dies auch eine stetige Neu- und Weiterentwicklung der Algorithmen und Bildverarbeitungs-Programme. Aus diesen Anforderungen ist ein völlig neues Arbeitsfeld namens „Bioimage Informatics“ entstanden. Fachleute aus der Informatik, Bioinformatik, Mathematik, aber auch anderen Disziplinen spezialisieren sich darin auf die Verarbeitung und Auswertung biologischer Bilddaten.
Die genannten Beispiele geben einen kleinen Einblick in die Möglichkeiten der LSFM in der Zell- und Entwicklungsbiologie. Die LSFM ist noch sehr jung und die Forscher beginnen erst, ihr Potential voll auszuschöpfen. Wir dürfen uns also in den nächsten Jahren auf weitere spannende Entwicklungen in der Lichtscheibenfluoreszenz-Mikroskopie freuen.
Letzte Änderungen: 02.09.2015