Ärger mit schrägen Basen
Vervielfältigung von NGS-Bibliotheken ohne PCR
Thorsten Lieke
Bei der Next-Generation-Sequenzierung gibt es verschiedene Fehlerquellen. Eine davon ist die Amplifikation der NGS-Bibliotheken mittels PCR.
Moderne Next Generation Sequenzier-Geräte arbeiten entweder mit Halbleiter-Techniken oder wie das System der Firma Illumina mit Mikroplatten auf denen die zu analysierende DNA in winzigen Poren fixiert ist. Unter jeder Pore befindet sich ein Messdetektor, der den Einbau der fluoreszierenden Nukleotide in Echtzeit misst.
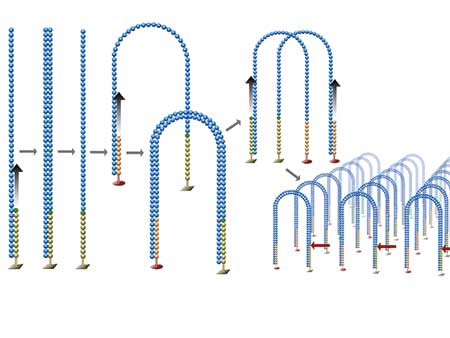
Für die Illumina-Sequenzierung wird die DNA-Vorlage zunächst so fragmentiert, dass auf einem Strang des Bruchstücks ein Adenin überhängt. An die überhängenden Enden ligiert man Adapter-Oligos mit einem Thymidin-Überhang, die die Annealing-Sequenzen der Sequenzier-Primer enthalten. Dies wird so lange durchgeführt, bis die DNA-Fragmente an beiden Enden mit Adaptern versehen sind. Mit einigen wenigen PCR-Zyklen vervielfältigt man anschließend die DNA-Fragmente und verlängert die Adapter-Enden, damit diese an Oligonukleotide auf der Trägerplatte binden können. Durch die Bindung der beiden Adapter mit dem dazwischen eingebundenen Template entsteht eine DNA-Brücke, die genau die Messpore überspannt.
Die anschließend mit Hilfe der DNA-Brücken durchgeführte Sequenzierung basiert im Grunde noch immer auf der Kettenabbruchreaktion von Sanger. Auf dem Träger befinden sich jedoch Myriaden Poren, in denen der Einbau von Nukleotiden in den komplementären Strang parallel erfolgt. Die Methode ist deshalb äußerst schnell und produziert riesige Mengen Sequenzdaten. Wie auch die anderen Varianten der NGS-Sequenzierung hat sie jedoch ein Problem mit dem so genannten Basen-Bias: PCR-Polymerasen produzieren häufig Artefakte, wenn die DNA-Fragmente sehr hohe GC- oder AT-Gehalte aufweisen. Diese führen schließlich zu Sequenzierfehlern.
Fast nur AT
Ein typisches Beispiel für eine sehr einseitige Basenaufteilung ist Plasmodium falciparum, der Erreger der Malaria tropica. Das Plasmodium-Genom besteht in den kodierenden Regionen zu 75% aus Adenin und Thymidin, in den nichtkodierenden Abschnitten steigt der Anteil auf bis zu 100%. AT-Paarungen sind thermolabil, so dass Polymerasen Abschnitte mit hohen AT-Anteilen oftmals nicht vervielfältigen.
Garbage in, garbage out
Bei der PCR-Amplifikation im Vorfeld der Sequenzierung entsteht dadurch ein Ungleichgewicht: Abschnitte, die von der Polymerase leicht prozessiert werden, liegen überproportional oft vor, während AT-reiche Regionen kaum amplifiziert werden. Das verzerrt die Sequenzierergebnisse und macht Aussagen über das Plasmodium-Genom nahezu unmöglich. Ähnliches gilt auch für Genome mit sehr hohen Guanin und Cytosin-Anteilen. Hier ist es jedoch die hohe Stabilität der Paarung, die die Amplifikation mit der PCR erschwert.
Forscher versuchen deshalb mit verschiedenen Techniken die Artefakte bei der PCR zu minimieren oder am besten ganz auf die PCR zu verzichten. So stellte Daniel Turner bereits 2009 (Kozarewa et al., Nature Methods, Vol. 6, 291-5) eine Methode vor, mit der sich die Sequenzier-Genauigkeit bei der Illumina-Sequenzierung verbessern lässt. Anstatt die Verbindungsabschnitte der Adapter durch die PCR herzustellen, verwendete Turner Adapter, die diese Verbindungen bereits enthielten. Dadurch kann man auf den Amplifizierungsschritt vor der Sequenzierung verzichten. Allerdings benötigt man eine ausreichende Ausgangsmenge genomischer DNA, da ja auch die Amplifizierung entfällt.
Im Fall der Plasmodien bringt Turners Methode nicht viel, da man nur eine sehr geringe Menge genomischer DNA aus den Parasiten isolieren kann. Die Vermehrung der Template-DNA mittels PCR ist hier obligatorisch. Turner ließ jedoch nicht locker. Mittlerweile hat sein Team gleich zwei Wege gefunden, mit denen man die AT-dominierten Sequenzen von Plasmodien ohne viele Fehler für die NGS-Sequenierung vorbereiten kann (Oyola et al., BMC Genomics, 2012, 13:1).
Spezielle Polymerasen
Der erste nutzt eine modifizierte PCR, bei der Tetramethylammoniumchlorid (TMAC) zugesetzt wird, das die Hitzestabilität der AT-reichen Abschnitte erhöht und dadurch die Arbeit der Polymerase erleichtert. Das Enzym muss jedoch mit Bedacht gewählt werden, da zum Beispiel die normalerweise verwendete Phusion-Polymerase in Anwesenheit von TMAC den Dienst quittiert. Polymerasen, die auch in Gegenwart von TMAC zuverlässig arbeiten sind zum Beispiel Kappa HiFi und T7. Turners Gruppe gelang es mit dem TMAC-Trick das Plasmodium Genom weitgehend fehlerfrei zu sequenzieren. Noch besser funktioniert ein PCR-freies System, das auf der sogenannten Recombinase Polymerase Amplification (RPA) beruht. Diese Technik wurde von Niall Armes‘ Gruppe in Cambridge entwickelt und wird mittlerweile von dessen Firma TwistDx vermarktet (Piepenburg et al., 2006, PLoS Biol, Vol.4, 1115-21).
Bei der RPA-Methode bindet man Primer an ein Rekombinase-Enzym und erzeugt so Filamente von aneinander geketteten Primer-Rekombinase-Komplexen. Diese Komplexe suchen die Template-DNA nach homologen Abschnitten für die Primer ab. Binden diese, so spalten sie den Doppelstrang auf und machen den Weg für die ebenfalls im System vorhandene Polymerase frei. Da hierbei das Denaturieren entfällt, kommt es zu keinem Basen-Bias in Regionen mit hohem AT-Anteil.
Turners Ziel ist es jedoch, komplett auf einen Amplifizierungsschritt zu verzichten. Mittlerweile ist er aus der akademischen Wissenschaft am Wellcome Trust Sanger Institute in Oxford ausgeschieden und zur Firma Oxford Nanopores gewechselt, die an einem Nanoporen-Sequenzierer arbeitet. Mit diesem wäre das Problem der PCR-Amplifikation bei NGS-Sequenzierungen mit einem Schlag gelöst – falls das Gerät tatsächlich noch den schon mehrfach angekündigten Weg auf den Markt findet.
Nachtrag
Jos de Graaf, vom Mainzer TRON-Institut, wies uns auf zwei Unstimmigkeiten im Neulich an der Bench-Artikel „Ärger mit schrägen Basen“ hin (LJ 7-8/2013). Illumina-Sequenzierer arbeiten nicht mit Mikroplatten sondern mit planaren „Flow Cells“. Die Adenin-Überhänge erreicht man nicht durch die Fragmentierung der DNA-Vorlage, sondern durch die Reparatur der entstandenen Fragmente.
Letzte Änderungen: 29.08.2013