Geht's noch?
Zielgerichtete Mutagenese (I)
Gabriele Grabowski
Will der Forscher wissen, wie das neuartige Protein funktioniert, so kommt er um eine zielgerichtete Mutagenese meist nicht herum. Im nachfolgenden erfahren Sie mehr über Teil 1 der dazu nötigen Vorarbeiten: das Entwerfen passender Oligonukleotide.
Kaum ist ein Gen kloniert und das kodierte Protein exprimiert und aufgereinigt, erhebt sich die Frage: "Und wie funktioniert das Ding jetzt?" Welche Bedeutung haben einzelne Aminosäurereste für die Proteinaktivität, wie steht es hinsichtlich Katalyse, Oligomerisierung, Substratbindung und -spezifität, Wechselwirkungen mit anderen Proteinen, Stabilität, usw., usw.? Die Sache wird jetzt erst richtig interessant. Wer einen phantasiebegabten Doktorvater hat oder hatte, der weiß, wovon ich rede... - Zitat: "Da machen wir doch noch mal ein schnelles (!) Experiment, zur Vervollständigung der Ergebnisse".
Zielgerichtete Sequenz-
veränderung ist angesagt
Bei der gezielten Veränderung einer vorhandenen DNA-Sequenz hat die Mutation einer oder mehrerer (nahe beieinander) liegender Basen sicherlich die größte Bedeutung für die Funktionsanalyse - sei es von DNA-Sequenzen selbst oder den kodierten Produkten. Bei Proteinen führt dies zum Austausch der Aminosäuren an den entsprechenden Positionen, beeinflusst aber nicht die Kettenlänge. Insertionen oder Deletionen von Basentripletts, also das Hinzufügen oder Entfernen von Aminosäureresten, kommen weniger häufig vor. Sie verändern zusätzlich die Kettenlänge, was dramatische Folgen für die Proteinaktivität haben kann. Das Anfügen von Affinitäts-Tags für die Aufreinigung ist aber inzwischen weit verbreitet (siehe dazu die Artikel zur Protein-Separation in
Laborjournal 06, 09 und 10/2004).
Ohne Oligos geht's nicht
Es gibt viele Methoden. Sie unterscheiden sich grundsätzlich darin, ob sie in einem ihrer Schritte eine DNA-Polymerase verwenden oder nicht. Alle Varianten verwenden allerdings ein kloniertes DNA-Molekül, in aller Regel ein rekombinantes Plasmid, welches die zu verändernden Abschnitte enthält und mindestens ein synthetisches, oligomeres DNA-Fragment ("Oligo"), das die Mutation mitbringt. Für entscheidende Beiträge zu diesen Methoden wurde 1993 der Chemie-Nobelpreis an Michael Smith und Kary Mullis vergeben.
Die im Folgenden aufgeführten Methoden stellen nur Beispiele dar und die Ausführungen erheben keinen Anspruch auf auch nur annähernd erschöpfende Behandlung. Für Anfänger, Quer- und Wiedereinsteiger und solche die es werden wollen, ist vielleicht trotzdem etwas Interessantes dabei.
Kassettenmutagenese
Im einfachsten Fall kommt man ohne DNA-Polymerase aus. Der zu mutierende Bereich wird mit Restriktionsenzymen aus der Ausgangs-DNA herausgeschnitten ("Mutagenese-Rahmen") und durch ein doppelsträngiges DNA-Fragment, die Mutagenese-Kassette, ersetzt. Die Kassette besteht aus zwei miteinander hybridisierten Oligos mit zum Rahmen kompatiblen Überhängen und kann einfach in diesen hineinligiert werden.
Je nach Ansatz kann eine Kassette einen oder zwei Mismatches ausweisen. Im ersten Fall wurde ein Oligo, das die Ursprungssequenz aufweist, mit einem mutierten Oligo hybridisiert. Im zweiten Fall bringen beide Oligos eine Mutation mit. So können diverse Mutanten in einem einzigen Ansatz gewonnen werden. Wenn die Mutagenese-Oligos so synthetisiert werden, dass jedes statistisch im Vergleich zur Ausgangs-DNA eine einzige Mutation an beliebiger Position enthält, bekommt man theoretisch alle möglichen Basenaustausche über die gesamte Kassettenlänge. Welche man erzielt, hängt letztlich auch von der Art der zur Transformation verwendeten Zellen ab und muss durch Sequenzierung festgestellt werden. Wer möglichst viele Mutanten will, sollte Mismatchreparatur-defiziente Zellen verwenden (mehr darüber in Teil 2).
Voraussetzung für diese Art der Mutagenese ist natürlich, dass in der Ausgangs-DNA überhaupt Restriktionsschnittstellen (singulär) vorhanden sind. Und mal ehrlich, wann findet man eine Schnittstelle schon mal da, wo man sie braucht?! Wenn keine geeigneten Schnittstellen vorkommen, können mit zielgerichteter Mutagenese vorher welche eingeführt werden oder man verwendet Mutagenesemethoden, die DNA-Polymerasen verwenden. Während wir bei der Kassettenmutagenese lediglich auf kompatible Enden angewiesen sind, muss für Polymerase-Methoden beim Oligodesign einiges berücksichtigt werden.
Primer-Extension-
Mutagenesen und Oligodesign
Bei Mutagenesen, die DNA-Polymerasen verwenden, wird die Sequenzveränderung durch ein einzelnes Oligo eingeführt. Für die DNA-Synthese muss dieses mit einem Matrizenstrang der Ausgangs-DNA hybridisiert werden und dient dem Enzym dann als Primer, daher auch die Bezeichnung.
Der Sequenzunterschied zur Ausgangs-DNA spiegelt sich in der Anzahl der auftretenden Basenfehlpaarungen wider, wenn man beide DNA miteinander hybridisieren würde. Die Anzahl der dabei auftretenden Basenfehlpaarungen (Mismatches) hängt natürlich von der Zahl der angestrebten Veränderungen ab, sollte aber grundsätzlich möglichst klein gehalten werden. Sehen wir uns mal ein mögliches Beispiel an:
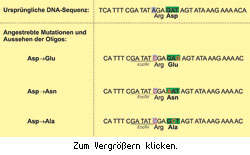
Nehmen wir an, es soll zur Funktionsanalyse eines Proteins die Aminosäure Asp an einer bestimmten Position mutiert werden (Sequenzausschnitt siehe Tabelle). Zunächst wird man sich darüber Gedanken machen, welche Aminosäuren an dieser Position aussagekräftige Ergebnisse ergeben. Aufgrund dieser Überlegungen wird beschlossen, Asp durch Glu bzw. Asn bzw. Ala zu ersetzen. Im vorliegenden Fall wird Asp durch GAT kodiert. Am günstigsten ist es, wenn man sich zunächst für die neu einzuführenden Aminosäuren alle in Frage kommenden Codons aufschreibt. Glu kann durch GAA oder GAG, Asn durch AAC oder AAT und Ala durch GCA, GCC, GCG oder GCT kodiert werden.
Da wir mit möglichst wenigen Mismatchen auskommen wollen, ist es am günstigsten, diejenigen Codons auszuwählen, die sich am wenigsten von GAT unterscheiden, d.h. in der Mutagenese durch die wenigsten Basen ersetzt werden müssen. Für Glu kämen danach immer noch beide Codons in Frage, für Asn würde sich sich AAT anbieten, für Ala das GCT-Codon. Da im Anschluss an die Mutagenese das mutierte Protein aber auch noch exprimiert werden soll, ist es sinnvoll, eine eventuell bestehende Codon-Präferenz der verwendeten Zellen in die Überlegungen einzubeziehen. Es kann da erstaunliche Unterschiede in den Proteinmengen geben, insbesondere wenn eukaryontische Proteine in prokaryontischen Zellen exprimiert werden sollen.
Für unser Experiment wurde beschlossen: Glu soll durch GAA, Asn durch AAT, Ala durch GCT ersetzt werden. Von der Ausgangs-DNA unterscheiden sich die Sequenzen in jeweils einer Base. Das genügt aber nicht, denn wir müssen die Mutanten von der ebenfalls vorhandenen Ausgangs-DNA unterscheiden können.
Natürlich bleibt einem die Sequenzierung nicht erspart, aber man kann positive Kandidaten schon vorher einkreisen. Wir brauchen die Möglichkeit, einerseits auf die erzeugte Mutation zu selektieren, andererseits darf diese Selektionsmöglichkeit die Aminosäuresequenz aber nicht zusätzlich verändern ("stille" Mutationen). Einfach selektieren kann man mithilfe von Restriktionsschnittstellen, man muss sie nur haben. Prinzipiell können Schnittstellen in der Mutagenese erzeugt oder zerstört werden, sie sollten sich nur möglichst dicht an der zu mutierenden Position befinden. Das erfordert einige Sucharbeit, aber die passende Software hilft einem dabei. Im optimalen (und daher seltenen) Fall fallen Screening-Schnittstelle und mutierte Position quasi "automatisch" zusammen. Meist wird man aber versuchen müssen, eine separate Schnittstelle zu erzeugen bzw. zu zerstören. Wir können z.B. in unserer Sequenz durch den Austausch nur einer weiteren Base eine EcoRV-Schnittstelle (GATATC) erzeugen, ohne die Aminsosäuresequenz zu verändern (AGA und CGA kodieren für Arg).
Länger erspart manchen Frust
Macht zusammen zwei Mismatches im Vergleich zur ursprünglichen DNA-Sequenz. Im nächsten Schritt muss festgelegt werden, wie lang die Mutageneseoligos werden sollen. Mein Tipp: Lieber die eine oder andere Base länger, kostet doch nicht mehr die Welt, erspart aber unter Umständen jede Menge Frust. Die Mismatche sollten nicht zu dicht an den Enden liegen, weil ansonsten vielleicht die Hybridisierungsschritte nicht richtig funktionieren. Richtwert: eine Länge von etwa 10 Basen bis zum jeweiligen Ende. Wenn diese Sequenz allerdings AT-reich ist, würde ich sie lieber etwas verlängern.
Für unser Experiment könnten die drei Mutageneseoligos also folgende Sequenzen haben (siehe Abbildung).
So, jetzt bestellen wir erst einmal Oligos. Im nächsten Teil werden dann einige Möglichkeiten vorgestellt, wie man diese mit möglichst hoher Effizienz in Primer-Extension-Mutagenesen einsetzen kann.
Letzte Änderungen: 10.01.2006