Die Bedeutung der Kleinsten
Larissa Tetsch
(10.12.2021) BREMEN/OLDENBURG: Jedes Frühjahr treten in der Nordsee Algenblüten auf, die gigantische Mengen Kohlenstoffdioxid fixieren. Nach ihrem Tod werden die Algen von Bakterien abgebaut. Die Rolle, die Viren dabei spielen, wird erst langsam deutlich.
Im Meer herrscht ein ständiges Werden und Vergehen. Besonders deutlich wird das bei einer Algenblüte, die innerhalb kurzer Zeit erblüht, bis sie sogar aus dem Weltall sichtbar ist, und dann so plötzlich wieder verschwindet wie sie entstanden ist. In der Nordsee spielt sich dieses Schauspiel jedes Jahr im Frühjahr ab, wenn die Sonneneinstrahlung zunimmt.
Verantwortlich für die Blüte sind hauptsächlich Kieselalgen (Diatomeen), die ihren Namen einer Schale aus Kieselsäure verdanken. Sie fixieren dank Photosynthese Kohlenstoffdioxid aus der Atmosphäre. Auf dem Höhepunkt der Blüte speichern die Algen große Mengen an atmosphärischem Kohlenstoff in ihrer Zellmasse und wirken damit dem Treibhauseffekt entgegen. Auf die Algenblüte reagieren Bakterien, die sterbende Algen und von diesen gebildete Zucker abbauen. Dabei wird nur ein Teil des von den Algen fixierten Kohlenstoffs in Bakterienbiomasse umgewandelt, während ein anderer Teil wieder zurück in die Atmosphäre gelangt. Die Vorgänge rund um Algenblüten zu verstehen, ist deshalb ungemein wichtig für das Verständnis des globalen Kohlenstoffkreislaufs.
Nach Viren fischen
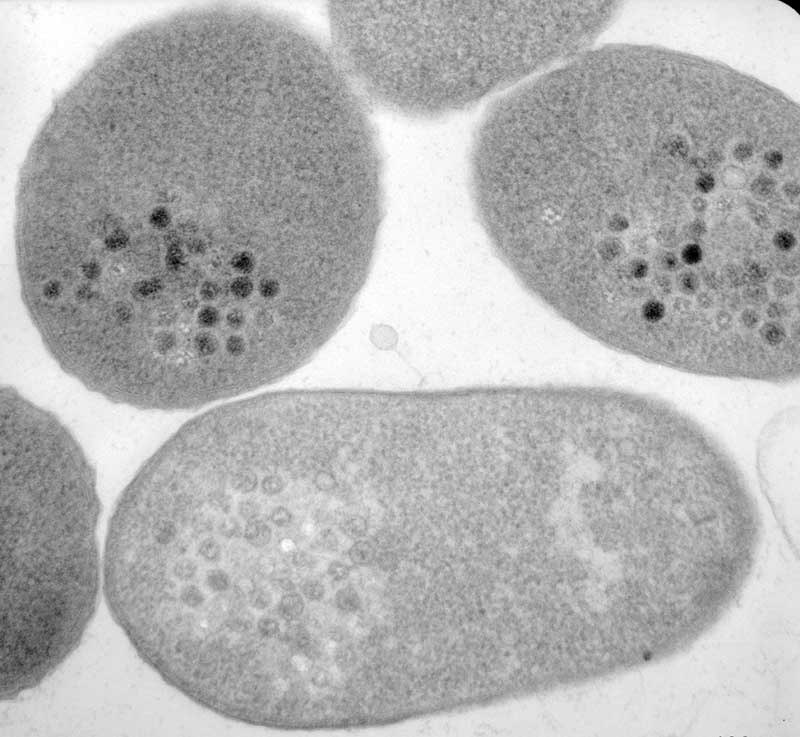
Im Zentrum des Interesses stehen die bakteriellen Algenabbauer unter anderem am Max-Planck-Institut für Marine Mikrobiologie in Bremen in der Arbeitsgruppe von Rudolf Amann. „Die wichtigsten unter ihnen sind die Flavobakterien“, erzählt Nina Bartlau, die als Doktorandin bei Amann geforscht hat. Sie erklärt, dass sterbende Algen eine große Menge an Polysacchariden freisetzen, die sie zuvor als Speicherstoffe gebildet hatten, oder die Bestandteile ihrer Zellwände waren. Flavobakterien aus verschiedenen Gattungen, die der Algenblüte in einer bestimmten Abfolge folgen, bauen die Polysaccharide zu niedermolekularen Verbindungen ab. „Wahrscheinlich wird die Abfolge von der Verfügbarkeit von Substraten bestimmt“, vermutet Bartlau. Entgegen der Erwartung ist die Nahrungskette bei den Bakterien jedoch noch nicht zu Ende: Eine weitere wichtige Rolle spielen Bakteriophagen. Schätzungen zufolge befinden sich in jedem Liter Meerwasser zusätzlich zu einer Milliarde Bakterienzellen noch einmal zehn Milliarden dieser Bakterien tötender Viren.
Dennoch wusste man bisher wenig über sie, weshalb sich Bartlau in ihrer Doktorarbeit den Meeresviren gewidmet hat (ISME J., doi: 10.1038/s41396-021-01097-4). „Phagen, die Flavobakterien befallen – sogenannte Flavophagen – haben wichtige Funktionen im Ökosystem“, weiß die Mikrobiologin. Zum einen schließen sie Stoffkreisläufe, indem sie Bakterien auflösen und ihre Zellkomponenten als Nährstoffe verfügbar machen. Gleichzeitig können Phagen aber auch das Absinken von Zellresten in die Tiefsee fördern, wo diese für rund 1.000 Jahre dem Kreislauf entzogen bleiben. Insbesondere aber erhöhen Phagen die mikrobielle Biodiversität in der Algenblüte, wie Bartlau ausführt: „Phagen infizieren vor allem die häufigsten Bakterien, sodass seltenere Gattungen die Chance bekommen hochzuwachsen. So halten sie das System divers, und diverse Systeme können Umweltstörungen besser abpuffern.“
Über die Vielfalt der Flavophagen in der Nordsee war bis zu Bartlaus Studie kaum etwas bekannt. Lediglich in der Ostsee hatte eine schwedische Gruppe bereits Phagen von einer einzigen Flavobakterien-Gattung untersucht. „Wir haben diese Analysen nun ausgedehnt und fünf weitere Bakteriengattungen untersucht, von denen wir wussten, dass sie in der Frühjahrsblüte wichtig sind“, so Bartlau. Dazu hat die Mikrobiologin in den Jahren 2017 und 2018 in der Zeit der Frühjahrsblüte von März bis Mai jede Woche Proben vor Helgoland genommen und die darin enthaltenen Phagen identifiziert.
Sie isolierte die Phagen schließlich mithilfe von Anreicherungskulturen, für die bestimmte Bakterienstämme, die als Wirte dienen sollten, in Meerwasser inkubierten. Das Wasser hatten Bartlau und Co. zuvor filtriert, um alle Bakterien zu entfernen. „Durch diese Vorgehensweise erhielten die Phagen die Chance, ihren spezifischen Wirt zu finden und sich darin zu vermehren“, erklärt die Mikrobiologin. Und tatsächlich ließen sich auf elf der als potenzielle Wirte eingesetzten Bakterienstämmen Flavophagen kultivieren. „Besonders fasziniert hat uns die große Vielfalt der gefundenen Viren“, so die Forscherin. „Diese spiegelte sich sowohl in der Genetik als auch in der Morphologie wider.“ Bakterienviren bestehen typischerweise aus einem Kopf- und einem Schwanzteil. Je nach Struktur des Schwanzes – also kurz/lang, steif/flexibel, vorhanden/fehlend – werden sie in verschiedene Strukturgruppen eingeordnet. „Alle uns bekannten Strukturgruppen konnten wir in unseren Isolaten wiederfinden“, freut sich Bartlau.
Zwanzig auf einen Streich
Die insgesamt 108 Phagenisolate gehörten zu 44 Stämmen, unter denen 12 neue Arten mit dabei waren. Die taxonomische Einordnung der neuen Viren war eine besondere Herausforderung. Wertvolle Unterstützung bekam Bartlau dabei von Cristina Moraru, die ebenfalls am MPI für Marine Mikrobiologie in der Gruppe von Amann ihre Doktorarbeit gemacht und dort anschließend auch als Postdoc gearbeitet hat. Inzwischen leitet sie am Institut für Chemie und Biologie der Meeresumwelt der Universität Oldenburg eine eigene Gruppe, die sich auch mit Meeresviren beschäftigt. Hier stehen allerdings vor allem Viren im Vordergrund, die heterotrophe Bakterien aus der Gruppe der Rhodobacteraceae befallen. „Wir mussten einen Großteil der Phagen ganz neu einordnen“, so Bartlau. „Cristina hat die Programme dafür wie VirClust und VIRIDIC entwickelt, und sie hat mit mir zusammen die bioinformatische Auswertung der Genomanalysen durchgeführt.“
Am Ende hatten die beiden Wissenschaftlerinnen zehn neue Viren-Gattungen und zehn neue Familien beschrieben, von denen neun beziehungsweise vier ausschließlich aus Phagen bestanden, die Bartlau in ihrer Doktorarbeit erstmals kultiviert hatte.
Wiedersehen in den CRISPRs
Phagen und andere Viren können sich auf zwei unterschiedliche Arten vermehren. Lytische Viren sorgen dafür, dass die Wirtszelle in ihrem Inneren Viruspartikel zusammensetzt, die dann den Wirt lysieren, um sich zu befreien. Zu diesem Zweck codiert das virale Erbgut sogenannte Endolysine, also Enzyme, die die bakterielle Zellwand öffnen können. „Über den Abbau der Zellhülle machen die Viren Nährstoffe gleichzeitig leichter verfügbar“, erläutert Bartlau.
Lysogene Viren integrieren dagegen ihr Genom mithilfe einer Integrase in das Erbgut des Wirts und vermehren sich gleichzeitig mit der Replikation des Bakteriengenoms. Von dort aus können sie entweder auf unbestimmte Zeit im Genom schlummern oder den lytischen Zyklus einleiten. Sowohl Gene von Endolysinen als auch von Integrasen wurden in den Genomen der Flavophagen gefunden. Einige Phagen scheinen sogar von der Zelldichte des Bakteriums abhängig machen zu können, ob sie den lytischen oder lysogenen Weg beschreiten wollen. Ihr Erbgut codiert einen Transkriptionsfaktor, den viele Meeresbakterien verwenden, um Gene in Abhängigkeit der eigenen Zelldichte zu regulieren.
„Besonders wichtig ist uns, dass wir zeigen konnten, dass unsere Phagen tatsächlich in der Umwelt aktiv waren, also dass genau diese Phagen in der Frühjahrsblüte Bakterien infiziert hatten“, betont Bartlau. Dazu waren zwei verschiedene Ansätze zum Einsatz gekommen. Zum einen untersuchte das Team CRISPR-Sequenzen in Bakterienstämmen, die sie bereits vor Beginn der Studie isoliert hatten. Zur kurzen Erinnerung: Die CRISPR-Sequenzen sind der Teil des bakteriellen Immunsystems, in denen die Sequenzen von Viren gespeichert sind, die das betreffende Bakterium zu einem früheren Zeitpunkt infiziert haben. Die Anwesenheit von Spuren von drei der zwölf neu isolierten Phagen in CRISPR-Sequenzen zeigt also, dass diese Phagen schon vor 2017 vor Helgoland aktiv waren.
Ein weiterer Hinweis auf ihre Relevanz in der Umwelt ist, dass sie in Metagenomen nachweisbar waren. „Wir konnten die Phagen in Metagenomen nachweisen, die wir aus einem Liter Meerwasser gewonnen hatten“, erklärt die Mikrobiologin. „Die Phagen waren zu bestimmten Zeiten in hohen Zahlen vorhanden, zu anderen Zeiten waren sie verschwunden. Das bedeutet, dass sie aktiv produziert werden mussten.“ Die neuen Flavophagen sind also ein stabiler und aktiver Bestandteil der mikrobiellen Gemeinschaft vor Helgoland und tauchen jährlich wiederkehrend auf. „Damit konnten wir ganz neue Modellsysteme aus der Umwelt ins Labor übertragen“, ist Bartlau überzeugt.
Inzwischen arbeitet die Wissenschaftlerin als Postdoc an der Universität Wien. In der dortigen Abteilung für Mikrobielle Ökologie untersucht sie weiterhin Meeresviren, allerdings nun die Phagen einer anderen Gruppe von Bakterien – der Vibrionen – im Atlantik. Die Flavophagen sind in der Deutschen Sammlung von Mikroorganismen und Zellkulturen (DSMZ) in Braunschweig hinterlegt und für andere Wissenschaftler und Forscherinnen frei zugänglich. „Aktuell arbeiten eine schwedische und eine finnische Gruppe damit weiter“, freut sich Bartlau und hofft dennoch, eines Tages selbst wieder auf „ihre“ Phagen zurückgreifen zu können: „Ich glaube, es gibt an den Flavophagen noch viel Neues und Spannendes zu entdecken.“