Angemalte Zellkompartimente – Nebenwirkungs-Screening von Wirkstoff-Kandidaten
Karin Hollricher
(07.10.2022) „Ein Bild sagt mehr als tausend Worte”. Das gilt auch für den Cell-Painting-Assay, der die biologische Wirksamkeit von Prüfsubstanzen durch morphologische, bildgebende Verfahren anzeigt.
Die Entwicklung neuer pharmazeutischer Therapeutika ist ein riskantes Geschäft. Neunzig Prozent aller potenzieller Wirkstoffe fallen durch, hauptsächlich wegen fehlender Wirksamkeit, gefolgt von zu hoher Toxizität.
Aus ökonomischer wie medizinischer Sicht sollte man daher schon in einem frühen Stadium der pharmakologischen Entwicklung testen, welche Wirkung eine Substanz auf das zelluläre Geschehen hat – und zwar im Hochdurchsatz mit vergleichsweise geringem finanziellem und personellem Aufwand. „Wir haben hier eine MPG-eigene Screening Facility, das Compound Management and Screening Center, kurz COMAS, in der wir neue Substanzen, die auf der Basis naturstofflicher Grundgerüste synthetisiert wurden, auf ihre biologische Aktivität prüfen. Deshalb suchten wir nach einem Verfahren, mit dem sich die zelluläre Wirkung einer Substanz möglichst umfassend untersuchen lässt, sodass wir Informationen über ihre Wirkweise gewinnen sowie potenziell schädliche Substanzen möglichst früh aussortieren können”, berichtet Slava Ziegler. Sie leitet am Max-Planck-Institut für Molekulare Physiologie in Dortmund eine Projektgruppe in der Abteilung Chemische Biologie.
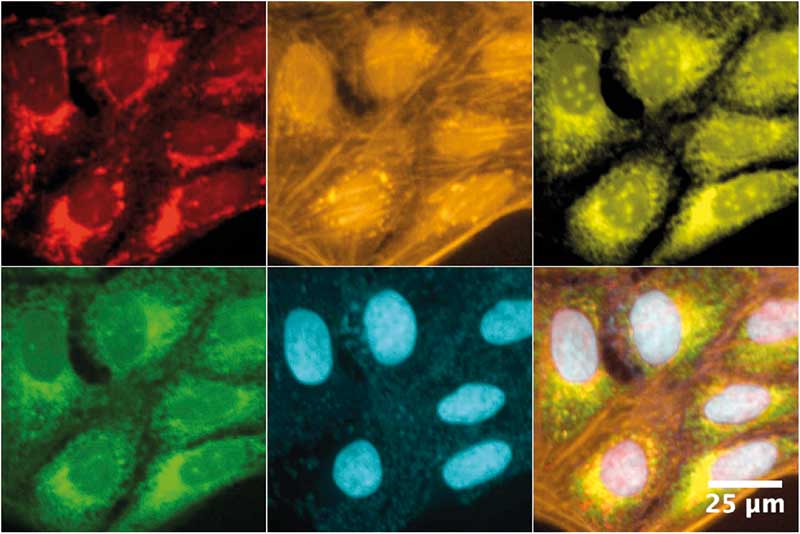
Tatsächlich gab es mit dem Cell-Painting-Assay (CPA), der am Broad Institute im US-amerikanischen Cambridge entwickelt worden war, bereits eine entsprechende Technik (Nat. Protoc. 11: 1757-74). Der Clou des CPA ist die gleichzeitige Markierung verschiedener Zellkompartimente, die die bildgestützte, automatisierte Analyse des Zell-Phänotyps ermöglicht. Die gewählten Farbstoffe sollten sich jedoch spektral nicht ins Gehege kommen. Laut Protokoll färbt man Zellkerne mit Hoechst 33342 blau, das endoplasmatische Retikulum (ER) mit Concanavalin A grün, die RNA der Nukleoli mit SYTO14 gelb und Mitochondrien mit MitoTracker rot. Den Golgiapparat markiert man mit Weizenkeim-Agglutinat und Aktin mit Phalloidin. Die beiden letztgenannten Farbstoffe fluoreszieren zwar bei sehr ähnlichen Wellenlängen orange, doch kann man sie aufgrund ihrer Bindung an sehr unterschiedliche Zellstrukturen leicht auseinanderhalten. Auf diese Weise lässt sich die Fluoreszenz der eingesetzten Farbstoffe auf fünf Kanälen gleichzeitig auslesen.
„Das klang auf dem Papier sehr verlockend und recht einfach”, sagt Ziegler. „Auf 384-Loch-Platten kann man damit über 1.000 Parameter auswerten und erreicht einen höheren Durchsatz als mit anderen Profilierungsmethoden wie Transkriptomik oder Proteomik.”
Dennoch dauerte es in Dortmund länger, bis der Cell-Painting-Assay reibungslos lief. Der Knackpunkt war die morphometrische Analyse, also die automatisierte Bildauswertung. Der Einfluss einer Substanz ist nur in seltenen Fällen so gravierend und eindeutig, dass man ihre Effekte schon mit bloßem Auge erkennen kann. In der Regel benötigt man eine Software, um in den dicht gepackten Zellen, in denen sich die bunten Markierungen überlagern, Veränderungen zu identifizieren. „Die Software ‚Cell Profiler‘, die am Broad Institute entwickelt wurde, sieht weit mehr als das Auge allein”, so Ziegler.
Nachdem der CPA einwandfrei funktionierte, testeten die Forschenden mit ihm zunächst Referenzsubstanzen, von denen sie wussten, was sie in der Zelle anstellen, woran sie binden und welche Signalwege sie beeinflussen. Darauf aufbauend überprüften sie die Wirkung neuer, noch nie getesteter (nicht annotierter) Substanzen – etwa auf Tubulin. „Es gibt zwar In-vitro-Tubulin-Tests, doch sie spiegeln aus verschiedenen Gründen nicht immer die zelluläre Realität wider“, erklärt Ziegler. Im CPA produzierten die Referenzsubstanzen Nocodazol, Vincristin und Vinblastin, die bekanntermaßen an Tubulin binden und damit die Zellteilung beeinträchtigen, reproduzierbar sehr ähnliche CPA-Fingerprints. Mit diesen verglichen die Forschenden die CPA-Profile ihrer Substanzbibliothek (Cell Chem. Biol. 29, 1-12). Moleküle verschiedenster Strukturen erzeugten Tubulin-spezifische Fingerprints, etwa Benzimidazol-Derivate. Dass diese heterozyklischen, aus je einem Benzol- und Imidazolring bestehenden Aromaten mit Tubulin interagieren, ist bekannt und wurde im CPA bestätigt. Auch der Naturstoff Rotenon lieferte den für Tubulin-interagierende Substanzen typischen Phänotyp. Eigentlich ist Rotenon ein Inhibitor des mitochondrialen Komplexes I und dadurch der Atmungskette; allerdings verhindert der Naturstoff auch die Entstehung von Mikrotubuli. So lässt sich der CPA-Phänotyp erklären.
Die Forschenden fanden Nocodazol-typische Phänotypen aber auch mit Substanzen, von denen bisher nicht bekannt war, dass sie Tubulin beeinflussen würden. Dazu gehörten auch verschiedene Proteinkinase-Inhibitoren. „Der biologisch-pharmakologischen Grundlage dieses Effekts kann man nun auf den Grund gehen“, so Ziegler. In der Bibliothek von COMAS fanden die Forschenden fünfzehn Verbindungen, die ähnliche phänotypische Profile wie Nocodazol zeigten. Alle inhibieren in vitro die Tubulin-Polymerisation sowie das Wachstum der Kontroll-Zelllinie U2OS.
Dieses Analyse-Prinzip lässt sich generalisieren: Bilden sich in Gegenwart völlig unterschiedlicher Substanzen ähnliche phänotypische Profile aus, kann man davon ausgehen, dass die Moleküle dieselbe biologische Aktivität beeinflussen oder sogar dasselbe Ziel beziehungsweise denselben Signalweg modulieren. Daher landen sie im selben Cluster – auch wenn sie strukturell völlig unterschiedlich sind (Chembiochem 21: 1-12). Hierdurch gelangte Rotenon in den Cluster für Tubulin-bindende Moleküle. Auch Inhibitoren der Kinasen mTOR, AKT und PI3K fanden sich in einem gemeinsamen Cluster wieder. Obwohl sie unterschiedliche Zielmoleküle ansteuern, aber im selben Signalweg aktiv sind, erzeugen sie sehr ähnliche phänotypische Profile.
„Wenn Veränderungen immer eindeutig denselben Zellbestandteilen zuzuordnen sind, wie bei den Tubulin-Experimenten, ist die Bildauswertung bei der Prüfung unbekannter Substanzen eher einfach”, berichtet Ziegler. „Schwierig wird es, wenn eine neue Verbindung ein bis dahin noch nicht beobachtetes Profil zeigt und man auch kein Target dieser Verbindung kennt.”
Genau das passierte, als die Forschenden vier Substanzen untersuchten, die auf der Basis eines Indolochinolizin-Grundgerüsts synthetisiert worden waren. Naturstoffe dieser Bauart zeigen vielerlei biologische Aktivität. Aber nur eine Verbindung lieferte einen Fingerprint, der den Profilen vieler anderer annotierter Verbindungen ähnlich war. „Trotzdem konnten wir dieser Substanz keine Aktivität zuordnen, da alle ähnlichen Referenzverbindungen sehr unterschiedliche annotierte Wirkweisen hatten“, erklärt die Wirkstoff-Forscherin.
Ihr Team führte daher ein Proteom-Profiling durch. Dabei stellte sich heraus, dass das besagte Molekül in die Cholesterin-Verstoffwechslung eingreift – ebenso wie 26 weitere Verbindungen. „Elf Prozent der von uns getesteten Referenzverbindungen zeigten dieses Profil, das waren 449 von insgesamt 3.951 im CPA getesteten Referenzsubstanzen“, so Ziegler. „Das hatten wir nicht erwartet.”
Der Effekt tritt häufig über Umwege auf, die zu Lysosomen führen. Die meisten Moleküle mit dem beobachteten morphologischen Profil erhöhen nämlich den pH-Wert in diesen Organellen – und das stört den Cholesterin-Metabolismus. „Über dieses morphologische Profil kann man den Einfluss von Substanzen auf die Cholesterin-Synthese nachweisen und erklären, was meist nichts mit ihrer annotierten Wirkweise zu tun hat“, freut sich Ziegler.
Die ergebnisoffene, morphologische, bildbasierte Bestimmung der biologischen Aktivität einer Prüfsubstanz im Hochdurchsatz erfüllt die technischen Vorgaben, die die US-Behörde Environmental Protection Agency für toxikologische Prüfungen vorschlägt. Die CPA-Entwickler um Anne Carpenter vom Broad Institute gehen deshalb davon aus, dass sich die Technik rasch weiterentwickelt (Nat. Rev. Drug. Disc. 20: 145-59).
Sie machten sich auch gleich an die Arbeit und starteten JUMP-CP (Joint Undertaking in Morphological Profiling with Cell Painting), an dem auch das MPI in Dortmund beteiligt war. Die Zielvorgabe lautet: Generiere einen Datensatz, der die Information aus einer Milliarde Zellen unter dem Einfluss von 140.000 Substanzen widerspiegelt.
Das wird spannend, denn mit dem Assay lassen sich nicht nur mögliche Nebenwirkungen vorhersagen, sondern auch (neue) Funktionen von Proteinen aufdecken und modulieren.