Riboswitch
von Michaela Petter (Laborjournal-Ausgabe 01, 2004)
Anfang der 80er Jahren entdeckten die beiden späteren Nobelpreisträger Thomas R. Cech und Sidney Altman, dass RNA enzymatisch wirken kann. Mit der Entdeckung der so genannten Ribozyme dämmerte ein Ausweg aus dem Paradoxon, dass Biologen lange Zeit zum Grübeln brachte: Wenn DNA den Bauplan für Proteine kodiert, Proteine jedoch für die Synthese von DNA unerlässlich sind, wie konnten diese beiden komplexen Moleküle je unabhängig voneinander entstehen?
Die Ribozyme waren der erste handfeste Hinweis darauf, dass möglicherweise selbstrepliziernde RNA der lang gesuchte Schlüsselspieler in der Entwicklung des zellulären Lebens sein könnte. Walter Gilbert gab dieser Theorie 1986 schließlich den griffigen Namen "RNAWelt".
Dass RNA mehr ist als ein primitives Botenmolekül, zeigten bald auch andere: So fand Sol Spiegelman, dass man bei RNA, die in Gegenwart von degradierenden Enzymen (Ribonukleasen) repliziert, Resistenz induzieren kann. RNA kann sich demnach an "Umweltveränderungen" anpassen und neue Eigenschaften annehmen. Eine grundlegende Voraussetzung für die Evolution von Leben.
Vitamin bindet an RNA
Darwinistische Prinzipien ermöglichten es auch, in vitro kurze RNA-Oligonukleotide anzureichern, die selektiv Liganden binden können. Ähnlich wie Proteine bilden RNAs einzigartige Tertiärstrukturen aus, sodass Bindungstaschen entstehen können, die solch hochspezifische Interaktionen erlauben. RNA kann demnach als Rezeptor funktionieren. Solch strukturierte RNA Moleküle bezeichnet man als Aptamer.
Diese und ähnliche Entdeckungen warfen natürlich die Frage auf: warum macht sich die Natur diese simplen Prinzipien nicht zu Nutze? Oder hatte man bisher nur kein Beispiel identifiziert?
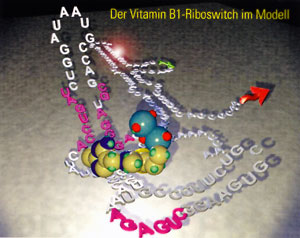
Eine Antwort kam 2002 aus dem Labor von Ronald R. Breaker an der Yale University. Seiner Gruppe gelang es, erstmals einen natürlichen "Riboswitch" zu charakterisieren - ein Kontrollelement in der Leader-Sequenz einer mRNA, das sich eben dieser Bindungskapazität bedient (Nature 419, S. 952). Auf der Suche nach Kofaktoren, die die Expression von Proteinen des Vitamin B1-Biosynthesewegs regulieren, entdeckte Erstautor Wade Winkler Erstaunliches: es gibt keine. Das Endprodukt bindet - genau wie beim Aptamer - direkt an die mRNA und greift dadurch in die Genexpression ein. Das THI-Element der entsprechenden mRNAs, mit dem Vitamin B 1 (Thiaminpyrophosphat) interagiert, funktioniert also wie ein molekularer Schalter: ein Riboswitch.
Fast wie eine Operon-Kontrolle...
Die Mechanismen, nach denen ein Riboswitch funktioniert, sind eigentlich altbekannt. in Bakterien unterliegen Gene, die in einem bestimmten Stoffwechselweg zusammenspielen, oft einer gemeinsamen Regulation. Die Expression solcher bakterieller Operons wird häufig durch die Ausbildung zweier alternativer Strukturen in der mRNA gesteuert. in der einen, der repressiven Konformation, verursacht entweder eine Terminationsschleife den frühzeitigen Abbruch der Transkription, oder die Translation wird inhibiert, weil die Shine-Dalgarno Sequenz als Erkennungsstruktur der Ribosomen durch intramolekulare Basenpaarung unzugänglich ist. In der alternativen Konformation dagegen werden weder Transkription noch Translation blockiert. Welche der beiden Strukturen sich ausbildet, hängt von der Konzentration eines Metaboliten ab, der das Produkt oder das Ziel des regulierten Stoffwechselweges sein kann.
... Allerdings ohne Kofaktor
Als Sensoren wirken in solchen Feedback-Kreisläufen normalerweise Proteine, die den Metaboliten binden. Durch die Interaktion mit dem Liganden verändert sich die Affinität des Sensors zu einer Erkennungssequenz im nicht-kodierenden Bereich einer bestimmten mRNA, was letztlich die regulatorische Umfaltung der RNA bewirkt. Ein bekanntes Beispiel für solch einen sensorischen Schalter ist das TRA-Protein, welches abhängig vom Tryptophan-Level in der Zelle die Expression von Enzymen der Tryptophan-Biosynthese reguliert.
Im Gegensatz dazu fehlt beim Riboswitch solch ein zwischengeschalteter Kofaktor: die RNA ändert die Konformation, nachdem der Metabolit direkt an deren Leader-Sequenz bindet.
Inzwischen beschrieben Breakers Leute und andere eine ganze Reihe weiterer Riboswitches. Neben Vitamin B1 steuern Vitamin B2 und Coenzym B12 ebenfalls über direkte Interaktion mit der mRNA ihre eigene Biosynthese. Lysin und Methionin regulieren sich ebenfalls auf diese Weise selbst, und weitere Daten zeigen, dass auch Purine solch allosterische Riboswitches nutzen.
Insgesamt sind bereits hunderte von Genen aus den unterschiedlichsten Genomen bekannt, die über die sechs bisher charakterisierten RNA-Schalter reguliert werden. Oft korreliert der bevorzugte Kontrollmechanismus mit der Taxonomie - so tritt bei Gram-positiven Bakterien vor allem Attenuation der Transkription auf, während Riboswitches bei Gram-negativen Bakterien eher die Translation inhibieren.
Uralter Mechanismus?
Die große Vielfalt von Organismen, über die Riboswitches verbreitet sind, deutet auf ihren frühen Ursprung in der Evolutionsgeschichte hin. Bemerkenswert ist außerdem, dass die Metaboliten, die die molekularen Schalter bedienen, häufig strukturell mit Nukleotiden verwandt sind - und somit zeitgleich mit der RNA existiert haben könnten.
Haben wir also ein molekulares Fossil aus der RNA-Welt entdeckt? Das zu beweisen steht noch aus. Sicher ist jedenfalls, dass der Riboswitch eine hypothetische RNA-Welt noch um eine Stufe komplexer macht - und damit womöglich wahrscheinlicher.
Letzte Änderungen: 20.10.2004