Sehen ohne Linse
(26.06.2019) Mikroskopie mit Nukleinsäuren und PCR-Reaktionen? Klingt ziemlich durchgeknallt. Scheint aber tatsächlich zu funktionieren.
Die Grundlagen der Mikroskopie haben sich seit ihrer Erfindung kaum verändert: Bei mikroskopischen Verfahren wird die Probe mit Photonen (Lichtmikroskopie) oder Elektronen (Elektronenmikroskopie) punktuell beschossen. Die Ergebnisse betrachtet der Experimentator direkt durch die Linse des Mikroskops oder indirekt als Rekonstruktion am Bildschirm eines Computers. Die räumliche Auflösung resultiert aus den physikalischen Ereignissen, die die Lichtquanten auslösen, wenn sie an einem bestimmten Punkt auf die Probe treffen. Hierzu zählt zum Beispiel die Anregung von Fluorophoren.
Eine neue Mikroskopie-Technik, die Joshua Weinstein, Aviv Regev und Feng Zhang vom Massachusetts Institute of Technology in Cell veröffentlichten, bricht radikal mit diesem klassischen Konzept der optischen, auf Lichtteilchen beruhenden Mikroskopie: Die von dem Trio entwickelte sogenannte DNA-Mikroskopie nutzt chemische Reaktionen, um die räumliche Position von Molekülen innerhalb einer Probe exakt zu bestimmen.
Mit Thermocycler und Sequenzierer
Bei der DNA-Mikroskopie werden keine optischen Linsen als technische Hilfsmittel verwendet, sondern Thermocycler und DNA-Sequenzierer. Im ersten Schritt werden RNA-Transkripte in situ mit randomisierten sogenannten Unique Molecular Identifiern (UMI) individuell markiert. Der zweite Schritt besteht darin, die in situ markierten Moleküle durch Overlap-Extension-PCR mithilfe von markierten Primern zu amplifizieren, wobei die PCR-Produkte lokal aufkonzentriert werden.
Die Amplikons bleiben aber nicht statisch an einem Ort in der Zelle liegen, sondern vagabundieren durch Diffusion in ihr herum. So gelangen sie zu benachbarten Sequenzen, die ebenfalls mit UMIs markiert sind, hybridisieren mit diesen und bilden schließlich nach der Amplifikation ein Concatamer, das durch den markierten Overlap-Extension-Primer mit einem sogenannten Unique Event Identifier versehen ist.
Aus der Rate, mit der sich die neuen Moleküle miteinander verknüpfen, lässt sich ihre ursprüngliche Position ermitteln. Um die Verteilung der Sequenzen innerhalb der Zelle zu verlangsamen, verwendeten die Forscher ein Hydrogel, das die Concatamere in der Nähe ihres ursprünglichen DNA-Moleküls hält. Während die markierte DNA durch die Zelle diffundiert, bilden sich immer wieder lokale Concatamere, bis die PCR gestoppt wird.
Straßenkarte ermittelt Herkunft
Die Concatamere werden anschließend sequenziert, wodurch eine Sequenz-Bibliothek mit einer langen Liste von DNA-Molekülen entsteht, die den Weg der Concatamere durch die Zelle abbildet. Diese Liste kann man sich wie die Entfernungs-Tabelle einer Straßenkarte vorstellen. Mithilfe der jeweils zurückgelegten Distanzen erstellte das Team eine räumliche Rekonstruktion der DNA-Verteilung innerhalb der Zelle. Dazu verwendeten die US-Amerikaner einen Algorithmus, der die wahrscheinlichste Distanz zwischen den einzelnen aufkonzentrierten Sequenzen berechnet und daraus die ursprüngliche Herkunft der jeweiligen Sequenz ermittelt.
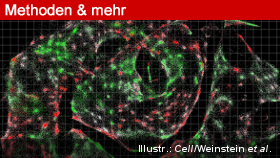
Diese Technik ähnelt dem Triangulations-Verfahren, das Kommunikationsunternehmen für die Handy-Ortung verwenden. Die DNA-Mikroskopie ist deshalb eher ein DNA-Mapping-Verfahren, das räumliche Informationen grafisch darstellt, als ein Mikroskop-Verfahren. Es entstehen aber die gleichen Bilder wie bei der optischen Mikroskopie: Die Gruppe um Zhang verglich die berechneten Bilder der DNA-Mikroskopie mit lichtmikroskopischen Aufnahmen und fand eine hohe Übereinstimmung.
Für viele zugänglich
Die DNA-Mikroskopie löst aber nicht nur die Position zellulärer Moleküle auf. Sie kann auch Mutationen in Sequenzen aufspüren und zum Beispiel zwischen den Transkript-Varianten zweier Allele unterscheiden. Sie liefert also nicht nur räumliche, sondern auch genetische Informationen, was insbesondere für Fragestellungen in der Pathologie interessant ist. Dabei beschränkt sich die DNA-Mikroskopie nicht nur auf RNA-Transkripte. Theoretisch kann man auch Proteine oder Antikörper mit entsprechenden DNA-Sequenzen markieren und dann für die DNA-Mikroskopie verwenden. Da für die DNA-Mikroskopie keine teuren Mikroskope, sondern nur molekularbiologische Standard-Techniken nötig sind, ist sie zudem für viele Labore zugänglich.
Frederique Wieters
Weinstein J. et al. (2019): DNA Microscopy: Optics-free spatio-genetic imaging by a stand-alone chemical reaction. Cell, DOI: 10.1016/j.cell.2019.05.019