Rätsel der RNA-Welt
(17.10.17) Die meisten Ribonukleasen P sind Ribozyme, bei denen die katalyische RNA von einem oder mehreren Proteinen flankiert wird. Es gibt aber auch RNA-freie Varianten. So baut etwa eine Gruppe extrem hitzeliebender Bakterien für die Reifung ihrer Transfer-RNAs ganz auf solch eine „Protein-only“-RNase P – und gibt der "RNA-Welt"-Hypothese damit was zum Knobeln.
Sidney Altmann und Thomas Cech erhielten 1989 den Chemie-Nobelpreis für ihre Entdeckung, dass nicht nur Proteine chemische Reaktionen katalysieren können, sondern dass auch manche RNA dazu in der Lage ist. Ein solches RNA-basiertes Enzym wird passenderweise als Ribozym bezeichnet.
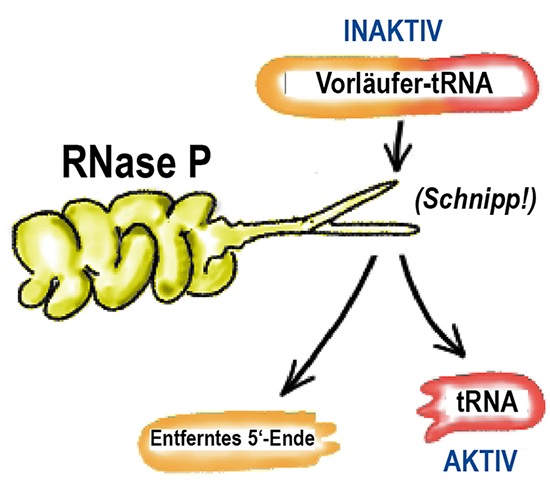
Vielleicht haben Sie früher einmal für eine Studienprüfung einige Ribozyme auswendig lernen müssen? Sciher war dann neben den Ribosomen und den Spleißosomen auch die Ribonuklease P (RNase P) dabei. Diese ist essentiell für jede Zelle und besorgt die Reifung der Transfer-RNAs, indem sie nach deren Synthese am 5’-Ende einen Teil der Vorläufer-tRNA abspaltet. Erst diese reife tRNA ist in der Lage, Aminosäuren zum Ribosom zu transportieren.
Normalerweise besteht die RNase P aus einer katalytisch aktiven RNA und einer (bei Bakterien) oder mehreren Proteinuntereinheiten (bei Archaeen und Eukaryoten). Daneben gibt es in menschlichen Mitochondrien, Landpflanzen und einigen Protisten eine zweite Variante der RNase P, die ihre katalytische RNA verloren hat und lediglich aus ein bis drei Proteinuntereinheiten besteht. Folglich hat die Proteinlastigkeit des Enzyms offenbar im Lauf der Evolution zugenommen.
Für bakterielle Varianten der RNase P interessieren sich unter anderem die Pharmazeutischen Chemiker der Universität Marburg unter Leitung von Roland Hartmann. Da die Ribozyme für die Bakterien überlebenswichtig und gleichzeitig ganz anders aufgebaut sind als diejenigen von Eukaryoten, stellen sie ein lohnendes Ziel für die Entwicklung von Antibiotika dar. So gelang es den Marburger Forschern, die RNA-Komponente mit einem Antisense-Oligonukleotid in eine inaktive Konformation zu überführen, die die Vorläufer-tRNA nicht mehr binden konnte. Mit Hilfe eines computergestützten Wirkstoffdesigns fanden sie außerdem einen Hemmstoff, der sich in die zentrale Furche der Proteinkomponente einlagert. Und kürzlich haben die Forscher in dem Bakterium Aquifex aeolicus gar einen ganz neuen RNase P-Typ gefunden (PNAS, doi: 10.1073/pnas.1707862114)...
Vertreter der Gattung Aquifex , die der Regensburger Mikrobiologe Karl Otto Stetter vor 25 Jahren in heißen Quellen entdeckte, mögen es heiß: mit einem Temperaturoptimum von 85 bis 95°C gehören sie zu den hyperthermophilen Bakterien (sind also trotz ihrer extremen Vorlieben keine Archaeen!). Aquifex und seinen Verwandten aus der Familie der Aquificaceae fehlen die Gene für eine typische bakterielle RNase P. Dies sind rpnA, das ein etwa 14 kDa großes Protein kodiert, und rpnB, das in die etwa 400 Nukleotide lange RNA transkribiert wird. Dennoch liegen die tRNA-Gene bei den Aquificaceae in Bakterien-typischen Operons aus mehreren Genen vor, von denen ein gemeinsames Transkript abgeschrieben wird. Dieses muss anschließend konkret in die einzelnen Genprodukte aufgespalten werden. Und da Aquifex aeolicus tRNAs mit den entsprechenden reifen 5‘-Enden bildet, war anzunehmen, dass das Bakterium dennoch ein Enzym mit RNase P-Aktivität besitzen muss.
Und jetzt kommt Aquifex…
Mit Hilfe von aufeinander aufbauenden, chromatografischen Schritten konnten die Marburger das verantwortliche Enzym reinigen und über SDS-PAGE und Massenspektrometrie als 22,6 kDa großes Protein mit RNase P-Aktivität identifizieren. Rekombinant hergestellte A. aeolicus-RNase P spaltete Vorläufer-tRNAs, ohne Kofaktoren oder Interaktionspartner zu benötigen, womit es sich um die kleinste bislang bekannte RNase P handelt. Umsatzrate und Affinität waren dabei vergleichbar mit denen der „Protein-only“-RNase P aus der Ackerschmalwand Arabidopsis thaliana. Offensichtlich bildet die A. aeolicus-RNase P ein stabiles Homotrimer, von dem sich wiederum sechs zu einem 420 kDa großen Komplex zusammenlagern. Im Unterschied etwa zur RNase P aus E. coli überstand dieser eine zehnminütige Inkubation bei 85°C ohne nennenswerten Aktivitätsverlust.
Bei eukaryotischen „Protein-only“-RNasen wird ein katalytischer Mechanismus vermutet, der zwei Metallionen einschließt, die von mehreren Aspartat-Resten koordiniert gebunden werden. Das A. aeolicus-Enzym weist Sequenzähnlichkeit zu der entsprechenden PIN-Ribonuklease-Domäne auf und besitzt drei konservierte Aspartat-Reste: D138, D142 und D160. Deren Deletion führte zu einem Aktivitätsverlust, während die Deletion von D144, das in der Nähe des aktiven Zentrums liegt, kaum einen Effekt hatte. In E. coli eingebracht konnte die A. aeolicus-RNase P einen Verlust der eigenen RNase P kompensieren und sogar den 400 KDa großen Ribozymkomplex aus Saccharomyces cerevisiae funktional ersetzen.
Moleküle aus der Frühzeit des Lebens
Homologe zur A. aeolicus-RNase P (HARP) finden sich bei vielen Archaeen und einigen Bakterien. Im Gegensatz zu den Bakterien besitzen die Archaeen alle zusätzlich zu den HARP noch die Ribonukleoprotein-Variante der RNase P. Die Marburger Forscher schließen daraus, dass beide Enzyme unterschiedliche Funktionen wahrnehmen müssen. Bakterien, die beide RNase P-Varianten aufweisen, scheinen dagegen eine Art evolutive Übergangsform darzustellen, die von den Aquificaceae überwunden wurde, indem sie die ursprüngliche RNA-Variante verloren. Im Einklang mit der Hypothese, dass die Katalyse in der Frühzeit des Lebens von der RNA und erst später von Proteinen wahrgenommen wurde („RNA-Welt“), vermuten die Forscher, dass die RNase P zuerst ausschließlich aus katalytisch aktiver RNA bestand. Später hätte nach diesem Szenario diese RNA bei den Bakterien ein und bei den Archaeen unabhängig davon fünf Proteine rekrutiert. Die eukaryotischen RNasen P hätten dann zusätzlich zu den Proteinen der Archaeen noch einmal vier bis fünf weitere hinzugewonnen.
Die Spur führt zu den Archaeen
Die „Protein-only“-Variante von A. aeolicus stammt wahrscheinlich ursprünglich aus Archaeen und wurde irgendwann von ihnen durch horizontalen Gentransfer erworben. Immerhin stammen stolze zehn Prozent der proteinkodierenden Gene des Bakteriums aus Archaeen – und zwei davon kodieren für RNA-Bindeproteine, wie sie für RNasen P aus Archaeen typisch sind. Die beiden Gene liegen in unmittelbarer Nähe zum Gen der A. aeolicus-RNase P und sind wohl gemeinsam mit diesem übertragen worden. Welche Aufgabe indes die beiden RNA-Bindeproteine in Aquifex wahrnehmen, ist noch völlig unklar. Bakterien, die zusätzlich zu einem HARP eine herkömmliche Ribonukleoprotein-RNase P produzieren, besitzen sie nämlich nicht mehr. Auch warum sich die Aquificaceae als bislang einzige bekannte bakterielle Gruppe vollständig auf eine „Protein-only“-RNase P verlassen, ist noch ein Rätsel. Eines, dass die Marburger Wissenschaftler allerdings in Bälde aufklären wollen.
Larissa Tetsch
Letzte Änderungen: 09.11.2017