Drei auf einen Streich: die Life Science Wochenschau (8)
(3.4.16) Drei besondere Fundstücke aus dem Forschungsticker der vergangenen Woche.
Zellkern aus Mito-Material?
Wie entstand der Zellkern? Möglicherweise aus Veskel-Material, das ursprünglich vom Mitochondrium stammt, spekulieren Düsseldorfer Molekularbiologen um Sven Gould, Sriram Garg und William Martin:
"Alle höheren Lebewesen wie Pflanzen und Tiere bestehen aus eukaryotischen Zellen. In ihrem Inneren gibt es durch Zellmembranen separierte Untereinheiten, so genannte Kompartimente. [...]
Ein Kompartiment aller tierischer und pflanzlicher Zellen, das Mitochondrium selbst, ist durch „Endosymbiose“ entstanden: Vor fast 2 Milliarden Jahren wurde ein Bakterium in einem anderen Prokaryoten (einem Archaeon) aufgenommen. Aus dieser Integration einer Zelle in eine andere entstanden nicht nur die Mitochondrien, die in keiner eukaryotischen Zelle fehlen, sondern die Eukaryoten selbst.
PD Dr. Sven Gould, Sriram Garg und Prof. Dr. William Martin vom Institut für Molekulare Evolution der Heinrich-Heine-Universität Düsseldorf fragten sich, wie während der Evolution die anderen Kompartimente entstanden. [..]
Demnach stand das Mitochondrium am Anfang von allem. Nachdem es in die Wirtszelle durch Endosymbiose integriert wurde, produzierte es Vesikel, die miteinander verschmolzen und so innere Kompartimente bildeten. Diese haben dann auch die DNA umhüllt und so den Zellkern gebildet."
Komplette Pressemitteilung hier.
Abb.: Sven Gould / Trends in Microbiology
 Malen nach Zahlen mit DNA
Malen nach Zahlen mit DNA
DNA-PAINT ist eine hochauflösende Mikroskopietechnik, bei der fluoreszierende Farbstoffe mit DNA-Sonden verknüpft werden. Ralf Jungmann und sein Team am Max-Planck-Institut für Biochemie haben diese Methode nun weiterentwickelt – die Forscher können jetzt auch die Anzahl der Zielmoleküle quantifizieren:
"Das gelingt, weil der Farbstoff und das gesuchte Molekül nicht direkt binden, sondern jeweils an einen kurzen DNA-Strang gekoppelt sind. Diese Abschnitte sind in ihrem Aufbau komplementär, so dass sie sich wie zwei Hälften eines Reißverschlusses zusammenfügen. Dann verrät der Farbstoff das Zielmolekül und dessen Position. Die Stärke der DNA-Bindung kann eingestellt werden. Ist sie schwach genug, lösen sich die beiden Stränge voneinander, und das Signal erlischt wieder. Nach dem sogenannten Exchange-PAINT-Ansatz können dann in nachfolgenden Schritten weitere Moleküle in der experimentell fixierten Zelle nachgewiesen werden. [...]
Wie die Forscher berichten, kann jetzt dank qPAINT erstmals auch die Anzahl der nachgewiesenen Moleküle präzise bestimmt werden. Dafür wird die Bindungsstärke der DNA-Stränge gezielt justiert. Sie ist so eingestellt, dass sich die Nukleinsäure-Abschnitte nach einer festgelegten Zeitspanne voneinander trennen. Dann sind sie frei für eine weitere Bindung, die den Farbstoff erneut zum Aufleuchten bringt."
Komplette Pressemitteilung hier.
Abb.: Maximilian Strauss / MPI f Biochemie
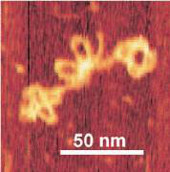 Nano-Bastelei
Nano-Bastelei
Ein Team um Michael Famulok von der Universität Bonn bastelt komplizierte Nano-Strukuren aus DNA:
"DNA ist ein ausgezeichnetes Baumaterial für die Nano-Welt: Sie bildet ein sehr stabiles Grundgerüst und an jeder beliebigen Stelle lässt sich einer der Stränge herausnehmen und als Anknüpfungspunkt für weitere Bauteile verwenden. Auch die Anknüpfung funktioneller Gruppen ist kein Problem. Auf diese Weise lassen sich komplexe Systeme aus DNA-Molekülen konstruieren.
Zur Herstellung ihrer beweglichen Bauteile wählte das Team um Michael Famulok von der Universität Bonn das Bauprinzip der sogenannten Rotaxane. Dabei handelt es sich um eine Molekülklasse, bei denen ein oder mehrere molekulare Ringe auf eine Achse „aufgefädelt“ sind, auf der sie sich frei bewegen können, wobei das Abfädeln durch „Stopper“ verhindert wird. Sind die DNA-Ringe selbst fest an ein Ende der Achse gebunden, so lässt sich der Ring eines Moleküls auf die Achse eines anderen auffädeln und umgekehrt. [..]
Wie werden aber die beiden DNA-Moleküle ineinander gefädelt? Famulok und seine Kollegen greifen hierzu auf das Prinzip der spezifischen Basenpaarung zurück: Sie lassen in der Mitte der Achsen sowie an einer seitlichen Stelle des Rings je eine kleine „Lücke“ aus einzelsträngiger DNA. Deren Sequenzen sind komplementär zueinander. Kommen die einzelsträngigen Bereiche von Ring und Achse miteinander in Kontakt, binden sie aneinander und „kleben“ dabei jeweils Ring und Achse zweier Moleküle wechselseitig aneinander."
Komplette Pressemitteilung hier.
Abb.: (c) Wiley-VCH








 Malen nach Zahlen mit DNA
Malen nach Zahlen mit DNA Nano-Bastelei
Nano-Bastelei