Ganz ehrlich — wir geben uns sehr viel Mühe, die teilweise komplexen wissenschaftlichen Zusammenhänge exakt auszudrücken.
Ein Beispiel: Nahezu alle Printmedien schreiben inzwischen, dass Gene etwas machen. „Gen X macht Krebs“, „Gen Y macht hässlich“, „Gen Z macht lange Finger“, und und und … Wir machen das nicht! Wir achten peinlich genau darauf, dass in unseren Texten Gene NICHTS machen. Denn in der Zelle machen sie schließlich auch nichts, da liegen sie nur mehr oder weniger verpackt herum. Vielmehr macht die Zelle mittels ihrer Enzyme was mit den Genen: Aktiviert und exprimiert sie, legt sie wieder still, vervielfältigt sie, repariert sie, modifiziert sie, ver- und entpackt sie,…
Nein, Gene an sich machen nix. Und selbst wenn es sich mittlerweile auch im Laborsprech etabliert hat, den komplexen Weg vom Gen zum Effekt schnell mal als „Gen X macht Effekt Y“ abzukürzen — in unseren geschriebenen Texten würden wir das als unexakt, ja sogar als schlampig empfinden. Daher versuchen wir, die „machenden Gene“ zu vermeiden — obschon uns sicherlich in manchem Text das eine oder andere doch mal durch die Lappen geht.
x
Wieviele Moleküle, wieviele Verbindungen?
x
Dass uns bisweilen aber noch weitere begriffliche Schlampigkeiten durch die Lappen gehen, darauf haben uns kürzlich zwei Leser aufmerksam gemacht. Und was sie jeweils genau anprangern, ist nicht nur richtig — sondern durchaus auch interessant, da die betreffenden Ungenauigkeiten womöglich auch vielen Leserinnen und Lesern nicht bewusst sind.
Leser Nr. 1 schrieb:
Sehr geehrte Damen und Herren,
in dem Untertitel zum Beitrag von Andrea Pitzschke innerhalb des Specials „Bioaktive Materialien“ ist mir ein Fehler aufgefallen:
Dort ist die Rede von „[…] regelmäßigen Sterilisationsmaßnahmen […]“ zur Prävention von Wundinfektionen. Hier hätte es Desinfektionsmaßnahmen heißen sollen. Eine nachträgliche Sterilisation einer Wunde ist nicht möglich, allenfalls eine Desinfektion, da die für eine Sterilisation erforderlichen physikalischen oder physikalisch-chemischen Verfahren für lebendes eukaryotisches Gewebe (und sicher auch für menschliches Gewebe) nicht verträglich sind.
Zum Trost: Ich bemühe mich schon einige Jahrzehnte Doktoranden und anderen Mitarbeitern die Unterschiede zwischen Desinfektion und Sterilisation beizubringen.
Mit freundlichen Grüßen, …
x
Und Leser Nr. 2 schrieb etwas ausführlicher:
Sehr geehrte Damen und Herren,
als ein Medium für Medizin- und Biowissenschaften, also im naturwissenschaftlichen Bereich angesiedelt, ist das Laborjournal dem exakten Ausdruck wissenschaftlicher Begriffe verpflichtet. Schließlich ist eine saubere Ausdrucksweise notwendig für die eindeutige Kommunikation und das unbeschwerte Verständnis zwischen den Wissenschaftlern, besonders wenn sie aus verschiedenen Bereichen kommen. Auch bemühen wir Hochschullehrer uns sehr, den Studierenden klarzumachen, warum es so wichtig ist, sich klar und eindeutig auszudrücken. Ich glaube, diese Aspekte sind nicht sonderlich kontrovers.
Als Chemiker stört mich deswegen die Nutzung des Begriffs „Molekül“, wo „Verbindung“ (oder „chemische Verbindung“) gemeint ist. Ein Beispiel: Im Laborjournal Nr. 1-2 lese ich […in der Rubrik „Frisch erforscht“; die Red.]: „Trüffelaroma kann nämlich im Labor synthetisiert werden, der typische Geruch beruht im Wesentlichen auf vier bis sechs Molekülen.“ So empfindlich dürfte nicht einmal eine trüffelsuchende Schweineschnauze sein, dass vier bis sechs Moleküle ausreichten!
Ich vermute, dass der Autor vier bis sechs chemische Verbindungen meinte — aber was er schreibt, bedeutet einfach was anderes und ist schlicht falsch. Der Ausdruck oben sagt etwas anderes aus als vom Autor beabsichtigt. Diese schlampige Nutzung der beiden Begriffe, mit „Molekül“ als Synonym für „Verbindung“, sieht man leider zunehmend auch in rein chemischen Zeitschriften. Chemical and Engineering News, immerhin publiziert von der American Chemical Society, ist ein abschreckendes Beispiel dafür.
Man kann nur darüber spekulieren, warum diese Fehlnutzung so um sich greift. Findet ein Laborjargon Eingang in die Literatur? Ist die sprachliche Präzision kein Wert mehr? Sucht der Autor vielleicht nach einem Synonym für Verbindung? (Dann gibt es andere und unkritische Möglichkeiten: Substanz, Spezies, Material, et cetera — je nach Situation.) Wie auch immer — jedenfalls ist es für jeden Autor ratsam, seine eigenen Formulierungen immer wieder kritisch zu betrachten.
Mit freundlichen Grüßen, …
x
Wir können uns bei diesen beiden Lesern nur bedanken. Vielleicht machen uns ihre Zuschriften in puncto exakte Begriffe ja wirklich künftig noch ein kleines bisschen besser.
x


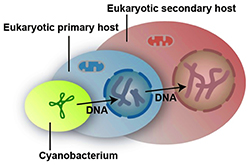 Da wir’s gerade sowieso mit Evolutionsthemen haben: Ein weiteres ist die
Da wir’s gerade sowieso mit Evolutionsthemen haben: Ein weiteres ist die  Schon mal gemerkt, dass Studien mit dem Fazit „höher/weiter/mehr als bisher gedacht“ gerade große Konjunktur haben? Neuestes Beispiel: Gleich zwei frische Paper verkünden, dass nicht nur in Bakterien, sondern auch in frühen Eukaryoten horizontaler Gentransfer — also die Aufnahme von Genen anderer Organismen ins eigene Genom — weitaus häufiger stattfand „als bisher gedacht“.
Schon mal gemerkt, dass Studien mit dem Fazit „höher/weiter/mehr als bisher gedacht“ gerade große Konjunktur haben? Neuestes Beispiel: Gleich zwei frische Paper verkünden, dass nicht nur in Bakterien, sondern auch in frühen Eukaryoten horizontaler Gentransfer — also die Aufnahme von Genen anderer Organismen ins eigene Genom — weitaus häufiger stattfand „als bisher gedacht“. Mitte September. Der Urlaub ist vorbei, der Rest der semesterfreien Zeit mit Tagungen und Kongressen vollgestopft — und wie jedes Jahr um diese Zeit sieht man den ein oder anderen Forscher bereits unruhig werden. Unauffällig stocken sie den Sektvorrat im Kühlraum auf und erkundigen sich beiläufig schon mal nach dem besten Partyservice der Stadt. Man kann ja nie wissen, ob etwa am 8. oder 10. Oktober nicht plötzlich das Telefon klingelt und vööööööllig unerwartet jemand aus Stockholm am anderen Ende ist…
Mitte September. Der Urlaub ist vorbei, der Rest der semesterfreien Zeit mit Tagungen und Kongressen vollgestopft — und wie jedes Jahr um diese Zeit sieht man den ein oder anderen Forscher bereits unruhig werden. Unauffällig stocken sie den Sektvorrat im Kühlraum auf und erkundigen sich beiläufig schon mal nach dem besten Partyservice der Stadt. Man kann ja nie wissen, ob etwa am 8. oder 10. Oktober nicht plötzlich das Telefon klingelt und vööööööllig unerwartet jemand aus Stockholm am anderen Ende ist…