Tipp 84:
Software-Tools für DNA- und Proteinmoleküle
Alignment-Klassiker: BioEdit 7.0
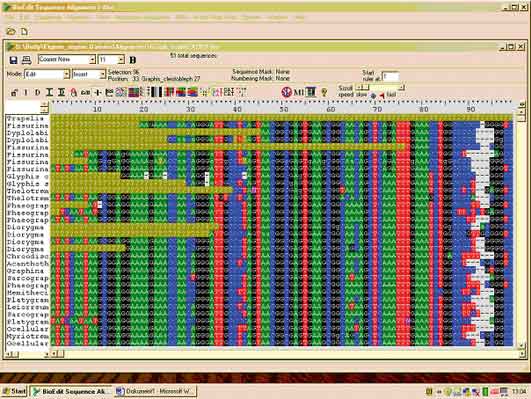
Seit der erstmaligen Veröffentlichung im Jahr 1997 hat Entwickler Tom Hall (Fa. Ibis Therapeutics, Carlsbad, USA) sein Sequenzanalyse-Programm BioEdit stetig verbessert. Die aktuelle Version 7.0.5 dieser Software ist unter Windows 95/98/NT/2K und neuerdings auch unter Windows XP lauffähig; sie beansprucht 12,6 MB Festplattenspeicher.
BioEdit bietet unter anderem eine "tolle Alignmentfunktion" (O-Ton eines computererfahrenen LJ-Lesers) sowie eine Reihe weiterer Werkzeuge, mit denen der Genforscher nach Herzenslust Sequenzen manipulieren und analysieren kann. Links zu externen Analyseprogrammen erleichtern zusätzlich die Arbeit.
Link:
www.mbio.ncsu.edu/BioEdit/bioedit.html
Anmerkung des Webmasters: Anfang September 2005 ist die Downloadsite geschlossen.
Restriktionsschnittstellen online: NEBcutter 2.0
Den NEBcutter müssen Sie nicht installieren, weil er übers Internet läuft. Sie geben einfach Ihre Sequenz in den Editor ein und erhalten nach wenigen Sekunden eine übersichtliche Darstellung aller möglichen Restriktionsschnittstellen sowie – auf Wunsch – der 0-Cutter (also der Enzyme, die nicht schneiden). Schön, dass sich NEB dabei nicht auf die Schnittstellen der eigenen Enzyme beschränkt. Von LJ-Lesern wird der NEBcutter als "super Software" bezeichnet, die zudem das Einbauen stiller Mutationen erleichtere.
Weiterhin bietet der NEBcutter ein nettes Gimmick: Unter "Silent Mutagenesis" kann der Anwender alle Sequenzpositionen finden, an denen mit Hilfe einer stillen Mutation neue Restriktionsschnittstellen ins interessierende Gen eingefügt werden können.
Link:
tools.neb.com/NEBcutter2/index.php
Wer sucht, der findet: Enzyme Finder
Ein weiteres kostenloses Tool von der NEB-Website ist der Enzyme Finder. Der Anwender kann damit online nach Restriktionsenzymen mit ganz bestimmten Eigenschaften suchen (Erkennungssequenz (z.B. GATATC) oder Enzymtyp (z.B. "Thermophile Enzyme")), sowie diejenigen Enzyme finden, die nach dem Verdau den gewünschten Überhang übriglassen (z.B. CCGG am 3´-Ende).
Ferner erlaubt es der "Enzyme Finder", nachzugucken, welche Enzyme unterschiedlichen Namens an den gleichen Erkennungssequenzen schneiden (zum Beispiel ist das bei Eco R V und Eco 32 I der Fall; beide schneiden ohne Überhang ("blunt") an der Sequenz "GATATC").
Link:
www.neb.com/nebecomm/enzymefindersearchbysequence.asp?
Klonieren per Drag & Drop: BioVL Virtual Lab Bench
7,7 MB Platz benötigen Sie auf Ihrer Festplatte, um sich eine Demoversion von BioVL Virtual Lab Bench downloaden zu können. Das Programmpaket der Martinsrieder Software-Firma Biomax erlaubt das Klonieren von DNA-Sequenzen per "Drag and drop" und bietet "Möglichkeiten, die vergleichbare Software-Produkte nicht haben", so ein LJ-Leser.
Einziger Nachteil: Mit der Demoversion können Sie weder speichern noch exportieren und auch nicht drucken. Dafür müssten Sie dann schon in den sauren Apfel beißen und die Vollversion erwerben. Die heißt ebenfalls BioVL und kostet laut Biomax 500 Euro (für akademische Arbeitsgruppen) beziehungsweise 1000 Euro (für industrielle Kunden).
Link:
www.biomax.de/trial/download/dl_biovl.htm
Schnippeln und SNPs finden im Web: WatCut
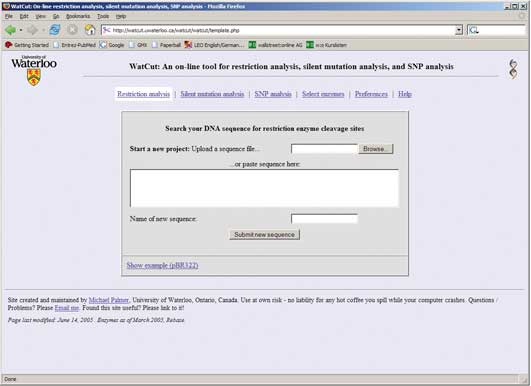
Das Online-Tool WatCut des in Amerika lebenden Lüneburgers Michael Palmer haben wir bereits in Laborjournal 1-2/2005 auf Seite 61 vorgestellt. Palmer, der nach Gießen und Mainz jetzt im kalifornischen Waterloo forscht, hat sein praktisches Programm zwischenzeitlich überarbeitet: Es kann jetzt auch "Amplification-created restriction sites" für die Restriktions-Analyse von Single nucleotide polymorphisms (SNPs) finden.
Link:
watcut.uwaterloo.ca/watcut/watcut/template.php
Rund um den Phosphatrest: PhosphoSite 1.0
Sobald sich das Protein vom Ribosom abgekoppelt hat, geht‘s bei Mehrzellern erst so richtig los: Hurtig wird negativ geladenes PO4 angehängt, bis die Phosphatasen rauchen. Das menschliche Genom enthält über 510 Proteinkinasen und 100 Phosphatasen; zudem werden über 40 % der zellulären Proteine irgendwann in ihrem Lebenszyklus phosphoryliert – oft sogar mehrfach.
Wie aber findet man die für eine Phosphorylierung wahrscheinlichsten Sequenzstellen? Mit Hilfe des kostenlos zu benutzenden Online-Tools PhosphoSite ist das kein Problem: PhosphoSite ist mit einer Proteindatenbank verknüpft und gibt seriöse Hinweise auf in vivo-Phosphorylierungsstellen in Mensch und Maus.
Link:
www.phosphosite.org/Login.jsp
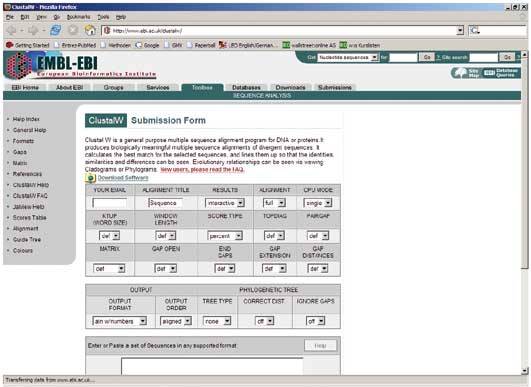
Alignments online überprüfen: Clustal W
Eine Website, auf der sich jeder Sequenzbastler sofort zuhause fühlt, ist die des European Bioinformatics Institute (EBI). Neben einer Unmenge wertvoller Tools und Informationen findet sich dort mit Clustal W auch ein universelles Online-Programm zum Erstellen von Sequenzalignments für DNA und Proteine.
Wer, aus welchem Grund auch immer, keine BioEdit-Version auf seinem Rechner installiert hat (beispielsweise die zahlreichen Apple-User), der kann mit Clustal W ebensogut seine Sequenzen vergleichen und sich anschließend verwandtschaftliche Beziehungen mittels Clado- oder Phylogrammen angucken.
Eine kleine Arbeitsvereinfachung bietet das Programm zudem für Wissenschaftler, die öfters Sequenzen per E-Mail versenden, wie ein Laborjournal-Leser weiß: "Forscher etwa in Indien oder China besitzen oft keine Programme, mit denen westliche Dateiformate geöffnet werden können. Aus ClustalW kann ich in solchen Fällen die entsprechenden Sequenzen einfach per "Paste & Copy" in die Mail kopieren und als "plain text" verschicken. Bei anderen Programmen ist das wesentlich umständlicher."
Link:
www.ebi.ac.uk/clustalw
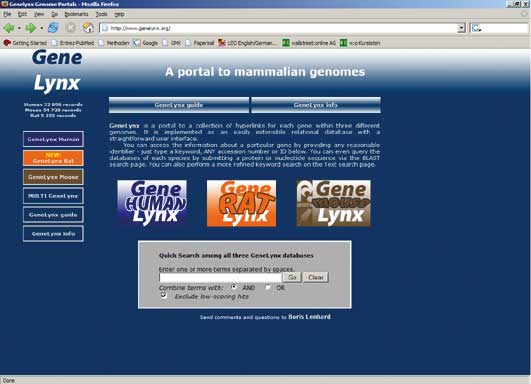
Suchmaschine fürs Gen: Das GeneLynx-Portal
Bestimmt kennen Sie das: Sie suchen eine ganz bestimmte DNA-Sequenz, kennen jedoch nur deren Trivialnamen, etwa "Insulin". Die Suche auf der NCBI-Website (www.ncbi.nlm.nih.gov) liefert unter den Stichwörtern "insulin" und "human" die schlappe Kleinigkeit von 9338 Treffern. Nur Faulpelze weigern sich, die alle nach dem einen, gesuchten Link zu durchforsten. Die anderen hocken sich hin und suchen – und wenn sie nicht verzweifelt sind, suchen sie immer noch.
Was aber machen Faulpelze? Die gehen einfach aufs GeneLynx-Portal klicken alternativ auf "Human", "Rat" oder "Mouse" (in unserem Beispiel: Human) und geben "Insulin" ein. Im Falle von Humaninsulin ist unter den lediglich 54 Treffern dann bereits der 29. der gesuchte. Ein weiterer Klick liefert eine Unmenge von Gendaten und sonstiger Informationen über Humaninsulin, inklusive (unter "RefSeq") die gesuchte Accession-Number für die Genbank. Tja, Faulpelze kommen eben manchmal leichter durchs Leben.
GeneLynx wird vom Ex-Karolinska-Wissenschaftler Boris Lenhard (siehe Foto) betreut, der seit Juli 2005 ans "Center for Computational Science" der Universität von Bergen (Norwegen) gewechselt ist.
Link:
www.genelynx.org
Links bis zum Abwinken: ExPASy
Alle, denen die genannten Links noch nicht reichen, seien auf die ExPASy-Website verwiesen. Dort sind jede Menge Verweise zu weiteren Internet-Ressourcen aufgelistet: Zu Datenbanken (Swiss-Prot, PROSITE, GermOnLine, und und und), zu weit über hundert Tools oder gar umfangreichen Software-Paketen, zu allen möglichen Online-Diensten und vielen weiteren, für Molekularbiologen und Genetiker mehr oder weniger hilfreichen Links. Allein unter der Adresse www.expasy.org/tools sind mehr Einzeltools verzeichnet, als ein Forscher in einem Leben ausprobieren kann. Viel Spaß beim Protein angucken und Genome vergleichen wünscht Winfried Köppelle.
Link:
www.expasy.org
T. Schödl/Fa. Geneart sei für die freundliche Beratung beim Entstehen dieses Artikels herzlich gedankt!
Letzte Änderungen: 07.09.2005