Zellen im Kraftraum
Spezial: Mechanobiologie und Zellphysik
Karin Hollricher
Leben ist Wahrnehmung der Umwelt und Bewegung. Das gilt nicht nur für Organismen, sondern auch für Zellen. Um herauszufinden, wie sie mechanische Kräfte registrieren und darauf reagieren, entwickelten Forscher zuletzt eine Reihe neuer Messmethoden. Drei davon stellen wir vor.
Seit langem weiß man, dass mechanische Kräfte biologische Prozesse beeinflussen. Schon 1817 beschrieb D’Arcy Wentworth Thomson in seinem Buch „On Growth and Form“ die unglaublich vielfältigen Formen der belebten Welt. Der Forscher konnte ohne Kenntnis von DNA und Genen zwar nicht erklären, wie diese Formen entstehen, doch er erkannte, dass sich viele durch simple mathematische Gesetze erklären lassen. Deshalb hält etwa der Neurobiologe Steven Rose von der University of London dieses Buch nach Darwins „Origin of Species“ für das zweitwichtigste Buch der Biologie.
Wachstum und Form lebender Organismen unterliegen indes nicht nur der Gestaltungskraft der Gene, sondern auch derjenigen physikalischer Kräfte. Zellen krümmen, strecken, dehnen und bewegen sich, sie wandern einzeln oder mit ihren Nachbarn – alles, damit sich Organismen entwickeln und leben können. Dabei üben sie Kräfte auf die eigenen Bestandteile im Zellinnern aus, wie auch auf umliegende Zellen – sind selbst aber auch Kräften von außen ausgesetzt.
Obwohl wir es demnach mit einem wichtigen Aspekt der Biologie zu tun haben, steckt die Untersuchung mechanisch-sensorischer Prozesse dennoch in den Kinderschuhen. Vermutlich liegt dies insbesondere daran, dass das Methodenspektrum zur Untersuchung solcher physikalisch-mechanischer Vorgänge ziemlich übersichtlich ist.
Vertraute Nachbarschaft
Doch gerade jetzt gibt es neue Entwicklungen. Einige davon wollen wir im Folgenden vorstellen:
- die Traction Force Microscopy (TFM) in ihrer neuesten Version, der referenzfreien TFM;
- die Elastic Resonator Interference Stress Microscopy (ERISM);
- sowie eine auf Förster-Resonanzenergietransfer (FRET) basierende Messmethode, mit der sich ermitteln lässt, ab welcher Zugkraft sich ein elastisches Biomolekül entfaltet.
Was weiß man heute über mechano-sensorische Prozesse? In aller Kürze und auf das Wesentliche reduziert: Zellen müssen ihre direkte Umgebung wahrnehmen, sich aneinander und auf einer Unterlage festhalten sowie wandern können. Mit sogenannten fokalen Adhäsionspunkten krallen sie sich förmlich in der extrazellulären Matrix (ECM) fest; über Zell-Zell-Kontakte koppeln sie sich an ihren Nachbarn.
Den Kontakt zwischen Außen und Innen vermitteln Adhäsionsproteine, die in der Membran verankert sind. Dazu gehören beispielsweise Integrine, die man auf allen menschlichen Zellen findet (außer Erythrozyten). Sie binden außen an Moleküle in der ECM und innen unter anderem an Paxillin, Vinculin und Talin, die gemeinsam eine Verbindung zu Aktin-Fasern herstellen.
An fokalen Adhäsionspunkten sammeln sich Integrine und konzentrieren damit intrazellulär viele Aktinfasern, die sich ihrerseits über Myosine zu sogenannten Stressfasern miteinander verbinden.
Aufgeben oder feshalten?
Innerhalb dieses Systems versuchen Forscher beispielsweise herauszufinden, mit welcher Kraft sich eine Zelle auf der Unterlage festhält, ob diese Kräfte über die gesamte Auflagefläche gleich groß sind, wie eine Zelle die Steifigkeit ihrer Unterlage detektiert – und wann eine Zelle einfach loslässt und den Kontakt aufgibt. Letzteres ist übrigens eine Kerneigenschaft metastasierender Krebszellen.
Diese Art Geschehen wird mit physikalischen Größen beschrieben. Man misst den Druck, also Kraft pro Fläche (in Pascal), den eine Zelle ausübt, oder stellt diesen als Verformung dar, also als Veränderung der Länge des Untergrunds. Das Verhältnis von Druck zu Verformung bezeichnet man als Steifigkeit oder Elastizität.
Druck und Verformung wurden in den letzten zwanzig Jahren mit verschiedenen Methoden gemessen – beispielsweise mit Rasterkraftmikroskopie, in optischen Fallen, mit magnetischen Pinzetten, oder auf flachen elastischen Untergründen mit TFM. Diese elastischen Untergründe können glatte Oberflächen haben oder eher wie ein Nagelbrett konstruiert sein. Bei diesem so genannten Pillar Array sind elastische Säulen mit einem Durchmesser von 100 Nanometer eng nebeneinander angeordnet; ihre Spitzen können funktionalisiert, also mit Biomolekülen oder chemischen Liganden bestückt werden. Wenn sich die Zellen auf dem Pillar bewegen, werden die Säulen gebogen. Daraus lässt sich die Kraft berechnen, die sie auf den Untergrund ausüben.
„Wir haben damit viel gearbeitet“, sagt Joachim Spatz von der Universität Heidelberg und zugleich Direktor am Max-Planck-Institut für Intelligente Systeme in Stuttgart. „Pillars werden heute aber kaum noch benutzt, weil die Traction Force Microscopy inzwischen so einfach geworden ist.“
Traction Force Microscopy misst, wie der Name andeutet, die Traktion, also eine Form der Zugkraft. Wobei ein Mikroskop ja nicht messen, sondern nur abbilden kann, was auf dem Objektträger passiert. Für eine klassische TFM wird eine glatte Unterlage mit elastischem Material beschichtet, in das in sehr regelmäßigen Abständen Fluorophore eingebettet werden. Darauf kommen die Zellen. Wenn sich jetzt eine Zelle bewegt, wird sie die Unterlage verformen und dabei die Position der Farbmoleküle verändern. Diesen Vorgang dokumentiert man im Bild. Natürlich braucht es dazu eine Referenz. Dazu wäscht man die Zellen von dem Material wieder ab und fotografiert die Unterlage abermals. Aus der Differenz der Positionen der Farbpünktchen lässt sich dann mit mathematischen Methoden (Fourier Transformation oder Finite Elemente Analyse) errechnen, welche Kraft die Zellen aufbringen mussten, um die Moleküle zu bewegen. 1995 beschrieb ein Team um Ken Jacobson von der University of North Carolina in Chapel Hill erstmals diese Art der TFM (Cell Motil Cytoskeleton 31: 225-40).
Die Messgenauigkeit von TFM steht und fällt mit der Fähigkeit, die Farbpünktchen wirklich exakt zu positionieren. Und das war dann auch lange das Problem. Eine weitere Fehlerquelle entsteht beim Abwaschen der Zellen – abgesehen davon, dass man mit ihnen dann auch keine weiteren Analysen mehr machen kann. „Wir haben daher eine Methode entwickelt, bei der wir erstens die Farbpünktchen sehr exakt drucken können und zweitens die Zellen nicht mehr entfernen müssen“, erklärt Tobias Lendenmann, Doktorand in der Arbeitsgruppe von Aldo Ferrari an der ETH Zürich – und einer von drei Erstautoren der Publikation, in der sie die Methode vorstellten (Nature Comm 7: 12814). Der Clou liegt darin, eine Silikonmatrix extrem exakt mit Quantenpunkten zu bedrucken. Das macht man natürlich nicht mit einem Laserdrucker; vielmehr werden die Punkte unter dem Mikroskop aus einer mit Gold beschichteten Glaskapillare unter der exakten Führung eines Piezo-gesteuerten Probentisches gedruckt.
Lendenmann: „Wir platzieren die 200 bis 300 Nanometer großen Dots mit exakt einem Mikrometer Abstand voneinander. Die Abweichungen liegen im einstelligen Nanometer-Bereich.“ So entsteht ein Muster, bei dem jeder Quantenpunkt im Zentrum eines Sechsecks liegt, an dessen Ecken jeweils ein weiterer Quantenpunkt sitzt. Diese Matrix wird mit Fibronektin beschichtet und dann kommen die Zellen darauf.
„Die Zellen strecken sich erst einmal und ziehen sich anschließend wieder zusammen“, beschreibt Lendenmann. „Dabei verschieben sie die Quantendots, was wir mikroskopisch detektieren. Anschließend berechnen wir die Nachbarschaften der Quantendots, das nennen wir Meshing. Aus dem Verhältnis von ursprünglicher und aktueller Position der Quantendots können wir dann die Kräfte berechnen, welche die Zellen auf die Matrix ausgeübt haben.“
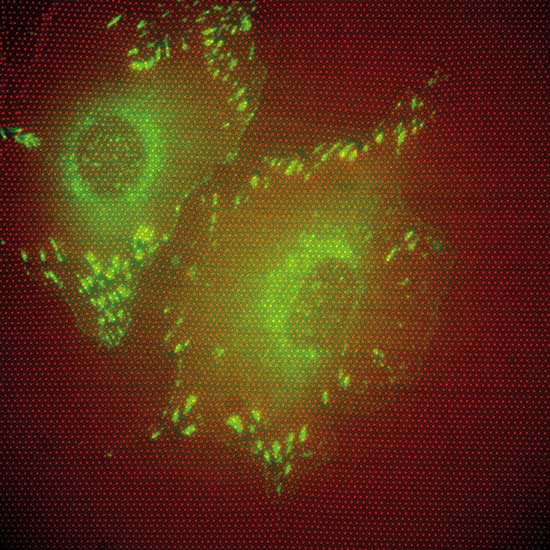
Die Zürcher stellten dabei unter anderem fest, dass die Positionen, an denen ihre Zellen am heftigsten auf die Unterlage gedrückt hatten, perfekt mit ihren fokalen Adhäsionspunkten korrelierten. Diese wiederum identifizierten sie mit Hilfe von markierten Paxillin-Molekülen, die sich in den Adhäsionspunkten konzentrieren. Die berechneten Traktionskräfte lagen schließlich zwischen einem und dreißig Piconewton – und damit im Bereich der Werte, die auch mit anderen Methoden bereits gemessen wurden.
Da Lendenmann et al. überdies konfokale Mikroskope verwendeten, konnten sie neben der lateralen auch die vertikale Verschiebung (in der z-Achse) der Quantendots beobachten. Sie fanden heraus, dass die Zellen lateral, also in der Ebene, bis zu zehnmal stärkere Traktionskräfte ausübten als in vertikaler Richtung.
Kräftemessen
Da man die Zellen – wie gesagt – nach den Messungen nicht abwaschen musste, konnten die Forscher daran weitere Untersuchungen vornehmen. Als Proof of Principle fixierten sie Epithelzellen direkt nach der Kraftmessung und markierten deren YAP-Moleküle mit Antikörpern. YAP ist ein Transkriptionsfaktor, der gemeinsam mit einem weiteren namens TAZ nach stärkeren mechanischen Reizen vom Zytosol in den Kern gelangt, wo sie die Genexpression und damit schließlich auch das Verhalten der Zelle verändern. In den Experimenten der Zürcher drückten die Epithelzellen am Rand einer dichten Zellpopulation am kräftigsten auf den Untergrund, was direkt mit der höchsten Konzentration von YAP im Zellkern korrelierte.
Eine solch koordinierte, ungleiche Verteilung der Kräfte entlang einer einzelnen Zelle beobachteten ebenfalls Forscher an der Universität von St. Andrews in Schottland – allerdings mit einer anderen Methode, der Elastic Resonator Interference Stress Microscopy (ERISM).
Mit Interferenz-Mikroskopie kann man generell die Dicke eines transparenten, durchstrahlten Materials messen. Beleuchtet man ein transparentes Objekt, geht nicht alles Licht hindurch, sondern ein kleiner Teil wird an der unteren, ein weiterer an der oberen Grenzschicht zwischen Umgebung und Material reflektiert. Da die reflektierten Lichtstrahlen unterschiedliche Weglängen bis zu den jeweiligen Grenzflächen zurückgelegt haben, sind ihre Phasen und Amplituden nicht mehr identisch. Wenn sie sich dann treffen, entsteht die Interferenz. Aus dem Interferenzmuster lässt sich schließlich der Wegunterschied und damit die Dicke des Präparats berechnen.
Schnell, aber schonend
Die Arbeitsgruppe von Malte Gather hat auf der Basis dieser schon älteren Technik in St. Andrews eine Methode entwickelt, um die Bewegung von Zellen zu beobachten und die Kräfte zu bestimmen, die sie dabei auf eine elastische Matrix ausüben. Sie vermessen dabei die Verformung der Matrix über die Durchlässigkeit, oder Transmission – was besonders sensitiv entlang der z-Achse möglich ist.
„Damit können wir vertikale Kräfte auf weniger als ein Piconewton exakt berechnen und haben eine Sensitivität von unter 300 Piconewton in der lateralen Fläche“, erklärt Gather. „Dabei sind wir sehr schnell, denn wir rastern die Bilder nicht, sondern nehmen wie bei konventioneller Lichtmikroskopie mit jedem Bild die gesamte Probenfläche auf.“ Allerdings müssen die Forscher viele Bilder entlang des Wellenlängen-Spektrums von 550 bis 750 Nanometern machen. Bei einer Aufnahme pro Wellenlänge sind das am Ende 201 Bilder, aus denen sie dann das Interferenzbild erhalten. Auf diese Weise benötigten sie vor einigen Monaten doch fünf Sekunden für ein vollständiges Interferenzbild, aus dem dann die Verformung berechenbar ist (Nature Cell Biol 19: 864-72). Mittlerweile, sagt er, sind sie schneller.
Um die Theorie in der Praxis zu beweisen, legten die Schotten für ihre Pionierstudie Makrophagen unter ihr ERISM. Aus den Interferenzbildern konnten sie dann schließen, dass die Ausläufer der Zellen, die für die Wanderung wichtigen Podosomen, mit etwa zehn Pascal auf die Matrix drückten, die Zellkörper aber deutlich weniger Druck ausübten.
Auch für ERISM ist kein Referenzbild nötig, sodass man an die Kraftmessung weitere Untersuchungen wie etwa Immunfluoreszenz-Mikroskopie anschließen kann. Außerdem ist die Methode für die Zellen sehr schonend. Die Lichtintensität liegt bei hundert Mikrowatt pro Quadratzentimeter und ist damit niedriger als bei TFM. Daher kann man die Zellen lange beobachten. Gather: „Wir haben mal eine ganze Woche lang Fibroblasten messen können – und dabei Mitosen und auch Differenzierungsprozesse beobachtet.“
Federleicht
Eine neue Methode, die Kräfte zwischen Molekülen zu bestimmen, hat die Gruppe von Carsten Grashoff am MPI für Biochemie in Martinsried entwickelt. Wobei sie nicht wirklich neu ist, sondern letztlich eine Verbesserung der Biosensoren darstellt, mit denen die Martinsrieder schon länger arbeiten. Das Prinzip basiert auf dem Förster-Resonanzenergietransfer (FRET). FRET benutzt man für gewöhnlich, um zu testen, ob zwei Moleküle miteinander interagieren. Das Prinzip ist nicht kompliziert: Man markiert die beiden Moleküle, die man intimer Kontakte verdächtigt, mit zwei unterschiedlichen Fluorophoren. Diese wählt man so, dass bei spezifischer Anregung das eine seine Energie auf das benachbarte zweite Fluorophor überträgt – woraufhin dann dieses fluoresziert.
Die Mitarbeiter der Grashoff-Gruppe benutzen FRET allerdings als Kraftsensor. Wie das geht, erklärt Pia Ringer, Doktorandin und Erstautorin der betreffenden Publikation (Nature Methods, doi: 10.1038/nmeth.4431): „Das Prinzip ähnelt dem einer Federwaage“, sagt sie. „Der Sensor besteht aus einer Feder, an die außen zwei Fluorophore gekoppelt sind. Diese Feder, ein artifizielles Molekül, entspannt sich schlagartig, wenn man mit einer Kraft von fünf Piconewton daran zieht. Diese Entspannung sieht man als Verlust des FRET-Signals, weil die Fluorophore sich dann zu weit auseinander befinden. Die Methode ist sehr sensitiv, wir können damit im einstelligen Piconewton-Bereich messen. Da wir zwei verschiedene Paare von Fluorophoren einsetzen, die sich beide mit 400 Nanometer anregen lassen, aber unterschiedliche FRET-Signale machen, konnten wir Multiplex-FRET machen, also zwei verschiedene Moleküle gleichzeitig testen.“
Konkret untersuchten die Martinsrieder in situ die Moleküle Talin und Vinculin, die in den fokalen Adhäsionspunkten lokalisiert sind. Talin-Moleküle sind die Verbindungselemente zwischen den Integrinen, die die physikalische Beschaffenheit der Umgebung wahrnehmen, und den Aktinfasern. Talin-negative Fibroblasten können keine Adhäsionspunkte bilden. Die Talin-Sensoren konnten diesen Mangel kompensieren. Nach der Adhäsion bildete sich ein Kräftegradient entlang des stäbchenartigen, an die Aktinfasern bindenden C-Terminus, der von drei bis über sieben Piconewton reichte. Dieser Kräftegradient war abhängig von der Anwesenheit von Vinculin. „Das ist sehr interessant“, findet Ringer, „denn nun können wir die auf Talin wirkenden Kräfte einstellen, indem wir weitere Vinculin- und Aktin-Bindestellen einfügen – oder die vorhandenen ändern und prüfen, was dann in der Zelle passiert“.
Mit der Methode können die Max-Planck-Forscher nun weitere Beiträge zum Verständnis mechanischer Zellkräfte im physiologischen Kontext leisten. Bisher war man mit solchen Experimenten noch nicht sehr weit gekommen. Jetzt aber scheint die Zeit langsam reif, dass sich die Mechanobiologen tatsächlich den konkreten Messungen an Zellverbänden und Geweben zuwenden können – statt sich vorwiegend der Entwicklung neuer Methoden zu widmen.
Letzte Änderungen: 03.11.2017