Partnersuche in Bakterien
(30.1.17) Heute in unserer Reihe über Teilnehmer am letzten international Genetically Engineered Machine (iGEM)-Wettbewerb: Das CeBiTec-Team der Uni Bielefeld und ihr Projekt „Evobodies“.
Bereits zum siebten Mal ging die Uni Bielfeld 2016 mit einem iGEM-Team ins Rennen, berichtet uns Marius Schöller. „Und bis jetzt haben wir jedes Jahr eine Goldmedaille gewinnen können“, freut sich der Student der Molekularen Biotechnologie, der kurz vor seiner Masterarbeit steht. Entsprechend standen dem 2016er-Team auch „alte Hasen“ vergangener Jahrgänge zur Seite. „Wir wurden von zwei Doktoranden betreut, die früher selbst Mitglied eines Bielefelder iGEM-Teams waren“, erklärt Schöller und meint damit Julian Droste (AG Microbial Genomics and Biotechnology) und Boas Pucker (Lehrstuhl für Genomforschung).
Angesiedelt sind die Studenten und Betreuer am Center for Biotechnology, weshalb die offizielle iGEM-Teambezeichnung „Bielefeld-CeBiTec“ lautet. „Bielefeld unterstützt iGEM auch mit Wertungsrichtern und einem Mitglied des Organisationskomitees“, ergänzt Schöller – und versichert, dass die Juroren natürlich keine Teams bewerten, die von ihrer jeweils eigenen Institution kommen.
Mit ihrem diesjährigen Team wollten die Westfalen einen Weg finden, wie man einem gegebenen Protein einen passenden Bindungspartner verschaffen könnte. Mögliche Anwendungen gibt es viele, weiß Schöller. „Zum Beispiel für die Diagnostik oder um eine Targeted Drug zu entwickeln. “ Aber auch die Grundlagenforschung nennt er – etwa die Lokalisation von Proteinen in der Zelle, die man über einen Bindepartner sichtbar machen könnte.
Ein anderes denkbares Beispiel: Es taucht eine neuartige Virusvariante auf, die der Forscher mit einem Schnelltest ohne langwieriges Sequenzieren nachweisen will. Falls er bereits ein charakteristisches Oberflächenprotein des Virus’ identifiziert hat, könnte der Test nun so funktionieren, dass ein spezifischer Indikator an dieses Virusprotein bindet, der sich dann über eine Färbereaktion oder Fluoreszenz verraten könnte. Dafür bräuchte man natürlich auf die Schnelle einen passenden Bindepartner für das nachzuweisende Protein.
Handliche Antikörper
Die Natur findet solche Bindepartner via Trial and Error über Mutation und Selektion. Dieses Prinzip der Evolution haben die Bielefelder abgekupfert und labortauglich weiterentwickelt. Denn wer hat schon Zeit, ein paar zehntausend Jahre zu warten? Entsprechend spricht man von „Gerichteter Evolution“, wenn man im Labor zufällige Änderungen im Genpool induziert und anschließend in eine ganz bestimmte Richtung ausselektiert.
Da die Bielefelder Studenten am Ende ihres Evolutionsprozesses im Labor Antikörper-ähnliche Proteine zu den vorher gewählten Zielproteinen erhielten, tauften sie diese „Evobodies“ – und gaben auch ihrem gesamten iGEM-Projekt diesen Titel. Als Startpunkt legten die Studenten zunächst eine Bibliothek mit Plasmiden an, die für unterschiedliche Nanobodys und Monobodys kodierten.
Nanobodys sind Antikörper, die man in Kameltieren findet. Deren Vorteil: Ein Nanobody lässt sich in einer einzigen Sequenz kodieren. Folglich müssen sich nicht erst verschiedene Untereinheiten zusammenlagern, bis ein funktionsfähiges Protein entsteht – wie das bei unseren klobigen Human-Antikörpern der Fall ist. Ähnlich wie die Nanobodys sind auch die sogenannten Monobodys kleiner als herkömmliche Antikörper und bestehen ebenfalls nur aus einer einzigen Aminosäurekette. „Zudem haben die Monobody- und Nanobody-Varianten, die wir benutzen, keine Disulfidbrücken“, fährt Schöller fort. „Folglich war auch deren Faltung in E. coli kein großes Problem.“
Evolution im Schüttelkolben
Am Ende hatten die Studenten auf diese Weise eine Library angelegt, die mehrere hunderttausend Mono- und Nanobodys kodierte. Für jeden einzelnen Nano- oder Monobody gab es dabei ein separates Plasmid. Der jeweilige Mini-Antikörper war zudem an eine Aktivierungsdomäne fusioniert, die dann die Transkription eines Ampicillin-Resistenzgens initiierte. Allerdings brauchte die Aktivierungsdomäne zusätzlich noch eine DNA-Bindedomäne. Die wiederum lag auf einem anderen Plasmid und war an das Zielprotein fusioniert – das „Target“, für das ein Bindepartner gesucht war.
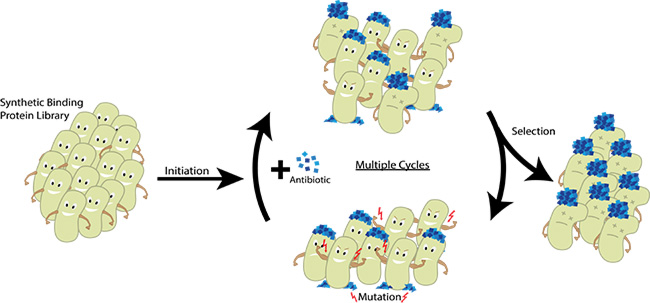
Der Clou dabei war also: Nur wenn Target und Binder eine Affinität zueinander haben, kommen auch Aktivierungs- und Bindedomäne zusammen – und nur dann exprimieren die Coli-Bakterien ihr Resistenzgen und überleben das Antibiotikum im Medium. Ursprünglich kommt dieses System aus der Hefe und nennt sich Yeast Two Hybrid. (Wer sich für die Suche nach Bindungspartnern und Two Hybrid-Systeme interessiert, dem sei auch unser Proteomik-Special vom November 2016 ans Herz gelegt.)
Die Bielefelder brachten also das Plasmid mit der Target-Sequenz zunächst einmal per Zufall mit je einem der Plasmide aus der Antikörper-Library zusammen und klonierten beide Plasmide in E. coli. Natürlich wählten sie nicht „von Hand“ aus der Library, sondern screenten im Hochdurchsatz – und stellten über geeignete Verdünnungen sicher, dass statistisch immer nur ein Antikörper-kodierendes Plasmid pro Bakterium vorhanden war. Auf dem Ampicillin-Medium wuchsen anschließend jedoch nur Bakterien, die bereits Kandidaten exprimierten, die zum Target passten. „Wir sind aber nicht davon ausgegangen, dass in dieser Library schon der perfekte Binder dabei war“, betont Schöller, „sondern setzten zusätzlich auf gerichtete In vivo-Mutagenese!“
Fehlerhaft replizieren
Los ging es nämlich zunächst mit einer geringen Ampicillin-Konzentration, damit auch Bakterienklone überlebten, bei denen Target und Binder noch nicht ideal passten. „Wir haben eine Error Prone Polymerase benutzt, die bei der Replikation immer wieder kleine Fehler einbaut“, erklärt Schöller das Mutationssystem. Die Plasmide mit den Nano- und Monobodysequenzen – also den möglichen Bindern – hatten hierfür einen eigenen Replikationsstartpunkt, der speziell von dieser Polymerase erkannt wurde. Das Plasmid mit der Target-Sequenz und auch die normalen E. coli-Gene blieben von dieser Fehler-Polymerase indes unberührt; sie hatten nämlich einen anderen Replikationsstart und benötigten eine andere DNA-Polymerase.
Durch Erhöhung der Ampicillin-Konzentration nach einigen Generationen steigert man in solch einem System den Selektionsdruck – bis man immer besser passende Binder bekommt. Für ein neues Zielprotein dauert das eine gute Woche, schätzt Schöller.

Konkret ausprobiert hat das Team sein Evobodies-System schließlich mit der Proteindomäne SH2 als Target. „Diese kommt in unterschiedlichen Geweben vor“, so Schöller. Wie bei anderen iGEM-Teams lief jedoch auch den Bielefeldern am Ende die Zeit davon, so dass die Studenten nicht alle Experimente schafften, die sie sich vorgenommen hatten. Möglicherweise werden sie das Projekt jedoch noch weiter verfolgen, deutet Schöller an. „Und vielleicht kommt am Ende ja sogar eine Publikation dabei heraus.“
Ob er auch seine Masterarbeit zu diesem Directed Evolution-System schreiben wird, weiß er allerdings noch nicht. „Die Biotechnologie bietet schließlich noch viele, viele weitere interessante Themen!“
Mario Rembold
Mehr über weitere deutsche Teams des 2016er iGEM-Wettbewerbs:
- Teams Aachen, Freiburg und München in unserer Dezember-Printausgabe (LJ 12/2016: 12-15)
- Team Hamburg im LJ online-Editorial vom 12.12.2016
- Team Düsseldorf im LJ online-Editorial vom 16.01.2016
Letzte Änderungen: 07.02.2017









