„Abfall“ – wie oft haben Forschende Dinge, die sie während ihrer experimentellen Prozeduren vermeintlich nebenbei erhielten, allzu schnell als solchen verworfen. Und lagen damit falsch.
Wie war das etwa mit den kleinen regulativen RNAs? Schon lange dürften die Spezialisten in ihren RNA-Gelen immer wieder „komische Signale“ nahe der Lauffront gesehen haben. Und immer wieder war deren Diagnose: Unspezifische Abbau-Produkte. Experimenteller Abfall also, der durch die Prozedur entsteht und demnach keinerlei biologische Funktion hat. Schließlich ist RNA deutlich instabiler als DNA, und RNAsen lauern auch quasi überall. Und so dachten sie nicht weiter darüber nach – auch wenn „kleine RNAs“ im nächsten und übernächsten Gel wieder vorneweg liefen.
Wer weiß, wie viele von ihnen sich letztlich mit der Hand kräftig vor die Stirn klatschten, als Ende der Neunzigerjahre klar wurde, dass viele Zellen ganz gezielt solche kleinen RNAs produzieren – als Regulatormoleküle, um damit die Expression ihrer Gene feinzusteuern?
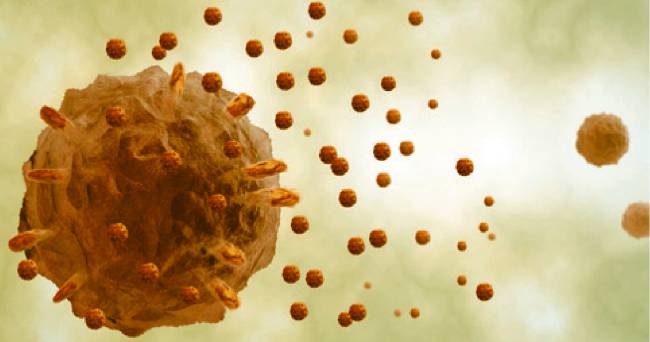
Extrazelluläre Vesikel: Unbedeutender Abfall? – Nicht wirklich!
Ähnlich dürfte es gerade denjenigen Kolleginnen und Kollegen gehen, die lange Zeit extrazelluläre Vesikel als unbedeutenden Zellabfall abqualifiziert haben. Diese kleinen Membrankügelchen werden zwar sicher nicht derart gezielt und systematisch von Zelloberflächen abgeschnürt wie kleine RNAs gebildet werden – aber reiner Abfall ohne jeglichen biologischen Einfluss sind sie keineswegs. Ganz im Gegenteil, stellen sie doch offenbar ein Hauptvehikel für den horizontalen Gentransfer zwischen Meeresorganismen dar, wie ein Team um Susanne Erdmann vom Bremer Max-Planck-Institut für Marine Mikrobiologie in ISME Communications (Vol. 3: 112) beschreibt.
Mittels gereinigter DNA-Datensätze aus Nordsee-Proben fanden Erstautor Dominik Lücking et al., dass ein Großteil der DNA eben nicht „klassisch“ nach Infektion über Viren oder Virus-ähnliche Partikel von einem Organismus in andere transferiert wurde, sondern vielmehr über ins Meerwasser abgeschnürte extrazelluläre Vesikel (EV). Ihr generelles Fazit daher:
Wir haben neuartige und weit verbreitete EV-Produzenten identifiziert sowie quantitative Belege dafür gefunden, dass der EV-vermittelte Gentransfer eine bedeutende Rolle beim horizontalen Gentransfer (HGT) in den Weltmeeren spielt.
Womit erneut ein Beispiel dafür geliefert wäre, dass der Begriff „Abfall“ in der experimentellen Bioforschung nur mit großer Vorsicht verwendet werden sollte. Und dass es sich durchaus lohnen kann, bereits deklarierte Abfälle nochmals zu durchwühlen.
Ralf Neumann
(Illustr.: Thermo Fisher)



 Weil’s so schön war
Weil’s so schön war
 Schon mal gemerkt, dass Studien mit dem Fazit „höher/weiter/mehr als bisher gedacht“ gerade große Konjunktur haben? Neuestes Beispiel: Gleich zwei frische Paper verkünden, dass nicht nur in Bakterien, sondern auch in frühen Eukaryoten horizontaler Gentransfer — also die Aufnahme von Genen anderer Organismen ins eigene Genom — weitaus häufiger stattfand „als bisher gedacht“.
Schon mal gemerkt, dass Studien mit dem Fazit „höher/weiter/mehr als bisher gedacht“ gerade große Konjunktur haben? Neuestes Beispiel: Gleich zwei frische Paper verkünden, dass nicht nur in Bakterien, sondern auch in frühen Eukaryoten horizontaler Gentransfer — also die Aufnahme von Genen anderer Organismen ins eigene Genom — weitaus häufiger stattfand „als bisher gedacht“.



